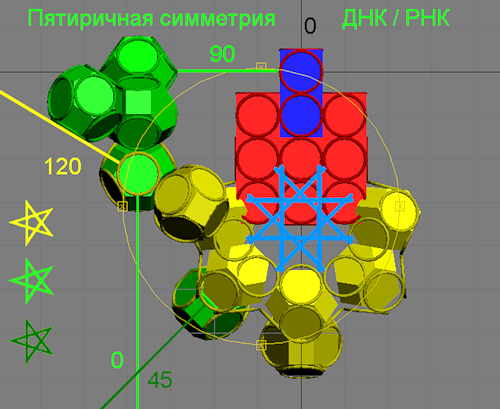
Пикотехнология белков
Начало работы. Пикотехнологические модели двух частей комплекса 1VOL
https://nano-world-articles.nethouse.ru/articles/326755
2016.12.16 16:28:47
Кушелев: В комплексе 1VOL не две одноцепочечные РНК, а двойная спираль из 16 ступеней:
http://www.rcsb.org/pdb/pv/pv.do?pdbid= … ionumber=1

2016.12.16 18:25:26

Так выглядят две субъединицы ДНК из комплекса 1VOL
Можно более или менее красиво изобразить пикотехнологическую модель ДНК изолированными атомами.

Так в программе Hyperchem изображаются атомы азотистого основания гуанина. В принципе сносно.
Осталось добавить координаты атомов рибозы и фосфатной группы. А дальше дублировать нуклеотид, поворачивать и смещать на вектор, который можно взять из скрипта-конструктора ДНК.
2016.12.16 20:43:58
Удалось вписать в PDB файл координаты атомов азотистого основания (гуанина) и рибозы.

Программа Hyperchem показывает атомы углерода светло-голубыми кружочками, атомы азота - синими, а атомы кислорода - красными. Верхний атом кислорода находится ближе к нам, чем плоскость азотистого основания, а два других - дальше. Все остальные атомы лежат в плоскости азотистого основания.
Осталось добавить атом фосфора и три атома кислорода, чтобы завершить модель нуклеотида.
После этого можно будет его дублировать, поворачивать и смещать на вектор, который можно позаимствовать из скрипта "Конструктор ДНК/РНК"
Координаты атомов для файла PDB были взяты по этой разметке:

Оригинал: https://img-fotki.yandex.ru/get/176331/ … 8_orig.jpg
2016.12.16 22:18:14

Координаты для PDB-файла определены.

Модель нуклеотида (гуанинмонофосфата) готова.
Теперь осталось построить 16 звеньев одноцепочечной РНК, дублируя, поворачивая и сдвигая на известный вектор модель одного нуклеотида.
Осталось найти программу, в которой удобно ввести PDB-файл с моделью нуклеотида, скопировать, сдвинуть и повернуть. Кстати, этот вектор нужно умножить на согласующий коэффициент, т.к. модель нуклеотида собрана в другом масштабе...
В скрипте "ДНК-конструктор" радиус кольца-электрона задавался равным 4.8, а в PDB радиус равен 0.7. Соответственно вектор и точка привязки должны быть умножены на коэффициент 0.7/4.8=0,1458333
В программе PyMol вроде можно было задать вращение и перемещение численно. А если не получится, то записать в виде скрипта для 3DS Max, построить модель ДНК и вывести в виде файла координат. После чего добавить координаты в PDB-файл.
Кстати, классная плоская модель нуклеотида получится. Всего 23 вершины тонкой фигуры. Надо посмотреть, как будет выглядеть. Очень компактное графическое представление...
2016.12.17 01:57:


Плоская модель нуклеотида, где вершинами являются выступающие ядра атомов, смотрится очень необычно...
Анимация: https://cloud.mail.ru/public/JXhm/Qz6NrbsRq
Построить на базе этой модели нуклеотида структуру одноцепочечной РНК - не проблема. Завтра мы с этого и начнём...
Плоские модели нуклеотидов могут очень пригодиться для моделирования гигантских РНК типа рибосомальной. Это очень компактное представление, в котором все ядра атомов могут быть установлены идеально.
2016.12.17 13:41:1
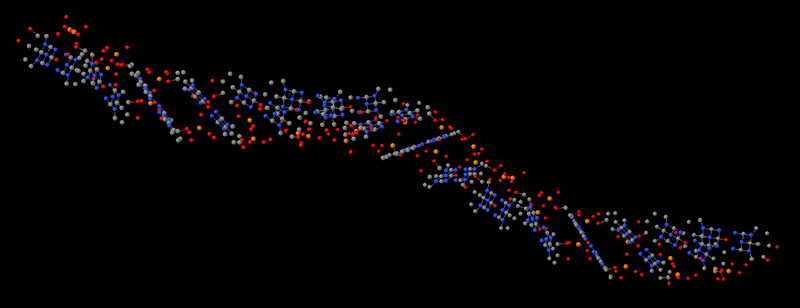
Модель одноцепочечной РНК, состоящей из 16 нуклеотидов: https://cloud.mail.ru/public/69Aq/DxC1qoVho
На этой модели заметно, что на один оборот спирали приходится не 10, а 6 оснований ДНК. При шаге спирали 34 ангстрема на каждую ступень приходится 34/6 =5.67 ангстрема. По общепринятой версии на каждую ступень приходится 34/10=3.4 ангстрема. Это связано с неправильной настройкой угла между нуклеотидами.
Позднее в процессе настройки я получил параметры, которые соответствуют данным РСА, т.е. 10 нуклеотидов на виток ДНК.
Так что углы поворота придётся подправить...
2016.12.17 14:25:55

анимация: https://cloud.mail.ru/public/2dCq/DYkHq7aJt
Теперь оформим эти координаты в виде PDB-файла.
2016.12.17 20:02:53
Но лучше сделать PDB-файл сразу для двухцепочечной РНК. Ведь удалить одну цепочку легко.
Анимация: https://cloud.mail.ru/public/3HLS/tEqiBm5wW



Понятно, что для 16 ступеней двухцепочечной ДНК PDB файл желательно сформировать с помощью скрипта для 3DS Max.
2016.12.17 20:28:56
Skype, 2016-12-17:
[20:19:39] Кушелев Александр Юрьевич: Привет, Валентин!
[20:19:54] Кушелев Александр Юрьевич: Может сохранить из скрипта PDB-файл?
[20:20:26] Кушелев Александр Юрьевич: Есть образец для 1 из 32 кусков, сделанный вручную
[20:22:20] Кушелев Александр Юрьевич: Правда, по образцу можно сделать 16 кусков PDB-файла, а другие 16 кусков на пару атомов меньше, поэтому я сделаю для них второй образец.
[20:25:56] Кушелев Александр Юрьевич: Второй образец отличается тем, что там отсутствует 14-й и 16- атомы из первого образца.
[20:28:25] Кушелев Александр Юрьевич: PDB-файл, который ты выведешь из скрипта, можно послать американскому заказчику, чтобы он вставил модель РНК в белковый комплекс. Если вставится, то это - победа пикотехнологии.
[20:29:16] Кушелев Александр Юрьевич: А если мы сами присобачим модель РНК к моделям белков, то можно будет опубликовать научную статью и подать на нобелевскую
2016.12.18 12:33:20 ПИКОМЕХАНИКА тРНК
2016.12.18 13:04:32 МОДЕЛЬ ФРАГМЕНТА ЛИЗОЦИМА
2016.12.18 13:42:11 МОДЕЛЬ ГИСТОННОГО КОМПЛЕКСА
2016.12.18 14:41:58 КОМПОЗИЦИОННЫЙ КОД
2016.12.18 15:28:05
Skype, 2016-12-18:
[15:26:25] Кушелев Александр Юрьевич: Образец PDB-файла для одной ступени РНК/ДНК
[15:27:46] Кушелев Александр Юрьевич: Нужно по этому шаблону сделать файл для 16 ступеней по этим координатам
[15:28:50] Кушелев Александр Юрьевич: Это можно было бы сделать вручую в программе типа "Волков командер", где можно стоблцами оперировать. Но у меня эта программа не установлена...
[15:42:05] Va12220(valqwa): Саша, привет!
попробую. сегодня до вечера
[15:43:05] Кушелев Александр Юрьевич: Благодарю!
[15:51:14] Кушелев Александр Юрьевич: Интересно сделать вывод файла именно из скрипта, чтобы в будущем выводить такие файлы для РНК/ДНК и комплексов "белок-РНК/ДНК"
2016.12.18 18:56:57
Victoriy
А.Ю., ещё раз сделала модели нуклеотидов и модель двух комплементарных пар А=Т и G=С

Кушелев: Класс! Благодарю!
Что касается комплекса 1VOL, то там двухцепочечная ДНК, а я делаю модели РНК, т.к. из них ДНК получаются исключением OH групп.
2016.12.19 00:32:46
[0:19:33] Va12220(valqwa): все. спать? )


[0:19:57] Кушелев Александр Юрьевич: Класс!
[0:20:29] Va12220(valqwa): долго ли умеючи, ну смотри. делай выводы и поправки. пиши мне их.
[0:20:32] Кушелев Александр Юрьевич: А как ты оцениваешь сложность задачи показать все возможные точки роста молекулы белка?
[0:20:55] Кушелев Александр Юрьевич: На первом шаге 3 точки, на втором 9 и т.д.
[0:21:09] Va12220(valqwa): если ты дашь определение "точек роста молекулы белка" - то как обычную задачу
[0:21:42] Кушелев Александр Юрьевич: Ну помнишь задачу, где треугольник перемещался по трём вариантам?
[0:21:55] Кушелев Александр Юрьевич: Центр треугольника нужно изобразить точкой
[0:22:04] Кушелев Александр Юрьевич: От неё три точки
[0:22:09] Va12220(valqwa): скорее не помню. записи остались. по ним вспомню. когда найду )
[0:22:13] Кушелев Александр Юрьевич: От каждой следующей опять три точки
[0:22:48] Кушелев Александр Юрьевич: Вот алгоритм: .............
[0:23:13] Кушелев Александр Юрьевич: Но здесь по композиционному коду строится конкретная модель белка
[0:23:29] Кушелев Александр Юрьевич: Каждый аминокислотный остаток изображается многогранником
[0:23:39] Кушелев Александр Юрьевич: Подробнее: http://nanoworld88.narod.ru/data/263.htm
[0:23:43] Кушелев Александр Юрьевич: А нам нужно точкой
[0:24:16] Кушелев Александр Юрьевич: И чтобы на каждом шаге было показано не одна точка, а три, т.е. показаны все варианты композиции
[0:24:31] Кушелев Александр Юрьевич: На каждом шаге должно быть "дробление на три"
[0:24:50] Кушелев Александр Юрьевич: Точки должны утраиваться на каждом шаге алгоритма
[0:25:22] Кушелев Александр Юрьевич: Мы должны увидеть все варианты роста белка сразу
[0:25:34] Кушелев Александр Юрьевич: На первом шаге 3 варианта в виде 3 точек
[0:25:45] Va12220(valqwa): да можно все сделать. вот только уже не сейчас. сейчас - спать )
2016.12.19 00:56:40
Татьяна Рясина пишет:
А чем вообще сегодня с учётом современных возможностей можно как следует до атома увидеть молекулы белков? Чтобы таки сопоставить 3D визуализацию по методу Пикотеха и данные экспериментов? Что вообще может прийти на смену РСА?
Кушелев: Кольцевые грани атомов удалось увидеть с помощью АСМ в 2014-ом году: http://nanoworld88.narod.ru/data/493.htm
Однако прощупывать молекулы белков таким способом очень трудоёмко. Я предполагаю, что раньше смогут определить формы некоторых молекул, например, азотистых оснований. Когда увидят, что атомы в азотистых основаниях расположены в шахматном порядке, ситуация снова резко изменится. Ведь во всех базах типа PDB сегодня формы молекул неправильные даже на уровне топологии, не говоря о форме.
После подтверждения формы ступени ДНК с помощью АСМ пикотехнологию будут воспринимать серьёзно.
2016.12.19 02:16:17
[18.12.2016 22:00:47] Andrei: пришел ответ от доктора
[18.12.2016 22:01:31] Andrei: у Соколик лучше результат
[18.12.2016 22:01:37] Andrei: ближе к РСА
[18.12.2016 22:03:33] Кушелев Александр Юрьевич: Она к этому и стремилась.
[18.12.2016 22:04:04] Кушелев Александр Юрьевич: А можно посмотреть на этот ответ целиком?
[18.12.2016 22:04:38] Andrei: I used the models of PDB files to align with the experimental structure in the PDB.
For Kushelev model and 1volA the RMSD was 18 Angstrom (please see figure attached)

For Sokolik TFIIB_204 model and 1volA the RMSD was 16.5 Angstrom
Finally for the alignment of of TFIIB and TFIIB_204 the RMSD was 18 Angstrom.
I used the Pymol software to calculate the alignments.
I wonder whether there is explanation to those results or I miss something.
In addition the sequence of TFIIB 1volA is attached in FASTA file just to make sure the the structural models are based on this specific sequence.
[18.12.2016 22:08:42] Кушелев Александр Юрьевич: Нужна последовательность FASTA и рисунки, вложенные в письмо
[18.12.2016 22:08:42] Andrei: еще есть файлы
[18.12.2016 22:08:49] Кушелев Александр Юрьевич: Давайте их посмотрим
[18.12.2016 22:09:13] Кушелев Александр Юрьевич: 18 анстрем - неплохо для РСА 
[18.12.2016 22:09:56] Andrei: так 2А пишут в базе
[18.12.2016 22:10:07] Andrei: технология улучшилась
[18.12.2016 22:10:14] Кушелев Александр Юрьевич: Там можно и 0.002А написать. Вы тоже поверите? 
[18.12.2016 22:10:45] Andrei: это официальная база, если обманут то это уголовное дело
[18.12.2016 22:10:55] Andrei: никто с этим не будет связываться
[18.12.2016 22:11:06] Andrei: там и репутация автора
[18.12.2016 22:11:31] Andrei: это не совок что кто что хочет то и пишет так как ответственности нет
[18.12.2016 22:11:55] Andrei: послал
[18.12.2016 22:11:59] Кушелев Александр Юрьевич: Вы же видели, что в 2012-ом году "обман" уменьшился на 3% по сравнению с 2010-ым годом
[18.12.2016 22:13:11] Кушелев Александр Юрьевич: Давайте лучше файлы посмотрим.
[18.12.2016 22:13:17] Andrei: послал говорю
[18.12.2016 22:13:26] Кушелев Александр Юрьевич: По почте?
[18.12.2016 22:13:30] Andrei: да
>1VOL:A PDBID CHAIN SEQUENCE
AMMNAFKEITTMADRINLPRNKVDRTNNLFRQAYEQKSLKGRANDAIASACLYIACRQEGVPRTFKEI
CAVSRISKKEIGRCFKLILKALETSVDLITTGDFMSRFCSNLCLPKQVQMAATHIARKAVELDLVPGRS
PISVAAAAIYMASQASAEKRTQKEIGDIAGVADVTIRQSYRLIYPRAPDLFPTDFKFDTPVDKLPQL














[18.12.2016 22:14:15] Andrei: забыл тот ответ - так почему днк должно войти в белок?
[18.12.2016 22:20:35] Кушелев Александр Юрьевич: Комплекс 1VOL - это две белковые молекулы, которые читают ДНК, поэтому структура белковых молекул должна быть согласована со структурой ДНК.
[18.12.2016 22:21:08] Кушелев Александр Юрьевич: РСА показывает, что ДНК проходит внутри белковых молекул. Но деталей не показывает.
[18.12.2016 22:21:35] Кушелев Александр Юрьевич: Если мы сделаем правильные пикотехнологические модели белков и ДНК, то увидим, как они сопрягаются на уровне пикометров.
[18.12.2016 22:22:00] Кушелев Александр Юрьевич: А реальная точность моделей, построенных по данным РСА - десятки-сотни ангстрем.
[18.12.2016 22:22:04] Andrei: когда это будет готово?
[18.12.2016 22:22:55] Кушелев Александр Юрьевич: Если сегодня Валентин сможет вывести PDB-файл молекулы ДНК, то сегодня же можно и попробовать совместить модель ДНК с моделями белков
[18.12.2016 22:23:53] Andrei: щас тогда напишу ей интересно ли ей это направление
[18.12.2016 22:24:46] Andrei: или лучше подождать?
[18.12.2016 22:24:53] Andrei: а то вдруг не сойдется
[18.12.2016 22:24:56] Кушелев Александр Юрьевич: Вы можете написать, что нам интересно построить полную модель комплекса, т.е. proteins + DNA
[18.12.2016 22:25:24] Кушелев Александр Юрьевич: Лучше немного подождать. Нужно проанализировать то, что она написала.
[18.12.2016 22:25:28] Andrei: ага
2016.12.19 09:12:30
Skype, 2016-12-19:
[2:47:17] Кушелев Александр Юрьевич: PDB-файл ДНК готов
[8:52:08] Andrei: нужно к нему описание
[8:52:35] Andrei: что сделано и что хотим показать этим
[9:13:14] Кушелев Александр Юрьевич: Нужно сделать модель всего комплекса. Вот тогда будет что показать на Нобелевскую
2016.12.19 12:03:48

Кушелев: Я так понял, что это сопоставление моей модели TFIIB с моделью из PDB

Модель комплекса из PDB
2016.12.19 15:18:09 ПИКОСОФТ СТАТЬИ, ПЕРЕПИСКА С CASP
2016.12.19 15:36:07
[15:39:17] Кушелев Александр Юрьевич: Несколько кадров из анимации комплекса TBP1+DNA (электронный слой):
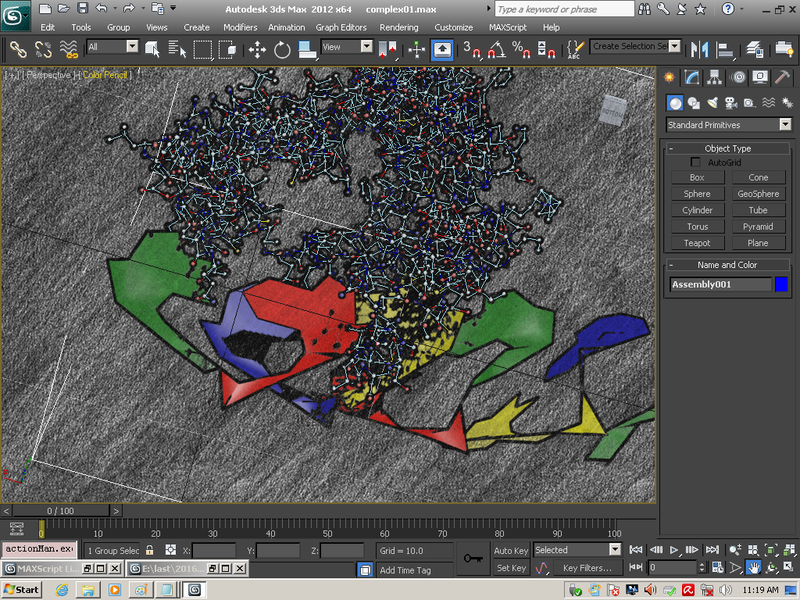





[15:44:34] Кушелев Александр Юрьевич: Тут ещё есть любопытный момент. В банке PDB в комплексе 1VOL показана общепризнанная, но неправильная модель ДНК. Там показаны 16 ступеней ДНК. В пикотехнологической модели комплекса TBP1+DNA (электронный слой) видно, что в белок входит фактически 2 (максимум 3) ступени ДНК. Таким образом можно констатировать факт, что по данным РСА размер ДНК определён с погрешностью типа 400%. Абсолютная ошибка превышает 10 ангстрем в длину участка ДНК.
2016.12.19 17:58:26
Victoriy
Ещё один момент: АЮ не торопитесь с обнародыванием своей модели ДНК, Вы точно отпугнёте заказчицу. Если с белками как говориться бабушка на двое сказала: у каждого своя конформация, которая ещё может и видоизменяться в процессе функционирования. То в структуре ДНК не сомневается ни один биолог и Вам на слово никто не поверит. Кстати с Вашей "новой" моделью ДНК я тоже не могу согласиться.
Не надо загружать заказчика Вашими подробностями и трудностями. Сейчас модели белков, нуклеиновых кислот, лигандов вручную никто не совмещает (это дилетантство), обычно используют метод виртуального молекулярного докинга. (дружеский совет во благо)
2016.12.19 18:11:41
Victoriy
Вопрос к А.Ю.: Ваш файл pdb для нуклеотидной последовательности соответствует общепринятой последовательности атомов в нуклеотиде? Если да, то я буду тоже ориентироваться на данную нумерацию и последовательность атомов, если Вы не сверяли, то это придётся уточнять для однозначного воспроизведения координатных файлов нуклеиновых кислот вьюерами.
2016.12.19 18:14:02
Victoriy пишет:
Вопрос к Андрею: Вы порекомендовали заказчице покрутить мои модели в Молекулярной Динамике? Мне кажется, что после такой процедуры сходство с моделями, построенными по данным РСА, должно стать ещё больше.
Предложение для А.Ю. и Андрея: посмотрите в архиве Наномира мои публикации по пикотехнологии (там и про углы, и про карты Рамачандрана, и что самое главное на языке биологии).
Кушелев: Самое лучшее - это книга "Пикотехнология белков". Андрей хочет прочитать эту книгу.
2016.12.19 18:27:09
Victoriy пишет:
Сейчас модели белков, нуклеиновых кислот, лигандов вручную никто не совмещает (это дилетантство), обычно используют метод виртуального молекулярного докинга. (дружеский совет во благо)
Кушелев: Отлично! Значит достаточно передать заказчику PDB-файлы белков и ДНК, и метод молекулярного докинга сделает своё дело.
Что касается
Victoriy пишет: Вашей "новой" моделью ДНК я тоже не могу согласиться.
то эта модель ДНК была создана в 2011 году. Сегодня я планирую создать окончательный PDB-файл двухцепочечной пикотехнологической модели РНК / ДНК (ядерный слой), который можно будет отослать заказчику для совмещения с моделями белков комплекса 1VOL.
Как видите, в среде 3DS Max модель ДНК идеально совместилась с моделью белка TBP1. Кстати, Вы тоже можете попробовать совместить Ваши модели белков и ДНК. Вдруг они у Вас тоже идеально совместятся

Анимировать: https://cloud.mail.ru/public/Ft3F/o4v8jS5ov
Совмещение пикотехнологической модели ДНК (ядерный слой) с пикотехнологической моделью белка TBP1. Отчётливо видно сопряжение на уровне трёх ступеней ДНК.

Анимировать: https://cloud.mail.ru/public/Ft3F/o4v8jS5ov
Совмещение пикотехнологической модели ДНК (электронный слой) с пикотехнологической моделью белка TBP1
2016.12.19 18:32:09
Victoriy пишет:
Вопрос к А.Ю.: Ваш файл pdb для нуклеотидной последовательности соответствует общепринятой последовательности атомов в нуклеотиде? Если да, то я буду тоже ориентироваться на данную нумерацию и последовательность атомов, если Вы не сверяли, то это придётся уточнять для однозначного воспроизведения координатных файлов нуклеиновых кислот вьюерами.
Кушелев: Я взял из базы PDB-файл с ДНК и просто поменял координаты.
2016.12.19 19:41:41
[16:18:31] Andrei: так подождем до второй модели?
[16:26:39] Кушелев Александр Юрьевич: Вообще-то и первая модель белка - гарантия успеха
[16:27:36] Кушелев Александр Юрьевич: Так что можно посылать 4 ролика анимации, а PDB-модель нужно смасштабировать на коэффициент 0.6 Лучше это сделать самим.
[16:27:53] Кушелев Александр Юрьевич: Жаль, что программист занят. Он бы это сделал за 5 минут
[16:28:05] Andrei: не проблема, подождем
[16:28:57] Кушелев Александр Юрьевич: Через час-два попробую разобраться со вторым белком
[16:29:19] Кушелев Александр Юрьевич: Он не такой очевидный, а в базе данных PDB вообще "полный привет" 
[16:30:04] Кушелев Александр Юрьевич: Там альфа-спирали нарисовали "как бог на душу положит", а модель ДНК с неправильной ориентацией ступеней, поэтому короче в 4 раза
[16:30:21] Andrei: https://cloud.mail.ru/public/5j2J/R81Y4QJjC
[16:30:29] Andrei: это не работает
[16:30:41] Andrei: и вторая тоже
[16:30:48] Кушелев Александр Юрьевич: Я заменил 2 ролика на 4
[16:30:51] Andrei: аа
[16:31:06] Кушелев Александр Юрьевич: Названия поменялись, поэтому я старые два стёр
[16:31:19] Andrei: надо было тут тоже стереть
[16:31:26] Кушелев Александр Юрьевич: ОК
[16:31:33] Кушелев Александр Юрьевич: Исправлюсь в будущем
[16:31:45] Кушелев Александр Юрьевич: Появлюсь через час-два
[16:34:46] Andrei: может там надо подписать в видео что там что?
[17:59:16] Кушелев Александр Юрьевич: Там в названии подписано
[17:59:36] Andrei: ну пусть так
[18:00:05] Кушелев Александр Юрьевич:
[16:28:20] Кушелев Александр Юрьевич: Привет! А ты можешь все координаты в PDB-файле умножить на 0.6 ?
[17:43:34] Va12220(valqwa): МОГУ )
сегодня как доеду и вспомню - то сделаю
[18:00:37] Andrei: отлично
[19:00:33] Кушелев Александр Юрьевич: Комплекс TBP1+DNA идеально вложился в полость белка TFIIB 
Сейчас компьютер делает анимацию.
[19:00:42] Andrei: супер
[19:01:55] Кушелев Александр Юрьевич: Виктория написала на форуме, что против моей модели ДНК. Интересно будет посмотреть, как вставится в комплекс модель ДНК от Виктории. Она пыталась сделать свою модель ДНК максимально приближенной к общепринятой, т.е. из банка PDB
[19:27:43] Кушелев Александр Юрьевич: Это ещё один ролик с TBP1, но без электронного слоя. Так больше видно. https://cloud.mail.ru/public/BR8K/MoUHKhtG5
[19:28:10] Кушелев Александр Юрьевич:
А это ролик всего комплекса 1VOL:
https://cloud.mail.ru/public/5eaU/5T7v7mCwM

[19:29:15] Кушелев Александр Юрьевич: Короче, ДНК вставляется в маленький белок TBP1, а он в свою очередь вставляется в полость более крупного белка TFIIB
[19:31:07] Кушелев Александр Юрьевич: Конечно, нужно ещё применить инструмент "молекулярная динамика", чтобы из жесткой геометрической модели получить гибкую физическую, но это уже профессионалы разберутся
[19:36:57] Кушелев Александр Юрьевич: Кстати, Виктория написала на форуме, что совмещение белков автоматизировано. Так что наш заказчик может самостоятельно совместить PDB-файл модели DNA/RNA с моделью белка TBP1, а потом совместить этот комплекс с белком TFIIB. Технологию молекулярной динамики нужно будет применить сначала после, а потом до совмещения. Если молекулярная динамика на уровне нанотехнологии портит пикотехнологические модели, то применение до совмещения даст хуже результат совмещения.
Кстати, комплекс 1VOL похож на 3 матрёшки. Самая маленькая - ДНК, вторая - белок TBP1, а самая большая - белок TBIIB
[19:37:43] Andrei: так ее модель тоже подошла?
[19:38:59] Кушелев Александр Юрьевич: Виктории? Это мы скоро узнаем
[19:39:03] Andrei: молекулярная динамика это известное что-то всем?
[19:39:10] Кушелев Александр Юрьевич: Да
[19:39:21] Кушелев Александр Юрьевич: Программа PyMol её может делать
[19:39:49] Кушелев Александр Юрьевич: У Виктории и у заказчика она есть, а у меня была, но сейчас нет
[19:40:19] Кушелев Александр Юрьевич: Для неё желательно отдельный компьютер иметь, иначе может всё посыпаться...
2016.12.19 20:15:00
Andrei01 пишет:Victoriy пишет:
Вопрос к Андрею: Вы порекомендовали заказчице покрутить мои модели в Молекулярной Динамике? Мне кажется, что после такой процедуры сходство с моделями, построенными по данным РСА, должно стать ещё больше.
Виктория, а вы можете примеры привести, что сходство увеличится? То есть разница в 18А там уменьшится?
Victoriy
Андрей, я уже Вам писала, что в отличие от Кушелева, моя программа моделирует не конечную конформацию белка, а его СТРУКТУРНЫЙ ШАБЛОН (хотя он оказался ближе к экспериментальным моделям, чем у А.Ю.). Т.е. это структура белка, которая синтезируется на рибосоме БЕЗ влияния микроокружения, но по собственному генкоду (точнее гену). В клетке после того, как структурный шаблон белка синтезирован он подвергается действию всевозможных физических сил (начиная от электростатических и заканчивая Ван-дер-вальсовыми). Этот процесс носит название фолдинг белка. Так вот, процесс фолдинга моего структурного шаблона в конечную конформацию белка можно смоделировать при помощи молекулярной динамики. Мы получим функциональную конформацию белка, которая будет сравнима с моделями, построенными по данным РСА. И разумеется разница в 16,5 Ангстрем должна в разы уменьшиться. А сейчас заказчик сравнивает жесткий скелет с гибким телом и естественно получает такие расхождения. Вот я и предлагаю заказчику самостоятельно превратить скелет в гибкое тело, удивиться и поверить в свой собственный результат, тем более, что нужен он ей, а не мне.
2016.12.19 20:18:40
Victoriy А.Ю., обратите внимание, что Вы сделали модель комплекса с 3-мя нуклеотидами, вместо 16. И если в узле состыковки нуклеотидные хвосты не актуальны, то комплементарные пары нуклеотидов должны быть. Я так думаю:)
2016.12.19 20:21:00
Andrei01
Виктория, хорошо. Напишу им об этом. Поэтому они и слали свои данные, чтобы мы поняли почему такое различие большое в 6 раз.
2016.12.19 20:26:08
Victoriy пишет:
в отличие от Кушелева, моя программа моделирует не конечную конформацию белка, а его СТРУКТУРНЫЙ ШАБЛОН (хотя он оказался ближе к экспериментальным моделям, чем у А.Ю.)
Кушелев
Уважаемая Виктория! А Вы не могли бы осуществить автоматических докинг моделей белков TBP1 и TFIIB ?
Интересно посмотреть, состыкуются ли Ваши модели между собой и мои модели между собой автоматически?
Вручную у меня получилось: https://cloud.mail.ru/public/5eaU/5T7v7mCwM

Теперь интересно проверить автоматизированный докинг. Насколько он работоспособен?
Кстати, непонятно, как Ваша программа строит 310-спирали? По Вашей таблице их в шаблоне быть не должно...
2016.12.19 20:33:02
Victoriy пишет:
А.Ю., обратите внимание, что Вы сделали модель комплекса с 3-мя нуклеотидами, вместо 16. И если в узле состыковки нуклеотидные хвосты не актуальны, то комплементарные пары нуклеотидов должны быть. Я так думаю:)
Кушелев: Я специально убрал половину нуклеотидов, чтобы виднее было. А на модели, где показаны только ядра, ступени целые (пары нуклеотидов). В файле PDB тоже модель двойной ДНК, причём все 16 ступеней.
А заказчик может укорачивать и разбирать модель ДНК из PDB-файла по своему усмотрению.
2016.12.19 20:36:03
Andrei01 пишет:
Виктория, хорошо. Напишу им об этом. Поэтому они и слали свои данные, чтобы мы поняли почему такое различие большое в 6 раз.
Кушелев: Самое интересное - провести автоматический докинг для комплекса 1VOL. Он должен дать разный результат для вариантов моделей Виктории Соколик и Александра Кушелева. Интересно же посмотреть, в каком варианте лучше состыкуются белки между собой и с ДНК
2016.12.19 20:37:25
Victoriy
Увы, у меня пока нет навыков самостоятельного выполнения докинга, тем более 3 высокомолекулярных полимеров. С коллегами из Киева в этом году был сделан докинг куска из 63 а.о. белка предшественника амилоида (AбетаPP), содержащего сам бета-амилоидный пептид, с куркумином. Опубликовано в Advances in Biochemistry. – 2016. – V. 4, Is. 4. – P. 34-46.
Поэтому я и призываю в нашу команду биоинженера/молекулярного биолога с опытом работы в молекулярном моделировании.
2016.12.19 21:01:56
Кушелев: А может быть программы типа PyMol могут автоматически выполнять докинг двух белковых молекул?
2016.12.19 21:31:03
Victoriy
Мы использовали инструмент CCDC GOLD Suite 5.1 для докинга и его же оценочные функции для характеристики устойчивости комплекса. Для меня PyMol не более чем вьюер.
2016.12.19 20:42:1
Татьяна Рясина
На сегодняшний день мы стали проще относиться к идолу РСА в результате продолжительной беседы.
Почему бы не выработать отношение попроще и к идолу ДНК?
Вот допустим ответы по задачке заказчицы готовы, пусть они даже отправлены.
Можно же написать что-то вроде, вдогонку:
Позвольте ознакомить Вас с нашей критикой общепринятой модели ДНК
Мы провели моделирование ДНК методами Пикотехнологии.
Наши поправки по алгоритмам Пикотехнологии выявили следующие различия с общепринятой моделью /....../
Корректность наших поправок подтверждается тем что /...../
В дополнении к только что решённой нами для Вас задаче и при совмещении полученной структуры и нашей модели ДНК мы получим следующее /......./ Таким образом /......./
И ещё вопрос к уважаемым изобретателям Пикотеха:
кто напишет в Германию в лабораторию которая увидела электроны-колечки на АСМ
про то что:
1) мы занимаемся моделированием атомов, молекул и молекулярных структур с помощью модельного представления об электронов как о закольцованных стоячих волнах (ссылки)
2) Ваша работа подтверждает эти модели
3) Мы разработали Пикотех для моделирования белковых структур
3) Проверьте пожалуйста базовые структуры белков экспериментально (список наименований, дизайн эксперимента)
Андрей и Виктория, вы можете сформировать максимально полый список критики, вопросов, опровержений и пр. по модели ДНК А.Ю., чтобы в итоге получить хорошую аргументацию в её пользу...
2016.12.19 21:16:46
Кушелев
Всё это актуально, но сложно на уровне голого энтузиазма. Нужно найти заинтересованных людей, которые продвинут новое научное направление "по полной программе". Ведь пикотехнология белков была создана уже в 1993-ем году, а развивается она без нормального финансирования "черепашьими шагами"...
2016.12.19 21:13:10
Victoriy пишет:
А.Ю., мне интересно: в Вашей модели комплекса последовательность нуклеотидов не имеет значение?
Кушелев: Нет, конечно. Если комплекс занимается транскрипцией ДНК, то он должен одинаково хорошо заниматься транскрипцией любых последовательностей.
Кстати, ключевые аминокислотные остатки как бы прикасаются к центральным зонам соседних ступеней ДНК:
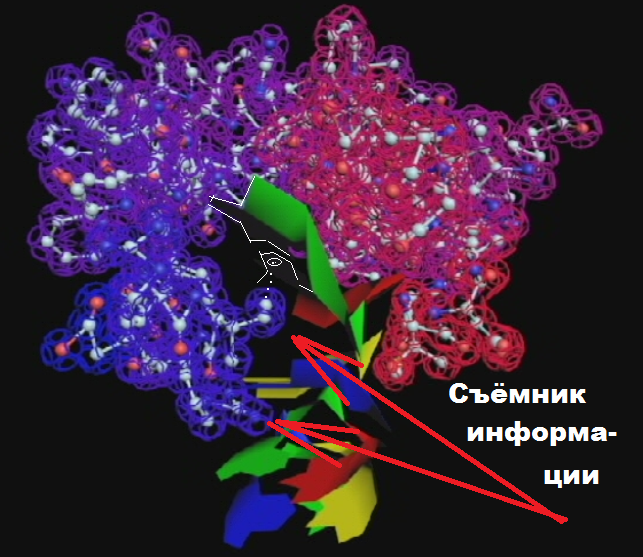
2016.12.20 00:14:09
[19.12.2016 19:44:15] Andrei: Щас попробую все оформить и послать им
[19.12.2016 19:47:32] Кушелев Александр Юрьевич: Скоро приедет домой программист. Тогда будет нормальный PDB-файл DNA / RNA
[19.12.2016 19:56:12] Andrei: а, я думал уже готово
[19.12.2016 19:56:42] Andrei: А это ролик всего комплекса 1VOL:
[19.12.2016 20:00:15] Кушелев Александр Юрьевич: Ролик-то готов, а PDB-файл ДНК/РНК ещё не готов
[19.12.2016 20:01:58] Andrei: Книга вроде 74 евро стоит
[19.12.2016 20:02:20] Кушелев Александр Юрьевич: Да. Электронный вариант 6 USD
[19.12.2016 20:02:31] Andrei: там не было электронного варианта
[19.12.2016 20:02:48] Кушелев Александр Юрьевич: У авторов есть
[19.12.2016 20:04:45] Andrei: как называется ядерный слой на английском?
[19.12.2016 20:05:23] Кушелев Александр Юрьевич: nuclear
[19.12.2016 20:05:56] Andrei: nuclear layer?
[19.12.2016 20:06:15] Кушелев Александр Юрьевич: Да. В файлах подписано nuclear и electronic
[19.12.2016 20:06:34] Andrei: ок
[19.12.2016 20:06:59] Andrei: Файлы лучше загружать на dropbox
[19.12.2016 20:07:36] Andrei: а то сайт на русском, они не поймут
[19.12.2016 20:08:29] Кушелев Александр Юрьевич: Загрузите, если сможете
[19.12.2016 20:08:42] Andrei: загружаю
[19.12.2016 20:08:44] Кушелев Александр Юрьевич: Или в письмо вложите
[19.12.2016 20:09:05] Andrei: в письмо не получится, тот сайт на русском
[19.12.2016 20:09:29] Кушелев Александр Юрьевич: Вы тогда ссылочку на эти файлы в dropbox опубликуйте, чтобы англоязычные читатели форума тоже смогли скачать и изучить.
[19.12.2016 20:09:46] Кушелев Александр Юрьевич: Сами файлы в письмо можно вложить по любому
[19.12.2016 20:10:04] Andrei: нет конечно, почта не пропустит
[19.12.2016 20:10:34] Andrei: я не даю ссылки для всех
[19.12.2016 20:11:13] Кушелев Александр Юрьевич: Только русскоязычным разрешаете файлы скачивать?
[19.12.2016 20:11:33] Andrei: там настройки есть
[19.12.2016 20:11:40] Кушелев Александр Юрьевич: Ушлые китайцы всё-равно с облака мэйл ру скачают
[19.12.2016 20:11:40] Andrei: надо настраивать специально
[19.12.2016 20:45:19] Andrei: так у вас тоже не учитываются силы на белок как у Виктории?
[19.12.2016 20:49:08] Кушелев Александр Юрьевич: Мой алгоритм пока реализован на геометрическом уровне, но генетический код дублирует физико-химические взаимодействия, поэтому подавляющее большинство участков белковых молекул не модифицируются после рибосомальной сборки белка. А те, которые модифицируются, можно смоделировать по дополнительной информации. Бывают и редкие случае, когда "всё сложно"
[19.12.2016 20:50:41] Кушелев Александр Юрьевич: В нашем случае 1VOL похоже, что белки не модифицируются, но в нормальных условиях они могут гнуться, защёлкиваться и т.д. Так что по хорошему учитывать физику и химию нужно.
[19.12.2016 20:50:57] Кушелев Александр Юрьевич: Это уже проблема заказчика.
[19.12.2016 20:51:30] Кушелев Александр Юрьевич: Если нам закажут исследование динамики белка, тогда это будет уже "совсем другая песня" и "совсем другие деньги"
[19.12.2016 20:51:58] Andrei: а почему же тогда оно налезло друг на друга само?
[19.12.2016 20:52:52] Кушелев Александр Юрьевич: Исследователи белков могут выращивать кристаллы в разных условиях, добиваясь фиксации молекул в разных состояниях. В результате они могут получить "фотки" в разных положениях частей молекулы белка.
[19.12.2016 20:53:38] Кушелев Александр Юрьевич: В нашем случае ДНК и белки совместились, т.к. их форма сильно не меняется после сборки рибосомой
[19.12.2016 20:54:09] Кушелев Александр Юрьевич: А бывают белки, которые надкусываются, из кусков собираются крупные агрегаты и т.д.
[19.12.2016 22:50:42] Кушелев Александр Юрьевич: Программист ещё едет домой 
[19.12.2016 22:50:59] Кушелев Александр Юрьевич: А Вы аннотацию к книге "Пикотехнология белков" заказчику посылали?
[19.12.2016 22:51:01] Кушелев Александр Юрьевич:
The greatest enemy of knowledge is not ignorance, it is the illusion
Stephen Hawking
Sokolik VV, Kushelev AY
Geometry live nanoworld. Pikotechnology proteins / VV Sokolik, AY Kushelev. -
Kharkov: Publishing, 2015. – 255 p.
ISBN 978-3-659-92862-8
The monograph is devoted to the 3D-structure of molecules and polymers in
living systems. The analysis of the modern understanding of the fundamental concepts
of the physical volume of the atom, chemical bonding, and the genetic code is
presented. Based on statistical analysis of the experimental data on the protein structure
is justified coding secondary structure and structural polypeptide template of protein
in the genome. Propose additions table of the genetic code of proteins and peptides,
which formed the basis of the geometric algorithm software decoding structural
template protein, are Molecular Constructor and Picotex. The hypothesis of recoding
information to third nucleotide codon in the corresponding peptide bond rotamer directly
3D-structure isoacceptors tRNA is formulated. Mathematical analysis of contingency
angles φ and ψ (Ramachandran map) revealed their frequency changes as
possible to substantiate the mechanism of post-translational protein folding.
The book is intended for professionals involved in research in the field of molecular
biology, bioinformatics, biochemistry and biophysics.
Table: 36. Ill: 117. Refs: 227 titles.
[19.12.2016 22:51:40] Andrei: Ну как доедет, не горит)
[19.12.2016 22:51:52] Andrei: Посылал
[19.12.2016 22:52:12] Кушелев Александр Юрьевич: А рецензии?
[19.12.2016 22:52:26] Кушелев Александр Юрьевич: Это самое научное...
[19.12.2016 22:56:29] Andrei: тоже, это все теория
[19.12.2016 22:56:34] Andrei: нужны примеры
[19.12.2016 22:57:52] Кушелев Александр Юрьевич: Какая же теория, когда речь идёт о модельных экспериментах, подтверждённых в 2015-ом году натурными (АСМ)?
[19.12.2016 22:58:15] Andrei: на практике нет применений
[19.12.2016 22:59:20] Кушелев Александр Юрьевич: Пикотехнология белков - это и есть практическая технология, которая базируется на модельных экспериментах теперь уже подтверждённых прямыми экспериментами (АСМ)
[19.12.2016 23:00:00] Andrei: коммерческой прибыли нет от этого на практике
[19.12.2016 23:00:05] Andrei: надо искать
[19.12.2016 23:01:25] Кушелев Александр Юрьевич: Виктория пишет, что не владеет программой, где делается автоматический докинг. Может быть имеет смысл попросить заказчика применить автоматический докинг для сравнения моделей белков и ДНК от Кушелева и Соколик?
А раз заказчик может сравнивать модель Кушелева с моделью из PDB, значит сможет сравнить и модель Кушелева с моделью Соколик. Интересно, на сколько ангстрем отличаются наши модели?
[19.12.2016 23:01:36] Кушелев Александр Юрьевич: Прибыль будет, не торопитесь.
[19.12.2016 23:01:52] Andrei: ничего просить не можем
[19.12.2016 23:02:22] Кушелев Александр Юрьевич: А предлагать?
[19.12.2016 23:02:31] Andrei: покажем идеи а дальше если стоящая то может быть сможет найти практическое применение
[19.12.2016 23:03:24] Кушелев Александр Юрьевич: Они же сами заинтересованы сделать модель комплекса 1VOL. Если они умеют делать автоматический докинг, то смогут попытаться сделать его в варианте моделей Кушелева и Соколик. В одном из вариантов должно получиться лучше.
[19.12.2016 23:03:38] Кушелев Александр Юрьевич: У меня вручную получилось, а у них автоматически должно получиться.
[19.12.2016 23:04:04] Andrei: зачем им это
[19.12.2016 23:06:16] Кушелев Александр Юрьевич: Их цель, как я понял, выяснить механизм работы комплекса 1VOL
[19.12.2016 23:06:30] Кушелев Александр Юрьевич: Применить для этого автоматический докинг - сам бог велел 
[19.12.2016 23:07:15] Кушелев Александр Юрьевич: А дальше они могут сравнить, какие модели лучше собираются в "матрёшки", от Кушелева или от Виктории. Они же рассчитали 18 и 16.5 ангстрем?
[19.12.2016 23:07:20] Кушелев Александр Юрьевич: Значит нужно.
[19.12.2016 23:07:30] Andrei: что дает докинг?
[19.12.2016 23:08:04] Кушелев Александр Юрьевич: Автоматическую сборку "матрёшки". Я-то делал вручную. Интересно получится та же структура в автоматическом режиме?
[19.12.2016 23:08:36] Andrei: пусть сначала это посмотрят
[19.12.2016 23:08:46] Andrei: посмотрим что скажут
[19.12.2016 23:08:47] Кушелев Александр Юрьевич: Безусловно
2016.12.20 00:50:03
Skype, 2016-12-20:
[0:33:35] Кушелев Александр Юрьевич: Программист добрался до дома. Сказал, что сейчас сделает PDB-файл ДНК
[0:52:15] Кушелев Александр Юрьевич: Файл готов

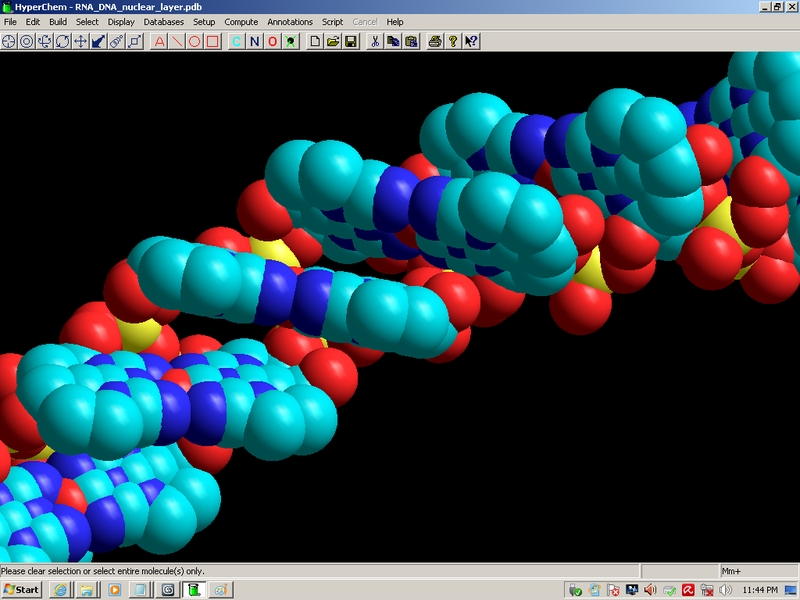
PDB-файл: https://cloud.mail.ru/public/LNUH/7JNWA75Nk
ОБНАРУЖЕНА ОШИБКА В ПРОГРАММЕ, ИСПРАВЛЯЕМ
2016.12.21 18:04:46
aest пишет:
вы пытались продавать наивным биологам ваши модели белков по 10000 уе, заверяя о пикоточности в то время, как у вас в алгоритме ошибка была ? Некрасиво как-то получается.
Кушелев: Конечно некрасиво. Поэтому пытаемся исправить ошибку, но это дело непростое... Углы уже удалось исправить, но в программе Дениса Савина где-то ещё запрятаны смещения. Ищем.















































