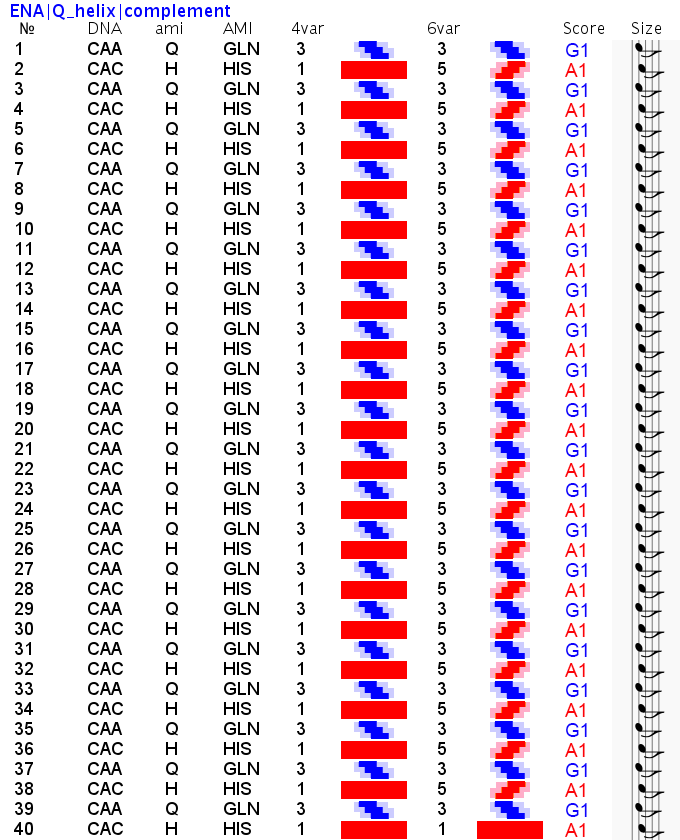
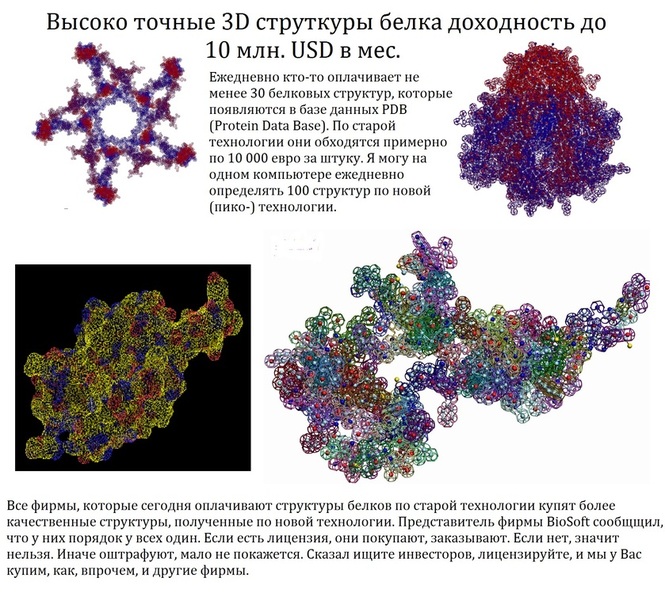
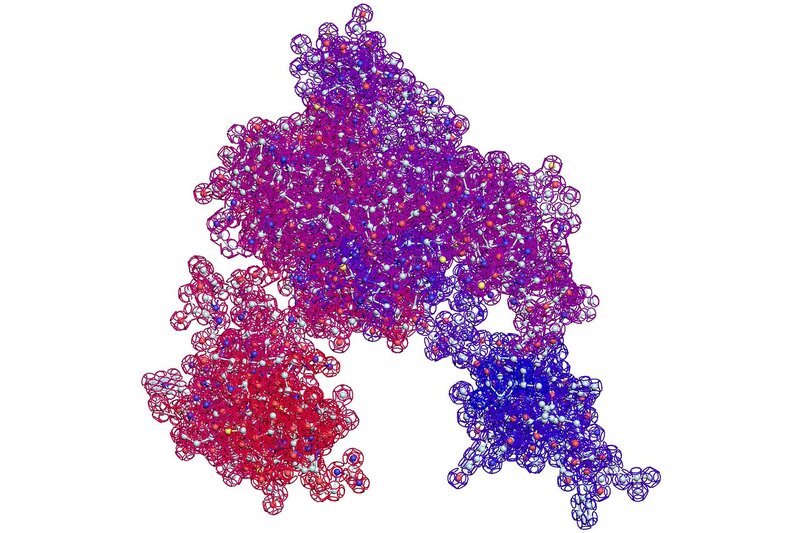
Геометрия живого Наномира
2017.01.17 10:53:57
Victoriy пишет:
могу предлагать продукцию, в которой уверена на 100 %, а это моя программа, а не кушелевская
Кушелев: Я бы не делал акцент на различиях программ, а делал акцент на коллективную работу. Мы же можем давать заказчику варианты, а он сам выберет, какой ему больше понравится. У него будет и Ваш вариант, и мой, и РСА, и ЯМР и т.д. Что ему важнее, тем он и будет пользоваться в первую очередь. Чем шире спектр возможностей, тем привлекательнее сервис. Согласны?
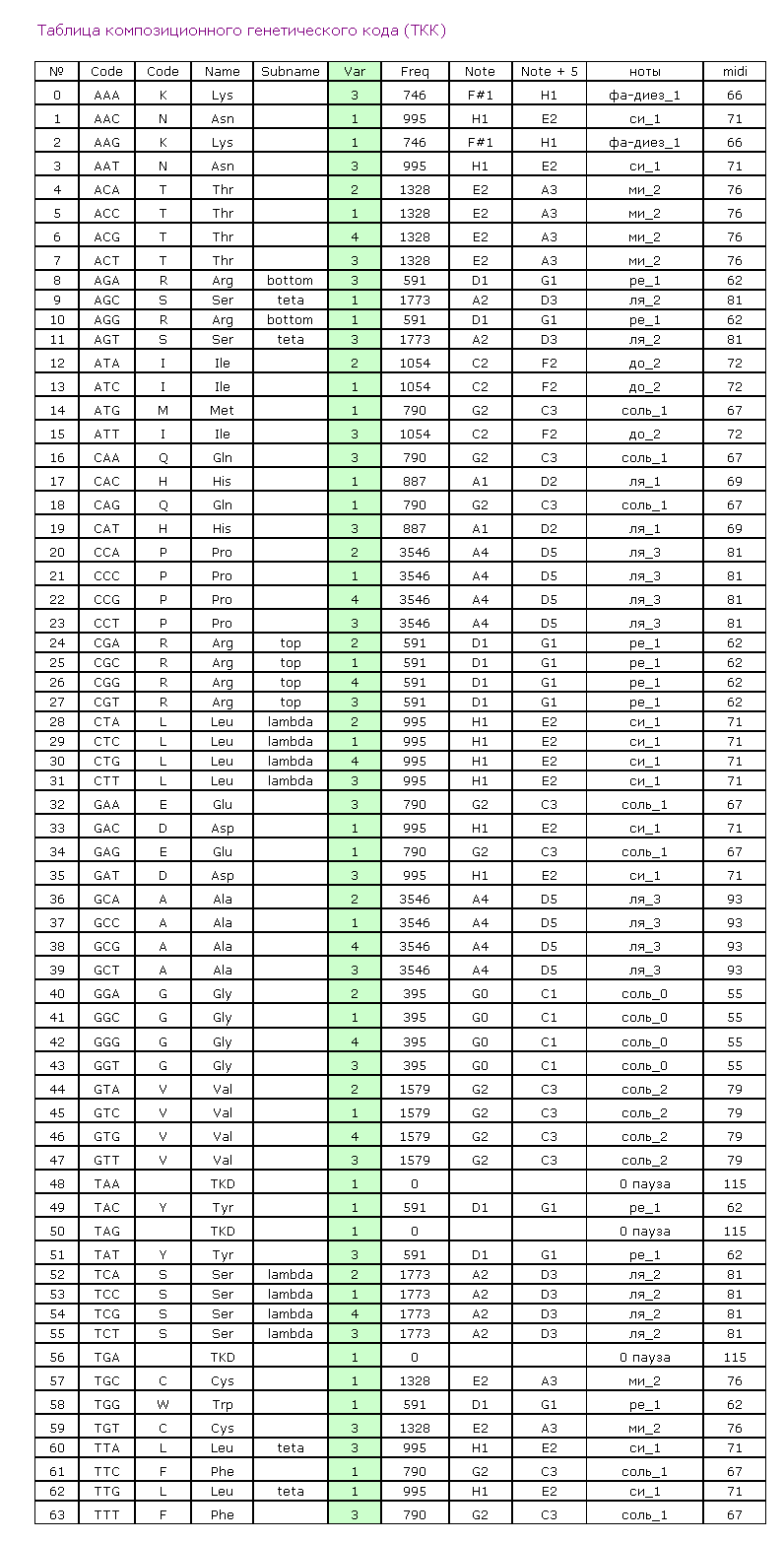
Тем более, что основа наших алгоритмов одна. Композиционный генетический код. А вариаций таблицы и алгоритмов может быть бесчисленное количество.
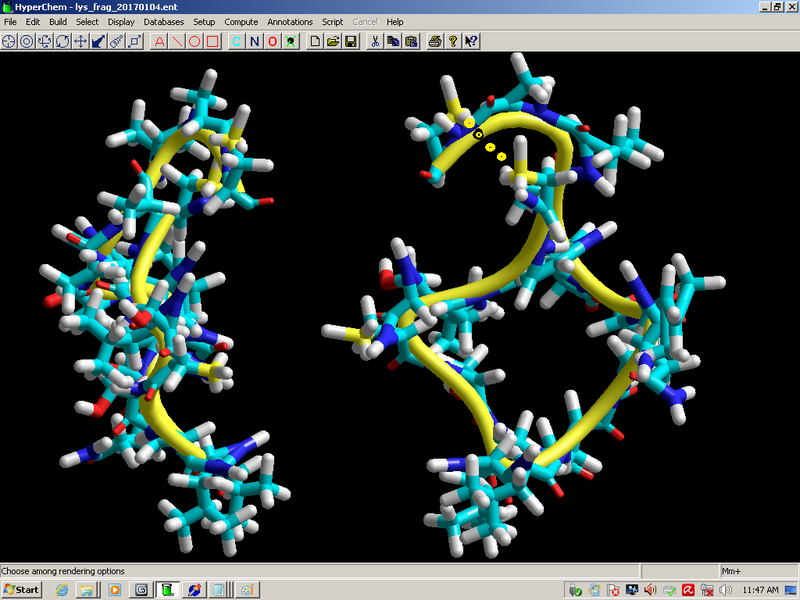
Я, например, для моделирования фрагментов лизоцимов использую сейчас три разных версии алгоритма с разными настройками. Одна версия настроена на вариант "5" с углами альфа-спирали, вторая - на вариант "5" с углами 310-спирали, и третья - на вариант "4" от Савина.
Эти три варианта оказываются эффективными для разных типов последовательностей.
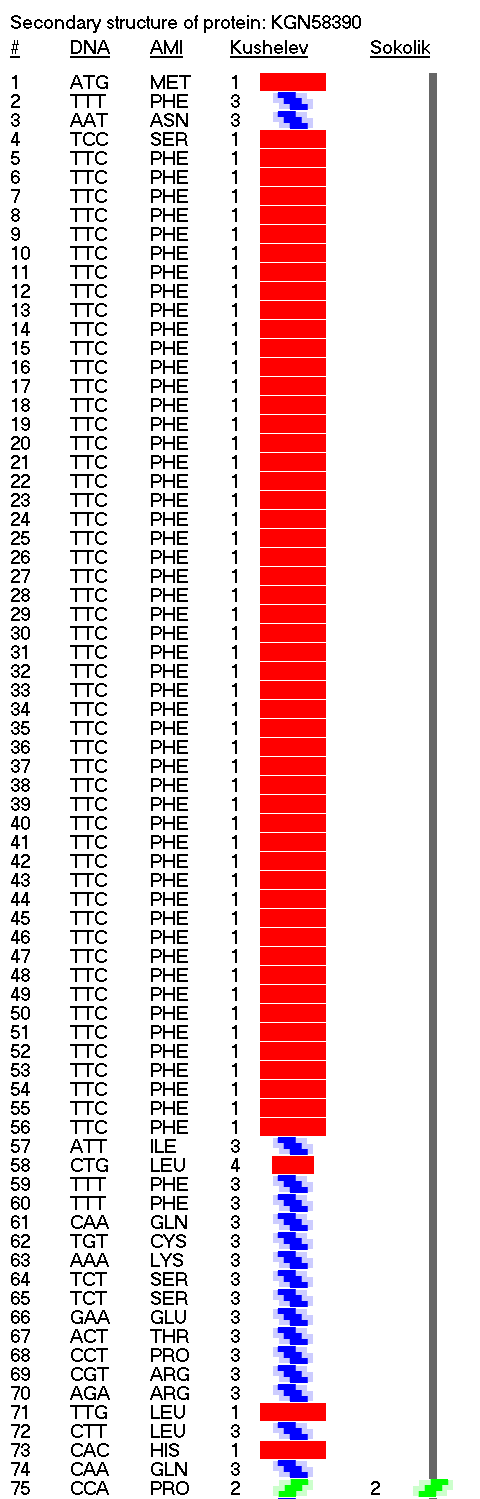
Следующая версия Пикотех объединяет эти варианты, в результате чего композиционный код становится не 1234, а 123456:

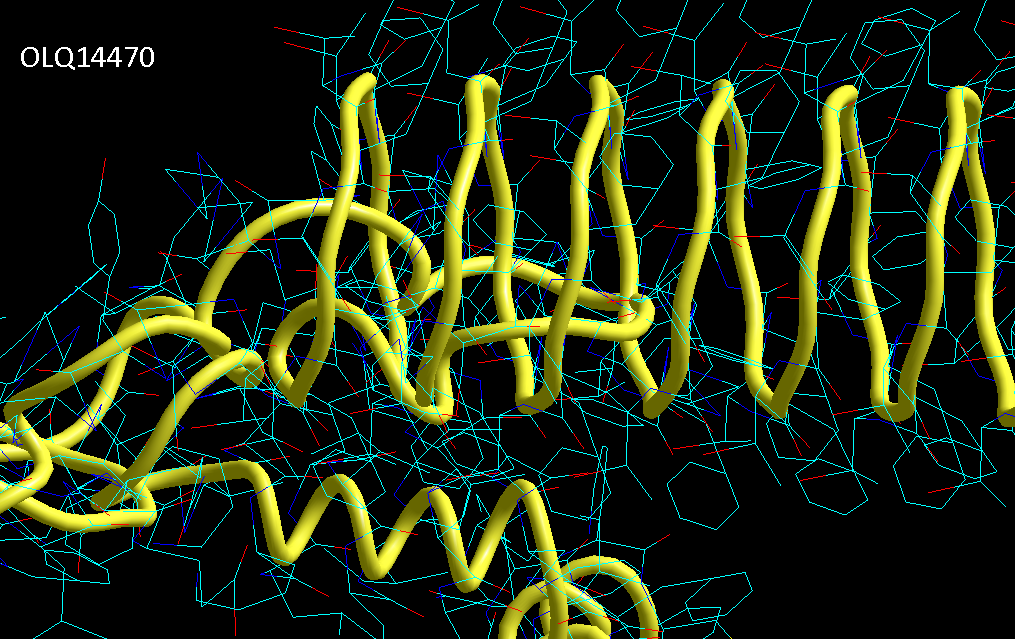
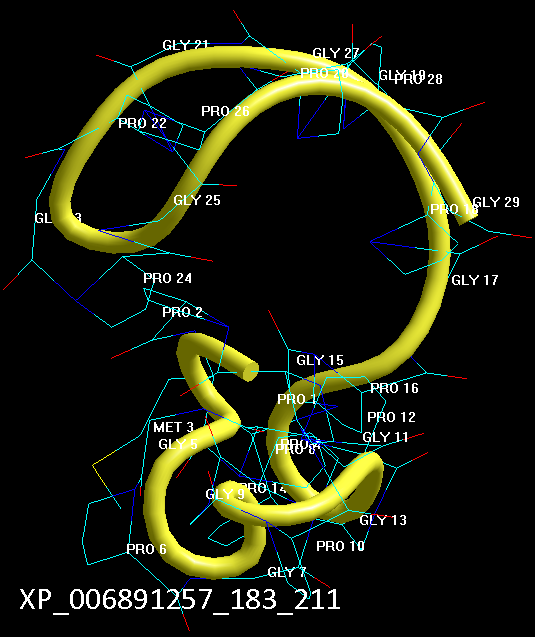
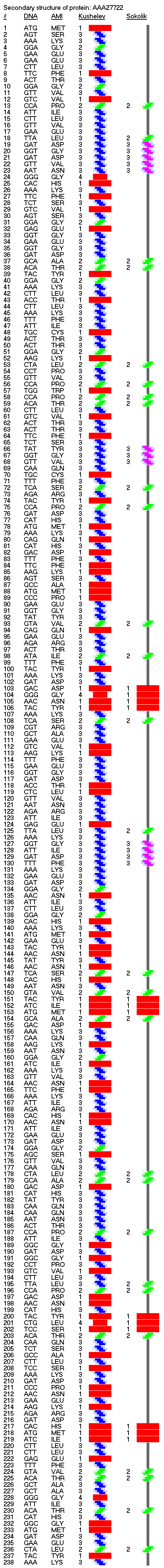
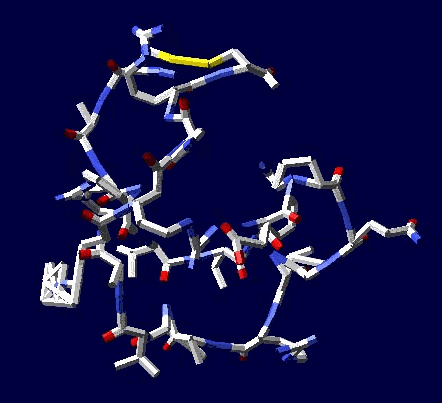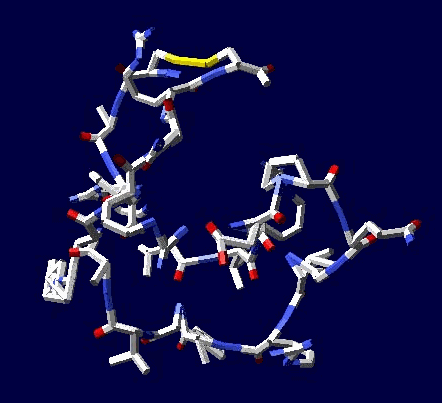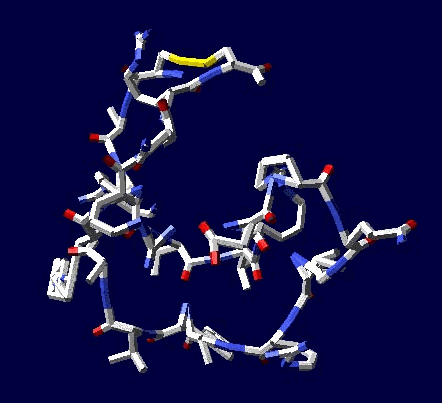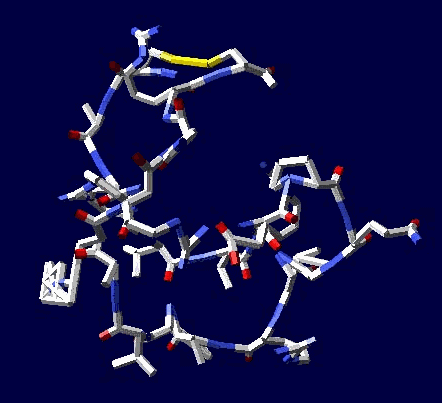
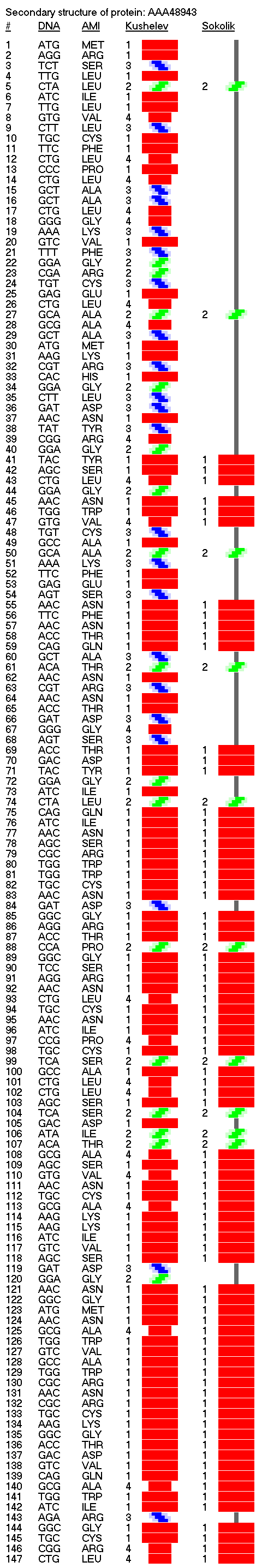
Обратите внимание, что схема вторичной структуры и визуально стала смотреться иначе, т.е. одиночные альфа-310-коды имеют ту же ширину, что и коды бета-пи-. Эта зона вторичной структуры теперь лучше ассоциируется с "неструктурированным" участком между спиралями. Речь идёт о прямых альфа- и 310-спиралях, а так же смешанной альфа-310-спирали, которая и является основным мотивом белков "спирального" типа.
2017.01 17 13:00:01
Пикотехнология белков, ДНК, РНК
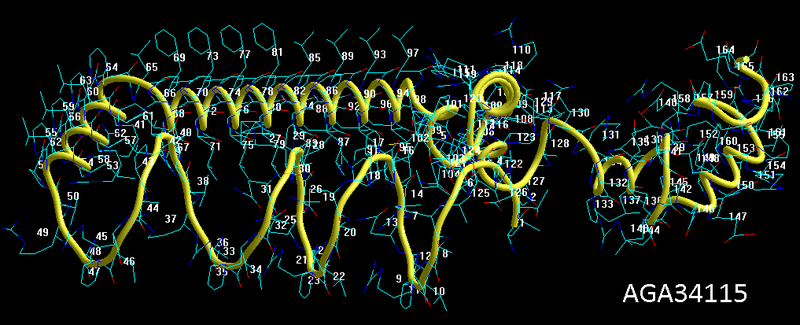
Кушелев: Начинаю тестировать новую версию Пикотех-2017:
>ENA|CAA30698|CAA30698.1 Escherichia coli hypothetical protein
ATGGCAGCTAAAGACGTAAAATTCGGTAACGACGCTCGTGTGAAAATGCTGCGCGGCGTA
AACGTACTGGCAGATGCAGTGAAAGTTACCCTCGGTCCAAAAGGCCGTAACGTAGTTCTG
GATAAATCTTTCGGTGCACCGACCATCACCAAAGATGGTGTTTCCGTTGCTCGTGAAATC
GAACTGGAAGACAAGTTCGAAAATATGGGTGCGCAGATGGTGAAAGAAGTTGCCTCTAAA
GCAAACGACGCTGCAGGCGACGGTACCACCACTGCAACCGTACTGGCTCAGGCTATCATC
ACTGAAGGTCTGAAAGCTGTTGCTGCGGGCATGAACCCGATGGACCTGAAACGTGGTATC
GACAAAGCGGTTACCGCTGCAGTTGAAGAACTGAAAGCGCTGTCCGTACCATGCTCTGAC
TCTAAAGCGATTGCTCAGGTTGGTACCATCTCCGCTAACTCCGACGAAACCGTAGGTAAA
CTGATCGCTGAAGCGATGGACAAAGTCGGTAAAGAAGGCGTTATCACCGTTGAAGACGGT
ACCGGTCTGCAGGACGAACTGGACGTGGTTGAAGGTATGCAGTTCGACCGTGGCTACCTG
TCTCCTTACTTCATCAACAAGCCGGAAACTGGCGCAGTAGAACTGGAAAGCCCGTTCATC
CTGCTGGCTGACAAGAAAATCTCCAACATCCGCGAAATGCTGCCGGTTCTGGAAGCTGTT
GCCAAAGCAGGCAAACCGCTGCTGATCATCGCTGAAGATGTAGAAGGCGAAGCGCTGGCA
ACTGCTGTTGTTAACACCATTCGTGGCATCGTGAAAGTCGCTGCGGTTAAAGCACCGGGC
TTCGGCGATCGTCGTAAAGCTATGCTGCAGGATATCGCAACCCTGACTGGCGGTACCGTG
ATCTCTGAAGAGATCGGTATGGAGCTGGAAAAAGCAACCCTGGAAGACCTGGGTCAGGCT
AAACGTGTTGTGATCAACAAAGACACCACCACTATCATCGATGGCGTGGGTGAAGAAGCT
GCAATCCAGGGCCGTGTTGCTCAGATCCGTCAGCAGATTGAAGAAGCAACTTCTGACTAC
GACCGTGAAAAACTGCAGGAACGCGTAGCGAAACTGGCAGGCGGCGTTGCAGTTATCAAA
GTGGGTGCTGCTACCGAAGTTGAAATGAAAGAGAAAAAAGCACGCGTTGAAGATGCCCTG
CACGCGACCCGTGCTGCGGTAGAAGAAGGCGTGGTTGCTGGTGGTGGTGTTGCGCTGATC
CGCGTAGCGTCTAAACTGGCTGACCTGCGTGGTCAGAACGAAGACCAGAACGTGGGTATC
AAAGTTGCACTGCGTGCAATGGAAGCTCCGCTGCGTCAGATCGTATTGAACTGCGGCGAA
GAACCGTCTGTTGTTGCTAACACCGTTAAAGGCGGCGACGGCAACTACGGTTACAACGCA
GCAACCGAAGAATACGGCAACATGATCGACATGGGTATCCTGGATCCAACCAAAGTAACT
CGTTCTGCTCTGCAGTACGCAGCTTCTGTGGCTGGCCTGATGATCACCACCGAATGCATG
GTTACCGACCTGCCGAAAAACGATGCAGCTGACTTAGGCGCTGCTGGCGGTATGGGCGGC
ATGGGTGGCATGGGCGGCATGATGTAA
Композиционный код4:
1,2,3,3,1,2,3,1,3,1,1,3,3,4,3,1,4,1,1,2,1,2,4,2,3,2,4,3,3,1,1,3,2,3,1,3,1,2,3,4,3,3,3,1,3,2,4,1,1,1,
3,3,3,3,1,3,3,3,3,1,3,4,3,1,1,1,3,3,1,3,4,1,1,4,3,3,3,1,3,3,2,1,1,3,2,1,1,3,1,1,3,2,1,2,4,3,1,3,1,1,
3,3,3,4,3,3,3,3,4,1,1,1,4,1,1,4,3,3,3,1,1,3,4,3,1,3,2,3,3,3,4,3,4,4,1,2,2,1,3,1,3,3,4,3,3,1,3,3,1,1,
1,3,1,1,1,3,1,2,3,3,4,1,3,3,4,1,1,3,1,3,3,3,1,3,1,1,3,3,1,3,1,3,4,1,1,3,4,1,4,3,3,3,1,1,1,1,3,1,1,4,
3,3,1,1,1,1,1,4,3,3,1,2,2,3,4,3,1,4,1,1,4,4,3,1,1,3,1,1,1,1,1,3,1,4,4,3,4,3,3,3,1,3,2,1,3,4,4,4,1,1,
3,3,3,2,3,1,3,4,4,2,3,3,3,3,1,1,3,3,1,1,4,3,1,3,4,3,3,2,4,1,1,1,3,3,3,3,3,1,4,1,3,1,2,1,4,3,1,3,1,4,
1,3,3,1,1,3,1,1,4,3,3,2,1,4,3,1,4,3,1,3,3,3,3,4,1,1,3,1,1,1,3,1,1,3,1,4,3,3,3,3,2,1,1,1,3,3,3,1,1,3,
1,1,3,3,3,2,3,3,1,1,1,3,3,3,4,1,3,1,2,4,3,4,2,1,1,3,2,3,1,3,4,3,3,3,1,3,3,3,1,3,1,3,3,2,1,3,3,3,1,4,
1,4,1,3,3,4,2,3,3,1,4,3,3,3,3,3,3,4,4,1,1,2,4,3,3,4,3,1,4,3,3,1,1,3,1,1,1,4,3,1,3,3,2,4,3,2,1,3,3,4,
4,3,1,1,2,1,1,1,1,3,3,4,3,3,3,3,1,1,3,3,1,1,1,1,1,1,3,1,1,2,2,1,3,3,1,1,1,1,1,1,1,3,1,4,3,2,1,3,2,3,
3,3,3,4,1,1,2,3,3,4,3,1,4,1,1,1,1,3,1,1,3,1,1,4,4,3,1,3,2,3,1,3,1,3,3,1,3,1,1,1,1,3,1,1,1,1,1,1,
Композиционный код6:
5,2,3,3,5,2,3,5,3,5,5,3,3,5,3,6,4,1,1,2,5,2,5,2,3,2,5,3,3,5,5,3,2,3,5,3,5,2,3,5,3,3,3,5,3,2,4,1,1,1,
3,3,3,3,5,3,3,3,3,5,3,5,3,5,5,5,3,3,5,3,4,1,6,4,3,3,3,5,3,3,2,5,5,3,2,5,5,3,5,5,3,2,5,2,5,3,5,3,5,5,
3,3,3,5,3,3,3,3,4,1,6,1,4,6,1,4,3,3,3,5,5,3,5,3,5,3,2,3,3,3,5,3,5,5,5,2,2,5,3,5,3,3,5,3,3,5,3,3,5,5,
5,3,5,5,5,3,5,2,3,3,5,5,3,3,5,5,5,3,5,3,3,3,5,3,5,5,3,3,5,3,5,3,5,5,5,3,4,4,4,3,3,3,6,1,1,1,3,4,4,4,
3,3,1,1,1,1,1,4,3,3,5,2,2,3,5,3,1,4,1,1,4,4,3,5,5,3,1,1,1,1,1,3,4,4,4,3,5,3,3,3,5,3,2,5,3,4,4,4,1,1,
3,3,3,2,3,5,3,5,5,2,3,3,3,3,5,5,3,3,4,4,4,3,5,3,5,3,3,2,4,1,1,1,3,3,3,3,3,5,5,5,3,5,2,5,5,3,5,3,5,5,
5,3,3,5,5,3,4,4,4,3,3,2,5,5,3,5,5,3,5,3,3,3,3,5,5,5,3,5,5,5,3,5,5,3,5,5,3,3,3,3,2,5,5,5,3,3,3,5,5,3,
5,5,3,3,3,2,3,3,5,5,5,3,3,3,5,5,3,5,2,5,3,5,2,5,5,3,2,3,5,3,5,3,3,3,5,3,3,3,5,3,5,3,3,2,5,3,3,3,1,4,
1,4,1,3,3,5,2,3,3,5,5,3,3,3,3,3,3,4,4,1,1,2,5,3,3,5,3,5,5,3,3,5,5,3,1,1,1,4,3,5,3,3,2,5,3,2,5,3,3,5,
5,3,5,5,2,1,1,1,1,3,3,5,3,3,3,3,5,5,3,3,1,1,1,1,1,1,3,5,5,2,2,5,3,3,1,1,1,6,1,1,6,3,5,5,3,2,5,3,2,3,
3,3,3,5,5,5,2,3,3,5,3,1,4,6,1,1,1,3,5,5,3,1,1,4,4,3,5,3,2,3,5,3,5,3,3,5,3,6,1,1,6,3,1,6,1,1,6,6,
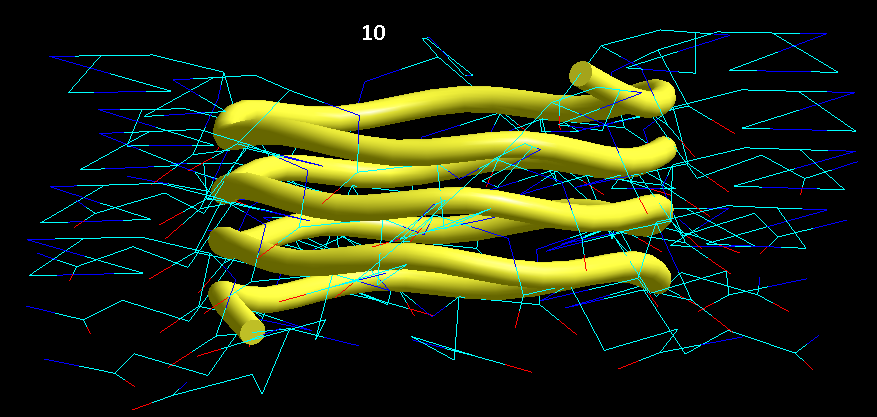
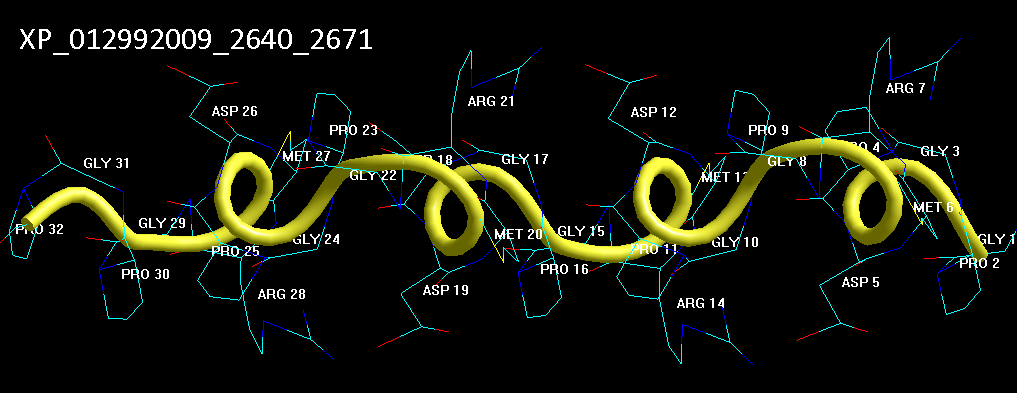
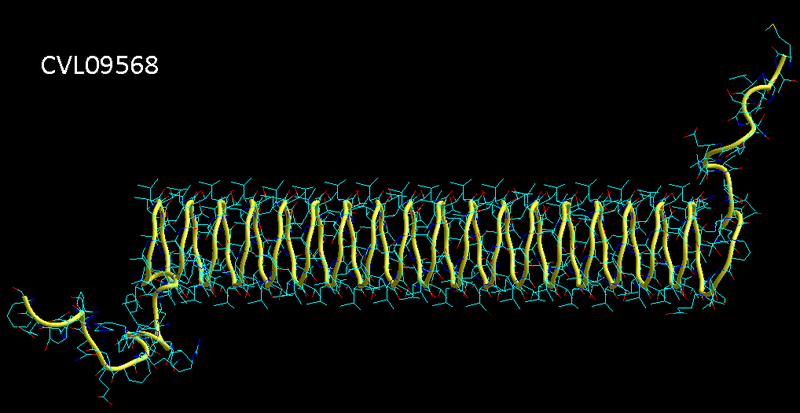
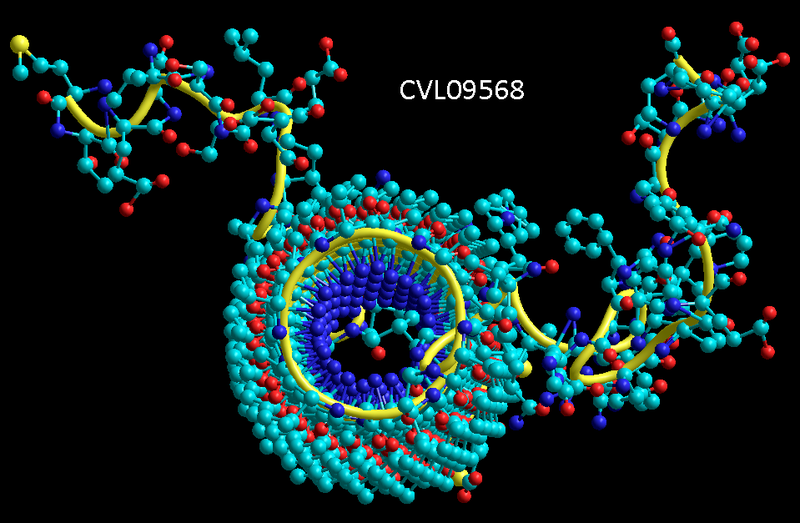
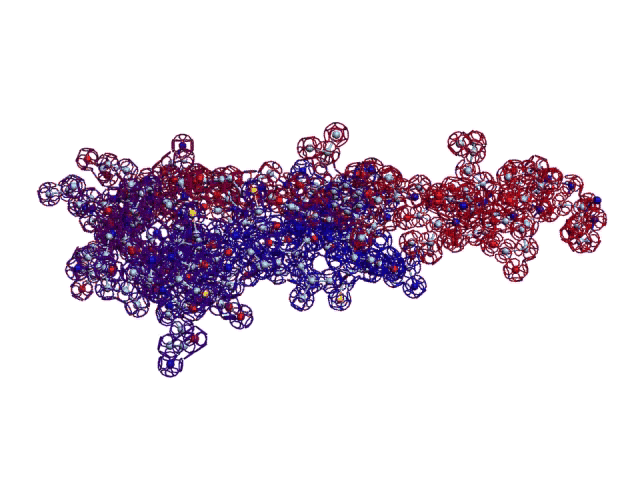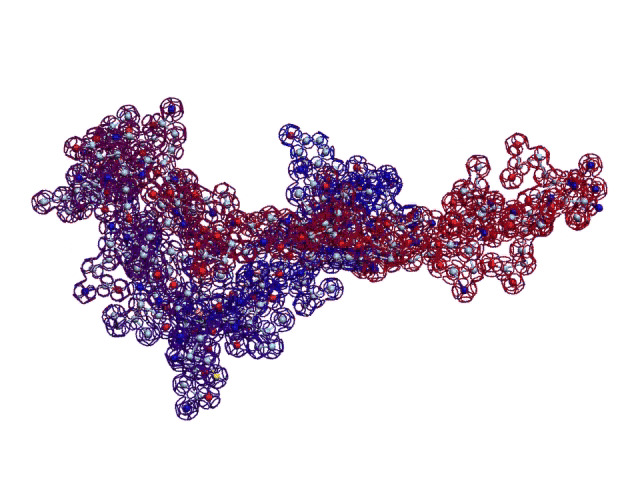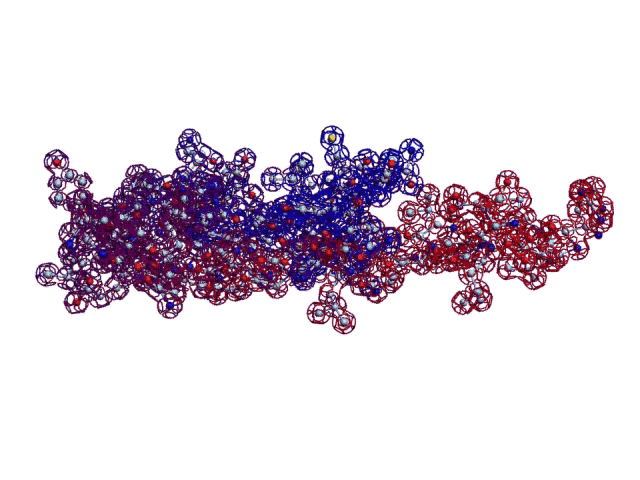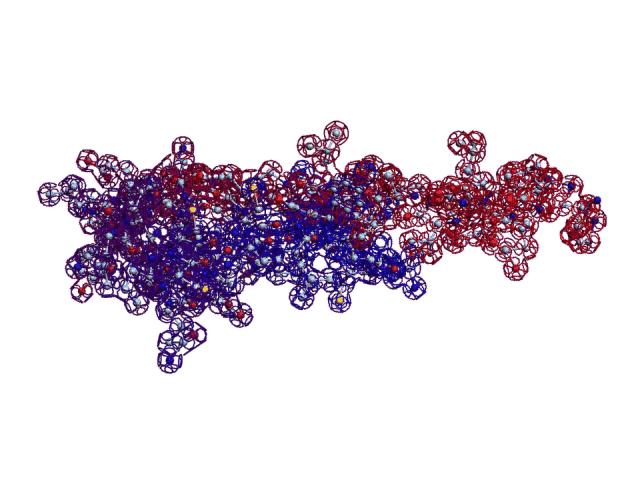
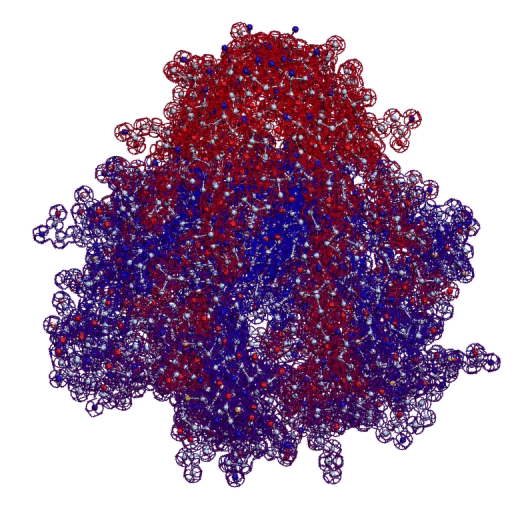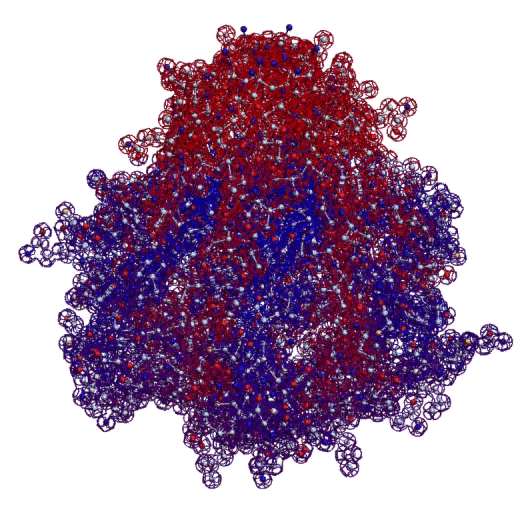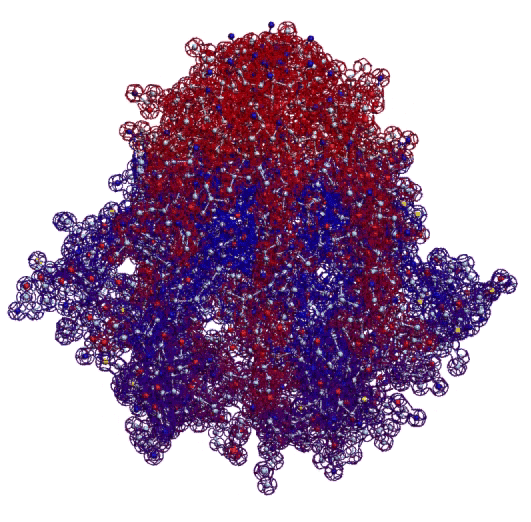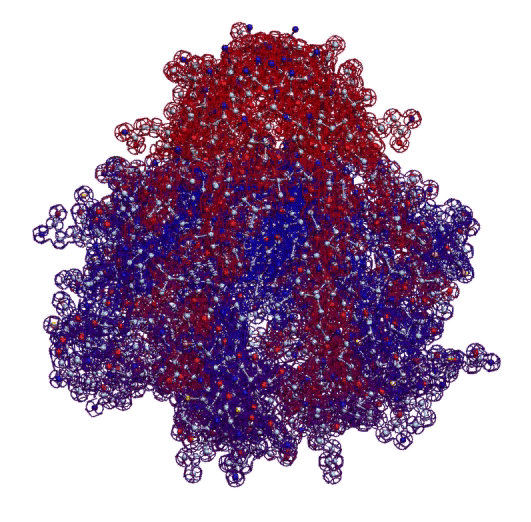
Вторичная структура (Пикотех-2017) (Программирование Ввалентин Яким)

2017.01.17 14:01:19
Татьяна Рясина пишет: Ахаха,
версия с шестью кодами больше напоминает диаграммы Виктории )))))) С длинными "перелётами" , у Виктории они серыми линиями обозначаются, между этими широкими блоками из толстых красных кирпичей
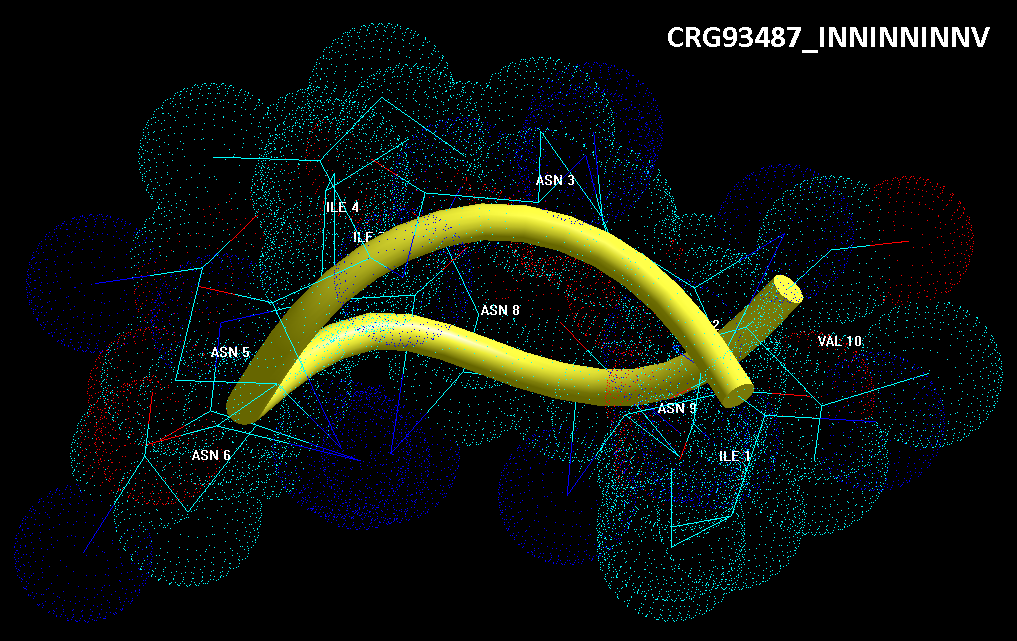
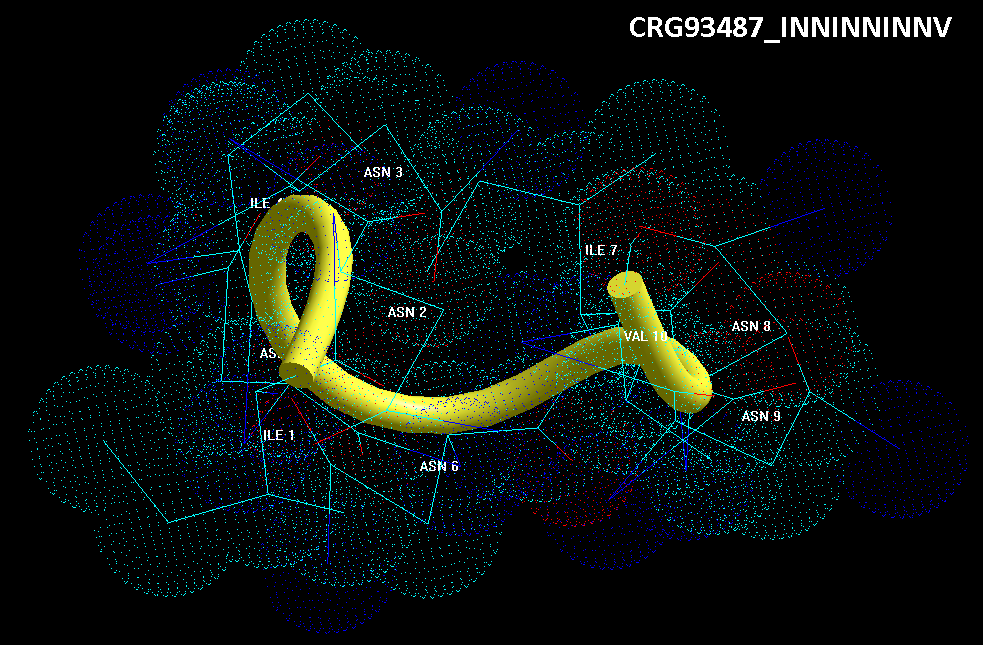
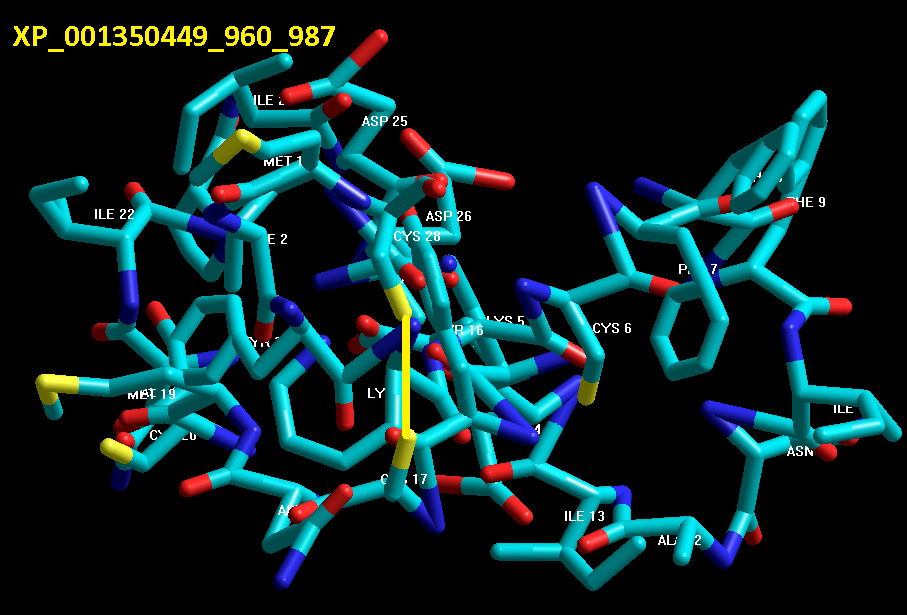
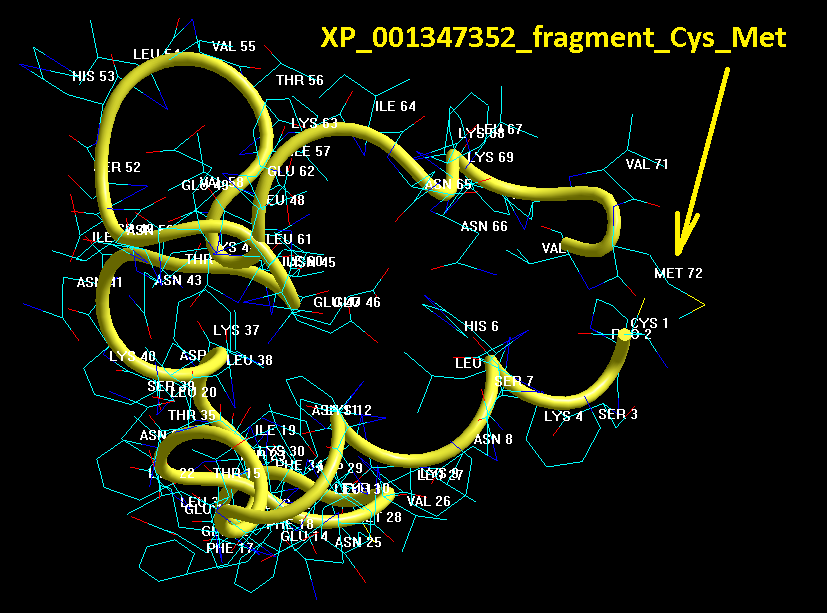
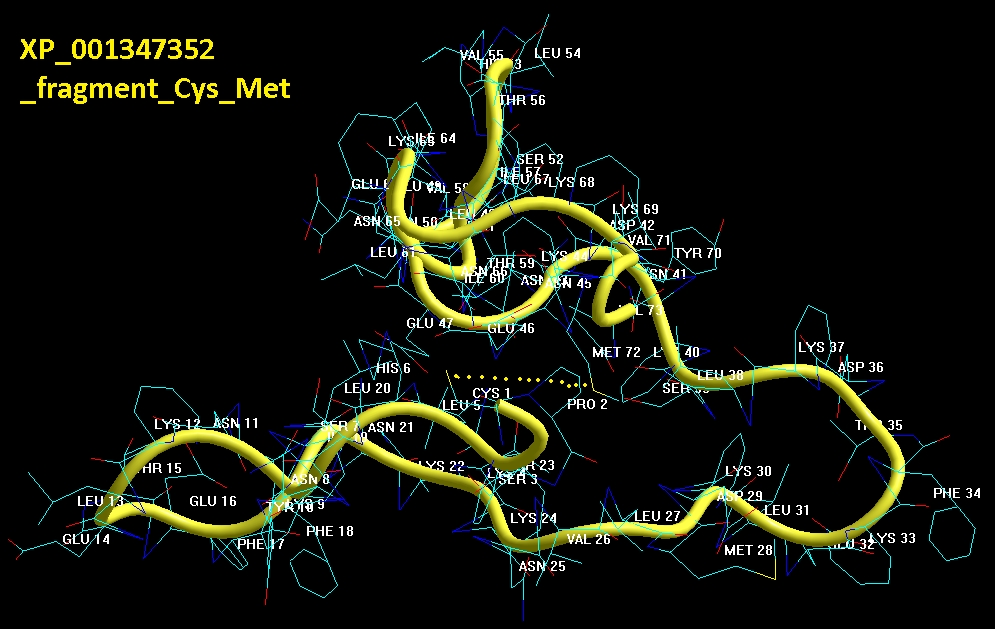
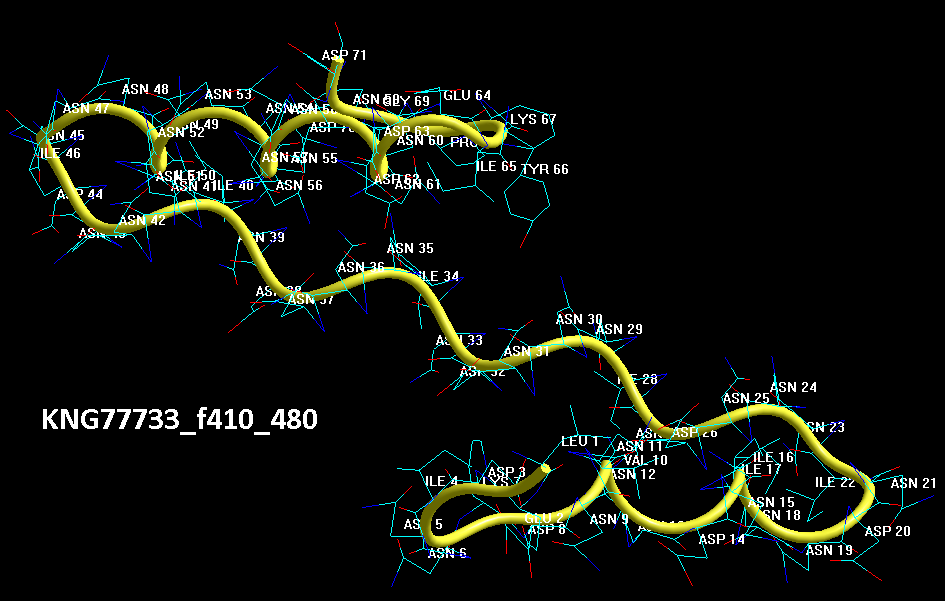
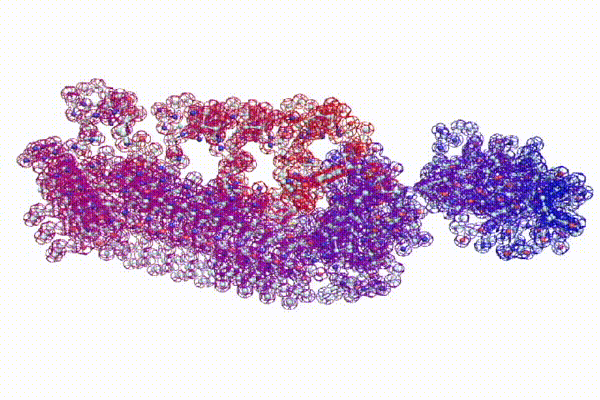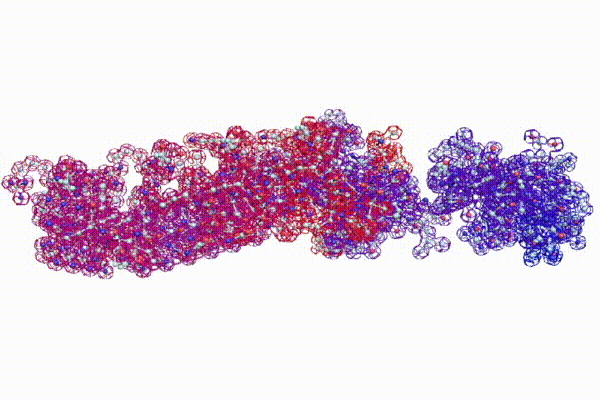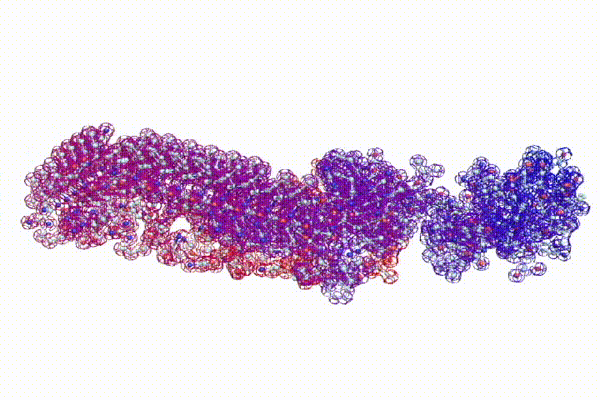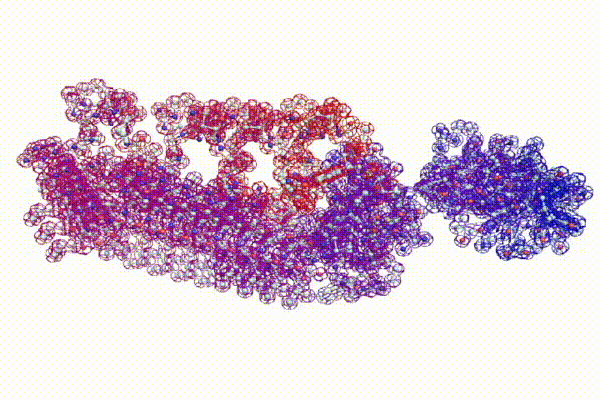
Кушелев: Верно! Только у этих "серых" линий конкретная структура есть. Виктория с этим и не спорит. Она говорит, что координаты в PDB файле конкретные, но такая структура называется "неопределённой", и она может легко меняться. Я же считаю, что "легко меняться" она не может, иначе по композиционному коду не могли бы замкнуться фрагменты лизоцимов:
В общей сложности построены модели 6 замкнутых через дисульфидные мостики участков.
1. Cys 95 - Cys 113 в человеческом лизоциме.
2. Cys 96 - Cys 117 в альтернативном человеческом лизоциме.
3. Cys 78 - Cys 82 в альтернативном человеческом лизоциме.
4. Cys 94 - Cys 98 в лизоциме куриного яйца
5. Met 1 - Cys 13 в мышином лизоциме
6. Met 1 - Cys 24 в лизоциме куриного яйца
Кстати, спиральные участки на схеме Var6 имеют переменную ширину, т.е. это коды гнутых спиралей 
2017.01.17 14:04:35
Татьяна Рясина пишет: А внешний вид построенный по 4 кодам и по 6 кодам отличаются сильно?
Кушелев: Для некоторых фрагментов, например, человеческого лизоцима, вообще не отличаются, т.к. там нет ни метионина, ни кодов "4".
А для белков, где есть Met или коды "4" в составе спиралей, форма белка может отличаться радикально. Ведь композиционный угол "4" отличается от композиционного угла "1" . И угол Met в спирали другой. Не такой, как между спиралями.
2017.01.17 16:25:41
Пикотехнология белков, ДНК, РНК
http://www.rcsb.org/pdb/explore/explore … ureId=5HK5
http://www.uniprot.org/uniprot/O88273
http://www.ebi.ac.uk/ena/data/view/BAA29038
>ENA|BAA29038|BAA29038.1 Mus musculus (house mouse) PRDC
ATGTTCTGGAAGCTCTCGCTGACCTTGCTCCTGGTGGCTGTGCTGGTAAAGGTAGCTGAA
ACACGGAAGAACCGGCCTGCGGGCGCCATCCCCTCGCCTTACAAGGATGGTAGCAGCAAC
AACTCAGAGAGGTGGCATCACCAGATCAAGGAGGTGCTGGCCTCCAGCCAGGAGGCCCTG
GTAGTCACCGAGCGCAAGTACCTCAAGAGTGACTGGTGCAAGACGCAGCCTCTGCGGCAG
ACAGTGAGCGAGGAGGGTTGCCGCAGCCGCACCATCCTCAACCGCTTCTGCTACGGCCAG
TGCAACTCCTTCTACATCCCGCGACACGTGAAGAAGGAGGAGGACTCCTTCCAATCCTGC
GCTTTCTGCAAGCCCCAGCGTGTCACCTCTGTCATCGTAGAGCTCGAATGCCCGGGTCTC
GACCCACCTTTCCGAATCAAGAAAATCCAGAAGGTGAAGCATTGCCGGTGCATGTCAGTG
AACCTGAGTGACTCCGACAAGCAGTGA

2017.01.17 16:28:16
http://www.uniprot.org/uniprot/P43026
http://www.ebi.ac.uk/ena/data/view/CAA56874
>ENA|CAA56874|CAA56874.1 Homo sapiens (human) hypothetical protein
ATGAGACTCCCCAAACTCCTCACTTTCTTGCTTTGGTACCTGGCTTGGCTGGACCTGGAA
TTCATCTGCACTGTGTTGGGTGCCCCTGACTTGGGCCAGAGACCCCAGGGGACCAGGCCA
GGATTGGCCAAAGCAGAGGCCAAGGAGAGGCCCCCCCTGGCCCGGAACGTCTTCAGGCCA
GGGGGTCACAGCTATGGTGGGGGGGCCACCAATGCCAATGCCAGGGCAAAGGGAGGCACC
GGGCAGACAGGAGGCCTGACACAGCCCAAGAAGGATGAACCCAAAAAGCTGCCCCCCAGA
CCGGGCGGCCCTGAACCCAAGCCAGGACACCCTCCCCAAACAAGGCAGGCTACAGCCCGG
ACTGTGACCCCAAAAGGACAGCTTCCCGGAGGCAAGGCACCCCCAAAAGCAGGATCTGTC
CCCAGCTCCTTCCTGCTGAAGAAGGCCAGGGAGCCCGGGCCCCCACGAGAGCCCAAGGAG
CCGTTTCGCCCACCCCCCATCACACCCCACGAGTACATGCTCTCGCTGTACAGGACGCTG
TCCGATGCTGACAGAAAGGGAGGCAACAGCAGCGTGAAGTTGGAGGCTGGCCTGGCCAAC
ACCATCACCAGCTTTATTGACAAAGGGCAAGATGACCGAGGTCCCGTGGTCAGGAAGCAG
AGGTACGTGTTTGACATTAGTGCCCTGGAGAAGGATGGGCTGCTGGGGGCCGAGCTGCGG
ATCTTGCGGAAGAAGCCCTCGGACACGGCCAAGCCAGCGGCCCCCGGAGGCGGGCGGGCT
GCCCAGCTGAAGCTGTCCAGCTGCCCCAGCGGCCGGCAGCCGGCCTCCTTGCTGGATGTG
CGCTCCGTGCCAGGCCTGGACGGATCTGGCTGGGAGGTGTTCGACATCTGGAAGCTCTTC
CGAAACTTTAAGAACTCGGCCCAGCTGTGCCTGGAGCTGGAGGCCTGGGAACGGGGCAGG
GCCGTGGACCTCCGTGGCCTGGGCTTCGACCGCGCCGCCCGGCAGGTCCACGAGAAGGCC
CTGTTCCTGGTGTTTGGCCGCACCAAGAAACGGGACCTGTTCTTTAATGAGATTAAGGCC
CGCTCTGGCCAGGACGATAAGACCGTGTATGAGTACCTGTTCAGCCAGCGGCGAAAACGG
CGGGCCCCACTGGCCACTCGCCAGGGCAAGCGACCCAGCAAGAACCTTAAGGCTCGCTGC
AGTCGGAAGGCACTGCATGTCAACTTCAAGGACATGGGCTGGGACGACTGGATCATCGCA
CCCCTTGAGTACGAGGCTTTCCACTGCGAGGGGCTGTGCGAGTTCCCATTGCGCTCCCAC
CTGGAGCCCACGAATCATGCAGTCATCCAGACCCTGATGAACTCCATGGACCCCGAGTCC
ACACCACCCACCTGCTGTGTGCCCACGCGGCTGAGTCCCATCAGCATCCTCTTCATTGAC
TCTGCCAACAACGTGGTGTATAAGCAGTATGAGGACATGGTCGTGGAGTCGTGTGGCTGC
AGGTAG

2017.01.17 18:39:29
Пикотехнология белков, ДНК, РНК
>NM_207361.5 Homo sapiens FRAS1 related extracellular matrix protein 2 (FREM2), mRNA
GGGATTTCCCTCAAAGCCCAGGGTCCGGGGACCTGGAAAGTAGAAGTGGAGGGATTCAATTCTCCGCGCG
ATTGAGGCGCTAGCGGCGGAGCTGGACGGCCTGGGAAGGCTTCGGCTCCTCGGCTGCGGCTCCAGCCCGG
ACGGCGCCGCGCAACTTTGCCATCCTTCTGGCCCAGCCCCGGCTACAGGAGGACCCCGCGGGCAACGCGC
GGAGTTCCTGGCACTTCCCGGCGGTGTCTCTTGTTGTCTGCCCGGGGACCGACTTCGCATGCTCTCAGGC
TGACCTGTCCAAGCCCGAACACCGGGACCATGCACTCAGCCGGGACTCCCGGGTTATCCTCGCGCCGGAC
AGGCAACTCCACCAGCTTTCAACCAGGACCGCCACCGCCGCCCCGGCTGCTGCTGCTGCTGCTGCTTCTC
CTGTCACTGGTAAGCCGCGTCCCGGCACAGCCCGCTGCCTTCGGCAGGGCGTTGCTGTCCCCTGGTCTCG
CGGGGGCTGCAGGGGTCCCTGCTGAGGAGGCCATAGTGCTGGCGAACCGCGGACTCCGGGTGCCTTTCGG
CCGTGAAGTCTGGCTGGATCCCCTGCATGACCTGGTGTTGCAGGTGCAGCCCGGGGACCGCTGCGCGGTT
TCGGTACTAGACAACGACGCACTGGCCCAGCGACCGGGCCGCCTGAGTCCCAAGCGCTTCCCGTGCGACT
TTGGCCCTGGCGAGGTGCGCTACTCTCACCTGGGCGCGCGCAGCCCGTCTCGGGACCGCGTCCGGCTGCA
GCTGCGCTATGACGCGCCCGGAGGGGCAGTAGTGCTACCACTGGTACTGGAGGTGGAGGTGGTCTTCACC
CAGCTGGAGGTTGTGACTCGGAACTTGCCTCTGGTCGTGGAAGAGCTGCTGGGGACCAGCAATGCCCTGG
ACGCGCGGAGCCTGGAGTTCGCCTTCCAGCCCGAGACAGAGGAGTGCCGCGTGGGCATCCTGTCCGGCTT
GGGCGCGCTGCCTCGCTATGGAGAACTCCTCCACTACCCGCAGGTCCCTGGAGGAGCCAGAGAGGGAGGC
GCCCCGGAGACTCTCCTGATGGACTGCAAAGCTTTCCAGGAACTAGGCGTGCGCTATCGCCACACAGCCG
CCAGTCGCTCACCAAACAGGGACTGGATACCCATGGTGGTGGAGCTGCGTTCACGAGGGGCTCCTGTGGG
CAGCCCTGCTTTGAAACGCGAGCACTTCCAGGTTCTGGTGAGGATCCGAGGAGGGGCCGAGAACACTGCA
CCCAAGCCCAGTTTCGTGGCCATGATGATGATGGAGGTGGACCAGTTTGTACTGACGGCCCTGACCCCAG
ACATGCTGGCAGCCGAGGATGCTGAGTCTCCCTCTGACCTGTTGATCTTCAACCTTACTTCTCCATTCCA
GCCTGGCCAGGGCTACTTGGTGAGCACCGATGATCGCAGCCTGCCCCTTTCCTCCTTCACTCAGAGGGAT
CTGCGGCTCCTGAAGATTGCCTACCAGCCCCCTTCTGAAGACTCTGACCAGGAGCGCCTCTTTGAACTGG
AATTGGAGGTAGTGGATCTAGAAGGAGCAGCTTCAGACCCTTTTGCCTTCATGGTAGTGGTGAAGCCCAT
GAACACAATGGCTCCGGTGGTCACCCGGAATACCGGTCTTATTCTCTATGAGGGTCAGTCTCGGCCCCTC
ACAGGCCCTGCAGGCAGTGGTCCGCAAAACTTGGTCATCAGCGATGAGGATGACCTAGAAGCAGTGCGGC
TAGAGGTGGTGGCTGGGCTCCGGCATGGTCACCTTGTCATTCTGGGTGCTTCCAGTGGCAGCTCTGCTCC
CAAGAGCTTTACAGTGGCTGAGCTGGCAGCCGGCCAGGTGGTCTACCAGCATGATGACAGAGACGGCTCG
CTGAGCGACAACCTGGTGCTTCGCATGGTGGATGGAGGAGGCAGGCACCAGGTACAGTTTCTGTTCCCCA
TCACCTTAGTGCCTGTGGATGACCAGCCACCTGTTCTCAATGCCAACACGGGGCTGACACTGGCAGAGGG
TGAAACAGTGCCCATCCTGCCCCTTTCCCTGAGTGCAACTGACATGGATTCAGATGATTCTCTGCTGCTT
TTTGTGCTGGAGTCACCCTTCTTAACTACGGGGCATCTGCTTCTCCGCCAAACTCACCCTCCCCATGAGA
AGCAGGAACTTCTCAGAGGCCTTTGGAGGAAGGAGGGGGCATTTTATGAGCGAACAGTGACAGAGTGGCA
GCAGCAGGACATAACAGAGGGCAGGCTGTTCTATAGACACTCTGGGCCCCATAGTCCTGGGCCAGTCACA
GACCAGTTCACATTTAGAGTCCAGGATAACCATGACCCTCCTAATCAGTCCGGGCTACAGCGGTTTGTGA
TTCGTATCCATCCTGTGGATCGCCTCCCTCCGGAGCTGGGCAGTGGCTGTCCCCTTCGTATGGTGGTACA
GGAATCCCAGCTCACACCACTGAGGAAGAAGTGGCTGCGCTACACTGACCTGGACACAGATGACCGAGAA
CTACGTTACACAGTGACTCAGCCCCCCACAGACACAGACGAAAATCACCTGCCAGCCCCACTGGGTACCT
TGGTCTTGACTGACAACCCCTCAGTCGTGGTGACCCATTTTACCCAAGCCCAGATCAACCATCATAAAAT
TGCTTACAGACCCCCGGGTCAAGAACTGGGCGTGGCTACTCGAGTGGCCCAGTTCCAGTTCCAGGTGGAA
GACCGAGCTGGGAATGTGGCTCCAGGTACCTTTACCCTTTACTTGCATCCCGTGGACAACCAGCCACCTG
AGATCCTCAACACCGGCTTCACTATTCAGGAGAAGGGTCACCACATCCTGAGTGAGACAGAGTTGCACGT
GAATGATGTAGACACTGATGTTGCCCATATCTCTTTCACTCTCACTCAGGCACCCAAACATGGCCACATG
AGAGTGTCTGGACAGATCCTGCATGTAGGGGGTCTCTTCCACTTGGAGGACATAAAACAGGGCCGAGTTT
CCTATGCCCATAATGGGGACAAGTCCCTGACTGATAGCTGCTCCTTGGAAGTCAGTGACAGACATCATGT
GGTGCCCATCACTCTCAGAGTAAATGTCCGGCCAGTGGATGATGAAGTGCCCATACTGAGCCATCCTACT
GGCACTCTGGAGTCCTATCTAGATGTCTTAGAAAATGGGGCTACTGAAATCACTGCCAATGTTATTAAGG
GGACCAATGAGGAAACTGATGACTTGATGTTGACTTTCCTCTTGGAAGATCCACCTTTGTATGGGGAAAT
CTTGGTCAATGGCATTCCAGCAGAGCAGTTTACTCAAAGGGACATCTTGGAGGGCTCTGTTGTATATACC
CACACCAGTGGTGAGATAGGCCTATTGCCTAAAGCGGATTCTTTTAACCTGAGTCTGTCAGATATGTCTC
AAGAATGGAGAATTGGTGGCAATACTATCCAAGGAGTTACTATATGGGTGACCATCCTGCCTGTTGATAG
CCAGGCCCCAGAAATCTTTGTAGGTGAACAGTTGATAGTAATGGAAGGTGATAAAAGTGTTATAACATCA
GTGCATATAAGTGCTGAAGATGTCGACTCCCTGAATGATGACATCTTGTGCACTATAGTTATTCAGCCTA
CTTCAGGTTATGTTGAAAACATTTCTCCAGCACCAGGCTCTGAGAAATCAAGAGCAGGGATTGCCATAAG
TGCTTTCAACTTGAAAGATCTCAGGCAGGGCCACATAAACTATGTCCAGAGTGTCCATAAAGGGGTGGAA
CCTGTGGAGGACCGATTTGTATTTCGTTGTTCTGATGGCATTAACTTTTCAGAGAGACAGTTCTTCCCCA
TTGTAATCATTCCCACCAATGATGAACAGCCAGAGATGTTTATGAGAGAATTTATGGTGATGGAAGGCAT
GAGTCTGGTAATTGATACACCCATTCTCAATGCTGCTGATGCTGATGTTCCCCTGGATGATTTAACTTTC
ACTATTACCCAATTCCCCACTCATGGTCACATCATGAATCAGCTGATAAATGGCACGGTTTTGGTCGAAA
GCTTCACCTTGGATCAGATCATAGAGAGTTCCAGCATTATTTATGAGCATGATGACTCCGAGACCCAGGA
AGACAGTTTTGTGATTAAACTAACAGATGGGAAGCACTCTGTGGAAAAGACGGTCCTCATTATAGTTATC
CCTGTTGATGATGAGACGCCCAGAATGACTATCAATAATGGACTAGAAATAGAAATTGGGGATACCAAGA
TTATCAACAACAAAATATTAATGGCAACAGATTTAGATTCAGAAGACAAATCTTTGGTTTATATTATTCG
TTATGGGCCAGGACATGGCTTATTACAGAGACGAAAACCTACTGGTGCCTTTGAAAATATCACACTGGGC
ATGAATTTTACCCAGGATGAAGTAGACAGAAACTTAATTCAGTATGTCCATTTGGGGCAAGAGGGCATTC
GGGACCTAATTAAATTTGATGTGACTGATGGAATAAATCCCCTCATAGATCGTTACTTTTATGTGTCCAT
CGGGAGCATTGACATTGTCTTCCCTGATGTGATAAGTAAGGGAGTGTCCTTGAAAGAAGGTGGCAAAGTC
ACTCTTACAACAGACCTACTAAGCACTAGTGACTTGAACAGTCCTGATGAAAACTTGGTTTTTACCATCA
CCAGGGCTCCCATGCGAGGTCACCTGGAATGCACGGATCAGCCTGGTGTGTCCATCACGTCTTTCACTCA
GCTGCAACTGGCTGGAAACAAAATCTACTACATCCACACAGCTGATGATGAAGTGAAAATGGACAGTTTT
GAGTTTCAAGTCACCGATGGACGTAACCCTGTCTTTCGGACATTCCGTATCTCCATTAGCGATGTGGACA
ATAAAAAGCCAGTGGTCACCATCCACAAGCTGGTTGTCAGTGAAAGTGAAAACAAGCTGATTACTCCTTT
TGAGCTCACTGTCGAAGACAGAGATACTCCTGACAAGCTCCTGAAATTCACTATCACCCAGGTGCCTATT
CATGGCCATCTCCTATTCAACAATACCAGACCTGTCATGGTTTTTACCAAGCAAGACTTGAATGAAAACT
TAATCAGCTACAAACATGATGGCACTGAGTCAAGTGAAGATAGCTTCTCCTTCACAGTGACTGATGGCAC
CCATACAGACTTCTATGTTTTTCCTGATACGGTGTTTGAAACAAGGAGACCCCAAGTGATGAAGATCCAG
GTCTTGGCTGTTGACAACAGTGTCCCCCAAATCGCAGTGAATAAGGGGGCCTCTACACTTCGCACTCTAG
CCACTGGCCACTTGGGGTTCATGATCACAAGCAAAATATTGAAAGTGGAGGACAGAGACAGCTTACACAT
TTCTCTTAGATTTATCGTGACAGAGGCCCCTCAACATGGATATCTTCTCAACCTGGACAAAGGCAACCAC
AGCATCACTCAGTTCACACAAGCTGACATTGATGACATGAAAATATGCTATGTCTTAAGAGAAGGGGCTA
ATGCCACAAGTGATATGTTCTATTTTGCAGTTGAAGATGGTGGTGGAAACAAGTTAACGTACCAGAATTT
TCGTCTGAATTGGGCATGGATCTCCTTTGAAAAGGAATATTACCTGGTCAATGAGGACTCCAAATTTCTA
GATGTTGTTCTTAAACGTAGAGGTTACTTGGGAGAAACTTCTTTTATAAGTATTGGCACAAGAGACAGAA
CTGCAGAAAAAGACAAAGACTTCAAGGGCAAAGCACAGAAACAAGTGCAGTTCAACCCAGGCCAGACCAG
GGCCACATGGCGAGTGCGGATCCTGAGTGATGGGGAGCATGAGCAGTCTGAAACCTTTCAGGTGGTACTC
TCAGAGCCCGTGCTGGCTGCCTTGGAATTCCCCACAGTCGCCACTGTTGAGATCGTTGATCCAGGAGATG
AGCCAACTGTGTTTATTCCCCAGTCCAAATACTCCGTTGAAGAAGATGTTGGTGAGCTGTTCATTCCCAT
CAGGAGGAGCGGAGATGTGAGCCAGGAGTTGATGGTGGTCTGTTATACCCAACAAGGAACAGCAACTGGA
ACTGTGCCGACTTCCGTGTTGTCTTACTCTGATTACATATCCAGGCCTGAGGACCACACCAGTGTTGTCC
GCTTTGACAAAGATGAACGGGAGAAACTGTGTCGGATAGTCATAATTGATGACTCTTTGTACGAGGAGGA
GGAAACCTTCCATGTCCTTCTGAGCATGCCCATGGGGGGAAGAATCGGATCAGAGTTCCCAGGGGCTCAA
GTTACAATCGTTCCTGACAAAGATGATGAACCCATCTTTTACTTCGGTGATGTGGAATACTCTGTGGATG
AGAGTGCTGGCTATGTGGAAGTGCAGGTGTGGAGAACGGGCACTGACCTGTCCAAGTCTTCTAGTGTCAC
AGTGAGGTCTCGGAAAACAGATCCTCCCTCTGCAGATGCTGGAACAGACTATGTGGGCATCAGCCGTAAT
TTAGATTTTGCACCTGGAGTCAACATGCAGCCTGTTCGTGTTGTCATTCTGGATGACCTTGGACAACCAG
CGCTGGAGGGAATTGAGAAATTTGAACTGGTGCTTCGCATGCCTATGAACGCAGCCCTTGGCGAGCCCAG
CAAAGCCACAGTGTCCATAAATGACTCTGTCTCCGATTTGCCTAAGATGCAATTCAAAGAACGAATATAT
ACTGGCAGCGAAAGTGATGGGCAGATAGTTACAATGATCCATAGGACTGGGGATGTCCAGTACAGATCTT
CAGTGAGATGCTACACCCGGCAGGGGTCTGCACAGGTGATGATGGACTTTGAAGAACGCCCAAACACTGA
TACCTCCATCATCACATTCCTCCCTGGTGAGACAGAAAAGCCCTGCATTCTTGAGCTGATGGACGATGTG
CTCTATGAGGAGGTAGAGGAGCTCCGCCTGGTACTCGGCACTCCACAAAGCAACTCTCCCTTTGGGGCTG
CAGTTGGTGAACAAAATGAAACTCTCATAAGGATCCGAGATGATGCTGATAAGACTGTTATTAAATTTGG
AGAAACCAAATTTAGTGTCACTGAACCCAAAGAACCTGGAGAGTCGGTGGTTATAAGAATTCCAGTGATT
CGCCAAGGAGACACTTCAAAGGTTTCCATTGTGAGAGTCCACACCAAGGATGGCTCGGCCACCTCTGGAG
AAGACTACCACCCTGTGTCAGAAGAAATTGAGTTTAAGGAAGGGGAAACCCAGCACGTGGTTGAAATCGA
AGTTACCTTTGACGGGGTGAGAGAGATGAGAGAGGCCTTCACTGTTCACCTAAAACCTGATGAAAATATG
ATAGCAGAGATGCAGTTGACGAAAGCCATTGTGTACATAGAAGAAATGAGCAGCATGGCAGATGTCACTT
TTCCTTCTGTCCCTCAAATTGTATCCCTGTTGATGTATGACGACACTTCCAAAGCTAAGGAGAGTGCTGA
ACCCATGTCTGGCTATCCTGTCATCTGTATCACAGCTTGCAACCCCAAATATTCAGACTACGATAAAACA
GGCTCTATCTGTGCAAGTGAGAACATCAATGACACTTTGACGCGGTACCGGTGGCTGATTAGTGCACCTG
CGGGCCCTGACGGTGTGACCAGCCCTATGAGAGAAGTGGACTTCGACACCTTTTTTACGTCATCCAAGAT
GGTCACACTGGACTCCATATACTTTCAGCCTGGCTCCCGGGTACAGTGCGCAGCTCGTGCTGTGAACACC
AATGGGGATGAAGGCCTGGAGCTCATGAGCCCTATTGTAACCATCAGCAGAGAAGAAGGTCTTTGTCAGC
CCCGTGTACCTGGGGTTGTTGGAGCAGAGCCGTTCTCAGCTAAATTGCGCTACACAGGCCCTGAGGATGC
AGACTACACAAACCTTATCAAGCTCACTGTCACAATGCCACACATAGATGGCATGCTCCCCGTGATCTCC
ACTAGAGAGCTTTCCAACTTTGAGCTCACCCTCAGCCCTGATGGCACAAGAGTTGGAAACCACAAGTGCT
CCAACCTCCTGGATTATACTGAAGTGAAGACTCATTATGGTTTCTTGACTGATGCTACCAAAAATCCAGA
AATAATTGGAGAGACATATCCTTACCAGTACAGCTTGTCCATCAGAGGTTCCACTACCTTGCGCTTCTAC
CGGAACCTGAACCTAGAGGCCTGTTTATGGGAGTTCGTTAGCTACTATGACATGTCAGAACTCCTTGCTG
ACTGTGGTGGCACCATTGGAACAGATGGACAGGTCCTAAACCTAGTGCAGTCCTATGTGACCCTTCGAGT
CCCTCTGTATGTTTCCTACGTGTTCCATTCCCCCGTGGGGGTAGGAGGCTGGCAGCATTTTGACTTGAAG
TCAGAGCTTCGTCTAACTTTTGTGTACGACACCGCCATCTTGTGGAATGATGGAATTGGCAGCCCCCCAG
AGGCTGAACTTCAAGGTTCTCTCTATCCAACCAGCATGCGCATCGGTGATGAGGGGCGCTTGGCCGTGCA
CTTCAAGACAGAGGCTCAGTTCCATGGCTTATTTGTGCTGTCACATCCCGCATCCTTTACAAGCTCAGTG
ATCATGTCAGCTGATCATCCAGGCCTGACATTTTCCCTCCGCCTCATAAGGAGTGAACCAACCTATAACC
AGCCAGTACAGCAGTGGAGCTTTGTCTCTGACTTTGCCGTACGTGACTACTCAGGGACCTATACTGTGAA
GCTGGTGCCATGCACTGCCCCATCACATCAGGAATACCGCCTGCCAGTCACCTGCAACCCCAGAGAACCT
GTCACCTTTGACCTTGACATCCGATTCCAACAGGTCAGTGATCCAGTGGCTGCTGAGTTTAGCTTGAACA
CCCAAATGTACCTGCTCTCTAAGAAGAGTCTCTGGTTGTCTGATGGATCCATGGGATTCGGGCAAGAGAG
TGATGTTGCTTTTGCAGAAGGTGATATAATTTATGGTCGTGTCATGGTGGATCCTGTCCAGAATCTGGGT
GACTCCTTTTACTGCAGCATTGAGAAGGTGTTTCTATGCACTGGAGCTGATGGCTATGTTCCCAAGTATA
GTCCAATGAATGCAGAATATGGCTGCTTAGCCGACTCTCCTTCACTCTTATATAGATTTAAAATTGTGGA
CAAAGCTCAGCCAGAGACACAAGCGACCAGTTTTGGAAATGTCCTATTTAATGCCAAACTAGCAGTGGAT
GACCCTGAAGCCATTCTCTTAGTGAATCAGCCTGGATCTGATGGATTTAAAGTCGACTCAACACCACTCT
TTCAGGTCGCTCTAGGCCGAGAATGGTATATACATACGATCTATACAGTGAGATCGAAAGACAATGCCAA
TCGAGGTATTGGCAAAAGAAGTGTGGAGTACCATTCTCTGGTGAGTCAAGGAAAGCCCCAATCCACCACC
AAGAGCCGGAAGAAGAGAGAGATCAGGAGCACACCCTCACTGGCATGGGAGATTGGTGCTGAAAACAGTC
GAGGAACAAACATCCAGCACATTGCCCTGGACCGCACCAAGAGGCAGATCCCCCATGGGAGAGCACCTCC
AGATGGCATCCTCCCCTGGGAGCTCAACAGCCCCAGCTCTGCAGTCAGCCTGGTCACTGTGGTGGGAGGC
ACCACGGTAGGGTTACTCACCATCTGCCTCACTGTCATTGCAGTGCTGATGTGCAGGGGCAAGGAAAGTT
TCAGGGGGAAGGATGCCCCGAAAGGCTCCAGCAGCAGTGAGCCCATGGTGCCCCCACAGAGCCATCACAA
TGACAGCTCAGAAGTTTGATGACTGCAGGTAGAATTCAACCTTTTCCGTAAGTGCCTCGGAAAAGATCAC
AATGGAACCTTAAATACTTCTGGTAAACCATAGAGAATGGAGGATGGCTGTGATGAAGCTGCTTAGAGAA
TATGACTGTACTAATGGAGTTTGATTTGAATTCTCCATACTCTTTTTTGCATCAAAGGACAAATTAAGGC
ATCTTTCCTCTTGCCCCAAAGGCAGCAACAACGTACTCATATGTACACAGAGCCATGATGTGAGGAATGT
ACTCCTCATTTTAGACATATTCTCTATGCAGTGGAGATAAATCTATTAAAAGGTGCTAACAGACTCTCTT
ACAAGTGTAAGAGGAATCTACTGTTGCTATGTTAGTGTGAATGTTGAAGGTGCAATTATCACATTGTTTA
TTAATTGTATAACAGATTATTACTAGAAAGGTTTTTGTTGCTAGTCTGGAAAACTGGTGAACACACTGTT
TATTATGACAAGTTTTCAAATAGGTGAAGACAGCATAGAATTATGACCAGGGTGGCTCAACCCACAAATC
AACTGATCTACTACTTGGGAGACAGTGGAAGGAAAGATAGAGGGAAGGAAGTTCACTCACTTGAATTTGA
ATTGCTTCTCATTCTCATCAGACTCTCCTTGTTTTGTTTGCATATTTAATTTTAAATTAAACCAAAGAAT
ATGTTAATTCTGAAAGAGATTTTTAAGAGATGCTGCTCTGTTTTGGCATATAGTGAGTAATGGTAGAGCT
CTTGCATGGTAATATACCTTATTGGTGCTCAACATTTGTGGGAAATTAGAGGGTTGGTGAGATTTTGGTG
ATGAATAAATGCCATAGAGTTAGAGTCACTACAAGACAATGTTCTCTAAGAACTGAGCCTAGATGTTTGC
AGTATTGAACCCATAAATGATAATAAGAAACATTGTTACAGATGGTGAAGGGAGAAAGTGGTATTTATTA
ATATAATGTGTATAATCAGAGTGCCTCTTATACATACTTTATAGAATAAAGGAACATTTTGACAGATGAG
GTTATCCCTCGGAGTAGCGTAATCACACAATAACATTTAGAACTTAAATTGGCTACAGGACATTTTATTA
GCTTTCTATCCAGCATCTGGTCCAAACGATGGGACTCTCTGCAACTATCATTCCAGAAATGGTCTTGGAG
GTGGCCAGAATCATTGACCATATTTTTATTTGGTTCAAGAAGACATCGCTCTGGCATCCCCAGTTCAGTG
AACTGGTACAATGTGGTCCCTCTCAATATTTAAAAATATAATTTGTAACAGTGCTTACCATTCACCTCCC
AAAGCCAAGGAACTTGAGTGAGCTTCCTTGGCAAGCCTCCCATTGCCTTTCTGCATCAATGTGTTTGTTA
CCAAATAGCAGAAATTTCGCTTAGAAGGGGAAAACTTCAGCTTTCCAAAAGCTAGTAAACATGTTTCAAA
GAAAAATAAGGTCAGGAGAAAAAGAATAATGCCTACAGATTATCCTTCAGATGCTGGCTAGAATCCCCTT
TGACTTCTAGCCATAAGCTGGCCACATTGGACATCTTGGTCACTGCAGAATTTTAAAAGGCAAACTTCTC
AGAACAGATGTGCACATCTTAATTTGGTGTCTGTAAGTATCAACCATTTCTTGATCAAGATAAGGAAGCA
ACACTGATACTGAGAGAGAGGTCATATGTCCAATAGTTGCCTTTCTTCTCTTAGGCAGCTCTGCACTTCA
TGTTCAGTTTTTTAAAAATAGACTGTAGTATCCTTTCATTGGAGAATTTACAAAAATATCATCTACAGCT
CAAAAGTCCCACTAACGGCTTATATGAACAGAAAGTACTGTGTAGCCCAGGAGTTTATTTAGCAAGATAA
ATAGTGGTAATTTCTTAATCATTCCCCTTTCCTTTGCTTTACCTTCTGGCAGATGCTTAGCTACTTCTTT
GGCTACCTCATAATTTGTATGGGAATGAACATGAATTAGGGAACTTCAAATAGTGCGTAGACTTTTCCCT
ACTTACCATTGGGTTCAATTGATGTAGACTGTTAGAGCAAGTATACTCAATCAACTTGCTGATATTTTAG
TAGTTAACAGCTTCCGAGCCTATCATGCAATTTGGAGTCTGTCAATGAATATAGCAATCGGTGAAGCATT
ACTGTACGAAGTCTGTTTTCAGGCTTTGGGTCAAGCTGGTTTTATACCAAATTTCACTCTACAATGCATA
TTCTAATGAAGCTACTTTATATAAATTGGAGAGCTTATAAAATGCAGAACATTTCTATCCTATCCTACCA
AATATTTATCCTTCTCTGTCCTCTATTCTCTTACTCTCCTCTCTCCCTTTTTCCTTCCCTTTATTCCCTT
GAAAAAAGAGAATCTGTGGGAAATAGGCAGATATAGCTTTTTAAAATATAAAGTACTTGGGATTTTGCAT
TATTTTTCTTAATATCTATTTACTAAGTATGTAATTTAATATACATTAATTATTTTTTGAAACTCTTGTT
AGTGGGAAGAATATGGTAAATTTTTTGTTAAATAAAATAGACCCTTATGTTTAGCATTTTGTTTTTAGAG
AACTATTCTGGTACTATCAGAACAAATACATAAAATAACTTCCCATAGAGAACAGGATATAGCAATAATA
GCTCCTTAGATACTCAGTGGCTTCTGACTCCAATCAAGGTCTTGTTGATATTATATAGTAAAAATAAAAC
CAAAAATAAATATTATTCAAGTGGCTCTTCTAAGCATGTGAATCATGAAGCACTGAAATATGTATTTTAA
TGATGATCTTATTTATTCCCATTTTTGCCCTTAGTTAACATTTACTGGTGCTCACCTAGGATTGGCTATT
CTGAGGGATTGCATAGAAACCAAGCTCCACTTGCTGTCCTTGGGAAGGTTATAACTGAATGCAGCTCTTT
ATTTGGACTAAAGTGTCAGGATATGCATTAGATTCTCTCCTGAACCAAAAACACAACAGTCATTATCTGT
GAACCATAATTTAAAAATCTTTCTAGAATAACAACAGCAGACTCCACTCTTGTTTGTCTAAAAGAGCCCT
ACTGGGTATGGATCATTCTGATGACAGATTTATACAAAATGATTCAAACCAGTAACTTAGTAAAATTGAC
CTTCGCAAAACCTCACTGGGGGAGTGCCTTGTAGAGCTGTGGGTGGGACTGCACATTCTTTTCCTCTTAG
TAAAAGATAGGCCCACTTTATTCCAAGAATAACACTTAGCACATAAACTCTTCTTCCAGCTCGTTAGCAG
CATTAGCACCTTCTGAATTCCACCCTCTCAGAAGAATCCACAGTGTTTGAACAATTTGCATAAAGGTCAG
CTAGCATCCTGCTGCCAAGCCACTGCATAGCATTTGTGATAAGAAGGACCAACTCTAGGCTCAATATGAA
GGGATTTAGTTCTGTAAGCAGCAAAAAAGCTTCTTTATCAAGTCATCTTACCTCTAATTCTTTTCCAGTG
TGCCAACTCCAAAGTCAACATTAAAAATGTAAATGGACCTGTGTAAATATCACAGAGAGCTTTTCCTTAT
ACATCTCAATGCTGAGAGTTAAAATATTCCCAGGTTAAAATTTTTTTAAAGTACCAATAATAGAGCTAAA
TACAATGACATTTGCTTTTAAAAGGTGGATATTTTATTTCTGCTTTTTGAAAATACTTATTTAGTATTGA
CTTGGAAGCCAATTTGGTCCTTTAATAAGTAAAGAAAATAATATGTTTAAAAATGTAAATGTTTTACAAA
TTTGAAACTTTCATAATTGTATTAATCAGAAAACAAGCACATTGCCATTCTTTGAAACTCATGTTTCTAG
ACATGACAGCAGTAATAAAAGGATGAAAACAAGTGTCTTCACTAAGCGTATGGCCAATAAATGGGACCCA
AACGTTCAATCTGTTCAGTTTACCAAGGTTCAGAAATACGTAATTTAGCAGGAAACTATAAATACCAGTG
CTATCACAGCCACACATACACACACACAGACATAAAATAACCAAACATCTCATTTCTAGGAAAGAGATAA
CACTAAAGGCATCATAGGTTTAACTGAAATACGTTATATGAAGTTTTACAAAAAGGTCAACAGAAAGCTC
ATTTGTGAAAACATACTCTCATGGGAGCTTCTTTAACATTAGTTCAGAGGTTAATATATTTCCTGGAGGT
GTTTTCCTAGAATTGATTGCACTATTGCATGGTAATAACATTTAATTGTTAAGGAAACATTATATATAGG
TTCAAATTATCCCTTAATGTTGATTTCTCCCCTTTTCCATGGATTTTGATACTAAGAAACAAAATGCTTT
GAGATTTTGGTAACTATTTTGATTTTGATAAAACATGTTAAAATAGAAGGACATGATATTTTTCTATAGT
TTCCATCAGGAAGAGTACATCAGAAACTTCTCCATAAGGAAAGAAAACTGACTCTCTCTTGAACTAGTGT
TGACAAAATACACTAATGTCTTTCTTAATTTTATTTTATTAGGAGAAAAATCAGAATATTAAATTTGCAA
ACTTTTGAACAGCAGTAATAGCTCCTTAGACACTCAGTATCTTTCTTCCCTATCTTTCAAGCACATTTAA
TTTCTTTCCCCTGGTATTTGTTTGCTTTGTGTTTTTTCAATATTTTGTTTGCTAAAGAACAATAACAACA
ACAAAGCTGGTCCAGCCCTGACACCTACTTTTGCTTTTTTTCTTTCTTTCTTTTAATTTGTTGAACTATA
GGTTTCTTTCTGAGATGTATTTTTCATTCTTCCAATAATGCTCTCTTTTTCATTTCAAAACCATTACCAC
TTTCAATATGTAATTAACATCCTCATTCATCAGAAAGCTGGTAAGTCCACACAGCATTTGTGCAGTATCC
CTTCTGTGTAAAATGTTAAGGCTTTCAGATGAAAGAAAAAAGAACTTCCTGCAAAAGATCAAAAGCATCA
TACTAAATAAAATGATGACAATATGTGCCTTGATTTTTCTCTCAATGGAGGTTCAACATTGACATTTAAG
ATGTAAAAAAAATTACTTTGATGGTTTAATTAAGAATAATAAGTCAAATATCACATGTATGTAGTTCTTT
TGGATGACCGAATACCTTAAAATGAACTAAATAACTGAAATAAAGTGTTAAAATGTTAACAGCGTTGAAG
TTTTTAAAATTATAATTATCAAATGCACAATGTTCTTTTACTATGCCAAATGTTGCCTACACTGATTACA
TGATTGTCACAGTTTAAGTGAATTTCTACTATGGATCTTTATTTTCCCCAATGTTTGGATCATCCTCTCA
GATAAGAATTGTCAGTATTGCACCTCAAGAAAGAAGAAAAAATAAGAATGTAAATTTTTAAAAGCTAGCA
TTAAAAGTAAAGAGCAGTACAATAAAAATCACTTAAAATAGAGAAAGGCAACTACTGAGTAAGAAGAAAA
GATAATTTCTGTAAAAGCTAAATTTATGCACATTACAAAAAGTAGTCAAGTCAGTCTTCCATTGTGACTT
CAGGAATTCAAATGCAAGTTGTTGGCAAAATATATATATACACACACACACACATACACACACATATATG
TACATACATATATATATAGAGAGAGAGAGATAGATTTTTTTTTTTTGAGACAGAGTTTCGCTCTTGTTGC
CCAGGCTGGAGTGCAATGGAATGATCTTGGCTCACCGCAACCTCCGCCTCCAGGGTTCAAGTGATTCTCC
TGCCTCAACCTCCTGAGTAGCTGGAATTACAGGTGCCCACTACCATGCCTGGCTAGTTTTTGTATTTTTA
GTAGAGACAGGGTTTCTCCATGTTGGTCAGGCTGATCTCGAACTCCCAACCTCAGATGATCCTCCCGCCT
CCACCTCCCAAAGTACTGGAATTACAATGGCGTGAGCCACCACAAGATATTTTTAAAGCCCATTTGTCTA
TAAAAGAAAACTCTTGGACAAGGAGCTTTGTGTTTTATAATTGTATTACGTGCCTGCTTCCTTACCAAGT
GCCCTCCATAACACTAAGTAAATTTATATTTATCTAGCCTCTGAGTGACATATCTGTGTGTAATGGAATT
ATTTGCAGAATTTAATAGCCTTTTTTATTTTCGCATGAGAATTTTTCAAAAACTATCAGGGTCTAGTTTT
ATCATACATTTACTAAGTTACTACATATTGGTGGATATTGACGTTTATTCCTGGGAATCTAATTTTGCAA
ACAATTTGGAGCCATTCAGTCATAAATTCATGTCAACAATAAGTCTTGCTGCCGCTTGTGTTTCATAACT
ACGCTTGCTTTCCTTCAAAATATACCAAGTGTGTAATATAAATAAAGCCCATATCAATATAAAAAAAAAA
AAAAAAAAA
Композиционный код 6:
5,3,5,5,3,5,5,3,4,4,1,1,3,2,3,5,5,2,5,3,3,5,5,3,5,5,3,5,5,2,3,2,5,3,5,5,3,5,3,3,4,4,4,1,1,1,2,5,5,5,
2,3,5,2,1,1,1,1,1,1,1,1,1,1,1,4,4,2,5,5,2,3,3,5,3,4,4,4,3,5,5,5,3,5,5,2,5,3,5,3,3,1,1,4,1,3,2,1,4,1,
1,4,1,6,1,2,5,5,3,5,5,3,1,4,1,4,2,1,1,1,1,1,3,3,2,2,5,2,4,4,1,4,4,4,4,4,4,4,3,5,5,2,5,2,1,1,1,4,2,5,
5,3,1,1,1,1,4,1,4,1,3,3,4,4,4,3,2,5,5,3,3,5,5,5,2,4,4,4,1,1,2,4,4,4,3,5,5,3,3,4,4,4,3,5,5,3,1,4,4,1,
1,4,1,1,4,1,1,1,4,3,5,2,2,5,5,5,2,5,5,5,2,4,1,1,4,3,1,1,1,1,4,1,1,3,5,3,1,1,4,1,1,3,1,4,1,4,1,1,4,3,
4,1,1,1,4,4,1,4,1,3,5,5,5,2,5,2,2,5,2,2,5,2,4,1,4,1,4,1,1,1,1,4,1,3,5,3,5,5,5,3,4,4,4,3,1,4,4,4,1,1,
3,1,4,1,4,4,1,4,1,1,1,1,1,1,1,2,1,1,1,1,4,1,1,4,1,1,1,1,4,4,3,5,3,2,3,1,1,1,1,4,1,1,3,2,2,5,3,5,2,1,
1,4,1,3,1,4,6,1,1,3,3,5,5,3,2,5,5,5,3,5,5,2,5,5,3,5,2,2,1,1,1,1,2,1,6,4,4,1,4,3,2,2,5,3,3,5,5,5,3,3,
5,3,1,1,1,1,1,3,4,4,1,1,2,2,4,1,1,1,3,2,5,5,5,3,1,4,1,6,6,6,6,1,4,1,1,3,2,4,4,1,4,1,2,4,4,4,2,5,5,3,
3,5,3,5,3,1,4,1,1,1,1,3,3,3,2,5,5,3,1,1,1,1,1,4,1,1,3,3,1,1,4,1,3,5,5,5,3,5,5,3,4,4,1,4,1,3,1,1,1,1,
3,3,3,5,3,1,1,1,1,1,3,3,5,3,5,5,2,5,3,2,3,2,2,3,2,5,3,3,5,5,5,2,4,4,1,1,6,1,2,5,3,4,4,1,1,4,3,5,3,3,
3,5,3,5,3,5,3,5,5,5,2,5,3,2,5,3,3,5,3,1,1,1,1,1,3,5,3,5,2,3,2,5,5,2,4,4,4,3,4,4,4,3,3,5,3,5,3,5,3,3,
5,3,5,5,3,3,5,5,5,3,2,5,3,5,5,2,1,1,1,4,1,1,1,3,3,5,3,1,1,4,4,1,1,1,4,4,3,4,4,4,3,2,2,1,1,1,1,2,5,3,
4,1,1,1,1,3,5,3,5,3,5,5,2,3,3,5,3,1,1,4,4,4,2,5,2,5,3,3,2,4,1,1,4,1,3,5,5,3,2,3,5,5,3,2,3,3,3,5,5,3,
3,5,5,5,2,5,5,3,3,5,5,3,5,3,5,5,3,3,5,3,5,3,5,5,5,3,3,5,3,5,3,1,1,1,1,4,2,3,3,5,2,2,5,2,1,1,1,1,1,1,
2,2,1,1,1,4,1,3,3,5,3,5,5,3,3,3,5,2,5,2,5,5,5,2,3,3,5,5,3,5,3,5,3,3,3,4,4,4,2,5,5,3,5,3,3,5,3,3,5,3,
5,5,3,4,1,4,1,3,5,3,5,3,3,5,5,2,5,3,5,5,5,2,2,4,1,1,1,1,4,1,1,3,5,5,5,2,3,5,2,3,2,3,5,2,5,3,5,5,5,2,
5,2,5,3,3,5,5,2,5,2,5,3,1,1,1,1,3,5,5,5,2,1,4,4,1,3,3,5,3,1,1,1,1,3,3,3,3,3,5,3,5,5,3,3,3,4,4,4,3,3,
2,4,1,1,1,1,1,1,4,3,5,2,3,5,3,5,3,2,3,5,3,5,3,5,5,3,1,4,1,1,1,2,3,1,1,1,1,1,1,1,3,3,5,5,5,3,1,1,1,4,
3,5,2,1,1,1,4,3,3,2,5,3,3,3,5,3,5,3,5,3,5,3,5,2,5,3,3,5,5,5,3,5,3,2,4,4,4,3,2,5,3,1,1,1,1,1,1,2,3,5,
5,2,3,5,3,5,3,3,4,1,1,1,4,3,3,1,1,1,1,3,5,3,5,3,3,3,4,4,1,1,3,5,3,2,3,5,5,2,5,3,3,3,5,5,2,5,5,3,3,3,
5,3,5,5,5,3,2,3,5,3,3,3,5,3,3,3,5,3,5,3,3,3,5,5,5,3,5,3,3,3,1,1,6,1,3,5,5,5,3,3,2,3,5,3,5,3,5,5,5,3,
5,3,2,2,5,5,3,3,3,1,1,1,1,1,1,3,3,2,3,5,5,5,3,3,5,2,5,2,5,3,3,5,3,3,3,5,5,3,5,2,3,5,3,3,3,5,3,3,3,5,
3,3,5,3,2,3,3,2,1,4,1,1,4,3,3,3,5,5,5,2,3,5,3,2,3,3,5,5,2,2,5,3,3,3,3,3,3,2,2,2,5,3,2,3,3,3,3,1,1,1,
4,3,3,1,1,1,1,3,2,3,3,5,3,3,2,3,3,3,3,5,3,3,2,2,2,5,3,5,3,2,3,2,5,3,5,2,3,3,5,5,5,3,3,1,1,1,1,1,2,5,
3,5,5,3,5,3,3,5,5,3,3,5,5,5,2,3,2,3,3,3,3,3,5,3,5,3,2,5,3,1,1,1,1,3,2,5,3,5,5,3,3,3,5,2,5,5,3,5,3,3,
3,5,5,5,3,5,5,3,5,2,3,3,2,5,3,5,3,3,3,3,3,3,3,5,5,3,3,3,3,5,3,3,5,3,5,5,3,3,3,5,5,5,3,5,5,2,3,5,5,3,
5,5,3,1,1,1,1,3,5,5,2,5,3,5,5,3,3,3,5,3,3,1,1,1,1,1,3,5,3,3,5,3,3,2,2,3,5,5,5,3,5,3,1,4,1,1,3,2,3,5,
3,3,3,3,5,5,5,3,5,3,5,3,3,2,2,3,2,3,3,5,3,5,5,3,5,5,5,3,2,3,5,2,2,3,3,3,2,3,5,3,3,5,3,3,3,3,3,3,5,2,
2,3,5,3,3,5,3,2,3,3,3,3,5,3,3,3,5,2,5,5,5,3,3,5,5,3,3,2,5,3,5,3,3,5,3,5,3,5,5,3,5,5,3,5,5,2,3,3,3,3,
5,3,3,2,2,3,5,5,2,3,3,5,3,3,4,1,1,4,1,3,5,3,5,5,3,3,5,2,3,5,2,5,5,5,3,3,3,5,3,5,3,3,2,2,5,2,2,5,3,3,
5,5,5,3,3,3,3,5,5,3,3,1,1,1,1,3,5,5,2,3,5,5,3,5,5,3,5,3,3,4,1,1,4,3,5,3,5,5,3,5,3,2,5,3,1,1,1,1,1,2,
3,3,3,3,5,3,5,5,3,3,5,3,3,5,5,3,2,3,5,3,5,3,5,2,5,3,5,5,3,5,3,5,5,3,3,5,2,4,1,1,1,1,1,4,3,5,3,3,3,3,
4,4,4,3,3,3,3,5,5,3,5,3,5,3,3,3,3,1,1,1,4,3,5,3,1,1,1,4,3,3,3,5,3,5,2,5,5,3,5,3,3,5,5,3,3,5,5,3,5,5,
3,3,5,3,5,5,5,3,3,3,5,3,5,2,3,3,3,1,1,1,1,2,5,3,3,5,5,3,2,5,5,3,3,3,3,3,5,5,3,3,2,5,3,5,3,4,6,1,1,1,
1,1,3,3,5,5,3,5,5,3,5,2,5,3,5,5,5,3,2,3,5,3,2,5,3,1,1,1,4,1,6,1,2,5,3,2,5,3,5,5,5,3,5,5,3,5,3,3,3,3,
3,5,5,2,5,5,3,3,3,2,3,3,1,1,4,1,3,1,1,1,1,1,3,5,5,2,3,3,5,3,3,5,5,3,2,5,3,5,3,3,3,5,3,3,5,2,3,3,5,5,
3,3,2,3,3,3,3,3,2,5,5,3,5,5,5,3,3,3,5,3,5,2,5,5,5,3,3,5,3,3,5,5,5,3,5,5,5,3,3,2,3,3,3,3,3,3,3,3,5,5,
2,3,3,3,3,2,3,3,5,2,3,5,3,3,2,3,3,5,3,1,1,1,1,3,2,5,3,3,4,1,1,1,2,1,1,1,1,1,2,5,2,4,4,1,4,3,3,5,5,3,
5,5,3,3,5,3,5,5,2,5,2,1,1,4,4,3,5,5,3,5,5,2,5,5,3,3,5,5,3,3,2,2,3,5,2,3,5,3,3,5,5,5,3,5,5,3,3,3,3,3,
3,5,5,5,3,1,1,1,1,1,2,3,4,1,1,1,1,6,4,1,3,3,5,3,3,2,2,2,3,2,3,5,5,3,5,5,5,3,5,3,3,5,2,5,5,3,1,1,1,1,
3,3,5,5,3,5,3,3,3,5,5,3,5,3,5,2,5,2,3,3,5,3,1,1,1,1,1,3,5,5,3,5,3,4,1,6,1,6,4,2,3,5,2,2,5,5,2,5,3,3,
3,2,5,3,3,5,3,3,3,3,5,5,3,5,5,3,3,5,3,5,3,5,3,5,3,3,5,3,5,3,4,1,4,1,3,5,5,3,1,4,1,1,3,3,3,5,2,5,5,3,
5,3,2,3,3,5,3,2,3,3,2,2,5,3,4,1,1,1,3,3,3,3,3,2,3,2,1,1,6,1,3,3,3,3,5,3,5,3,5,3,2,3,2,5,5,5,2,3,5,3,
3,3,5,5,3,5,5,3,5,5,2,5,3,1,1,1,1,3,5,2,5,5,2,3,5,3,5,5,3,5,3,5,5,3,5,3,3,2,2,3,3,5,5,3,3,3,5,5,2,3,
2,5,5,3,5,3,5,3,5,5,5,3,3,2,5,3,1,1,1,4,1,4,3,2,1,4,6,6,1,3,3,3,5,2,5,3,3,1,1,1,1,2,5,5,3,3,5,2,3,5,
5,5,3,3,1,4,6,1,3,5,5,3,5,5,2,1,1,1,1,4,2,5,5,3,2,3,5,5,3,5,3,5,3,2,3,3,3,3,3,3,3,5,2,5,5,2,3,3,3,3,
5,3,3,3,3,3,2,3,5,3,3,3,5,3,3,5,3,3,3,2,4,4,4,3,2,3,3,2,5,3,5,3,2,5,3,2,5,3,5,3,5,3,1,1,1,1,3,1,4,1,
1,3,2,3,5,5,5,3,5,2,3,3,3,5,3,5,3,5,3,1,1,1,4,3,3,5,3,3,5,3,4,4,4,3,5,5,3,5,5,5,3,3,5,2,3,3,3,3,3,5,
2,2,1,6,1,1,4,3,5,3,5,5,2,3,3,6,1,1,6,2,3,5,3,3,3,3,5,3,3,3,2,1,4,1,6,3,5,5,3,5,3,3,5,5,3,3,3,5,5,3,
5,3,3,5,5,3,5,2,3,5,5,5,3,3,2,5,5,3,3,2,5,3,5,3,2,3,5,5,5,3,5,3,1,4,4,1,4,1,4,3,3,2,3,5,5,3,5,3,5,5,
5,3,5,3,3,4,1,1,1,1,3,3,5,2,1,1,6,1,2,5,5,5,2,5,3,5,3,4,4,4,2,5,5,2,3,3,3,5,5,5,3,5,3,3,1,4,1,1,6,1,
3,3,2,5,5,5,3,3,3,3,3,3,5,5,3,2,3,5,3,3,2,2,5,5,5,2,3,3,5,5,5,2,5,3,5,3,2,5,5,2,5,3,5,5,5,3,5,2,5,2,
5,2,3,1,6,1,1,4,1,1,3,3,5,3,5,5,3,1,1,1,1,1,3,3,5,2,3,3,2,1,1,1,1,1,1,1,4,3,3,3,3,5,5,3,3,3,3,5,5,3,
3,3,5,3,3,2,3,2,3,2,5,2,3,3,1,1,1,1,1,1,1,3,3,5,3,1,1,1,1,1,4,1,4,1,2,5,5,3,3,5,5,5,3,5,5,3,5,5,2,3,
5,3,3,5,3,3,5,5,3,2,2,3,2,5,5,2,5,2,5,5,5,3,5,5,3,2,5,3,5,3,3,1,1,4,1,3,1,1,4,4,2,2,5,5,5,3,3,5,5,5,
2,5,3,3,2,3,3,4,1,1,1,1,1,1,1,3,3,2,3,5,5,5,2,5,3,3,3,3,3,3,5,3,2,1,1,6,1,1,3,3,1,4,1,1,1,4,1,1,1,2,
5,3,5,5,3,5,3,3,5,5,2,3,5,2,5,3,2,5,2,5,5,5,2,3,3,3,2,5,5,2,3,1,1,1,1,2,5,3,3,2,5,3,5,5,2,2,1,1,1,1,
3,5,3,5,3,5,2,3,5,5,2,5,5,3,3,4,1,4,4,2,5,3,5,2,2,3,5,3,4,4,4,2,1,1,1,1,1,3,3,3,5,5,3,5,3,5,5,2,5,3,
5,5,3,3,2,5,3,3,5,3,1,1,1,1,3,6,1,4,1,3,5,5,3,5,5,5,3,3,2,5,5,2,5,5,3,5,3,3,3,3,3,2,3,3,3,2,3,3,3,3,
4,4,4,3,3,5,5,3,5,3,5,5,3,5,5,5,3,4,4,4,3,2,5,3,2,3,3,5,3,3,5,5,3,3,2,5,3,2,3,3,5,5,3,5,5,3,3,2,5,3,
3,3,3,3,3,5,5,3,3,5,2,5,2,3,5,5,3,3,2,3,5,2,3,3,5,3,2,2,5,3,5,3,3,5,3,5,3,5,3,5,3,2,3,3,2,3,3,5,5,2,
2,2,5,3,5,5,3,2,5,2,3,5,3,2,3,5,5,3,2,5,3,5,3,5,3,5,3,2,3,3,5,3,3,3,5,5,5,3,3,5,5,3,3,2,5,5,3,1,1,1,
1,1,4,1,1,3,1,1,1,1,2,5,2,5,2,5,5,3,3,3,3,5,3,2,2,2,1,1,1,1,3,1,4,1,1,1,1,1,1,1,1,3,5,3,2,3,2,3,1,1,
1,1,1,1,1,1,1,1,1,3,2,1,1,4,1,3,5,5,2,4,4,4,2,5,3,1,1,1,1,1,3,5,3,2,4,4,6,1,1,1,1,3,3,1,1,4,1,3,5,5,
3,1,1,1,1,3,1,1,6,4,1,2,5,5,3,5,3,5,5,2,3,3,1,1,4,1,2,3,5,5,3,5,3,5,5,5,2,3,3,5,3,2,5,3,3,3,3,3,3,2,
5,3,5,5,3,5,3,3,3,3,3,5,3,2,1,4,1,1,1,3,5,3,5,3,5,3,3,3,3,5,5,3,2,3,3,5,2,3,1,1,1,1,3,5,5,3,3,3,3,3,
5,5,2,5,2,5,3,5,3,3,3,3,3,3,3,3,3,5,3,2,5,5,2,3,2,3,3,3,3,5,3,5,3,5,5,3,5,5,3,2,5,5,2,3,5,3,3,3,5,5,
5,3,3,5,3,3,3,5,5,3,5,3,3,3,2,3,5,3,5,5,2,5,5,3,3,3,3,5,3,3,3,3,5,5,3,2,3,5,5,3,5,3,3,3,6,1,1,4,3,3,
5,2,3,3,4,1,1,1,1,2,5,3,2,5,3,3,3,5,5,3,3,5,2,3,3,3,3,5,3,5,3,5,5,3,5,5,5,3,3,5,3,3,5,3,5,3,3,2,5,3,
5,3,3,5,3,5,3,3,5,3,5,5,3,3,5,2,3,3,1,1,1,1,1,3,5,5,2,2,5,3,3,3,3,3,3,5,5,3,3,3,4,4,4,3,3,5,5,5,2,3,
5,2,5,3,5,2,2,3,5,3,3,5,5,3,5,5,2,3,3,5,3,3,3,5,2,3,3,3,5,3,3,3,5,3,1,1,1,1,3,5,2,3,3,3,2,5,3,2,5,3,
5,3,3,2,3,3,3,5,3,3,2,2,3,1,1,6,1,3,5,5,2,2,5,5,2,5,3,5,3,5,5,3,3,5,2,5,5,3,3,3,3,5,3,2,2,3,3,2,5,3,
5,3,5,5,5,3,3,5,3,3,3,3,2,3,5,5,3,3,5,2,3,3,3,5,3,3,3,5,5,3,5,2,5,2,3,5,5,3,3,2,6,1,1,1,2,2,3,3,3,2,
3,3,5,3,3,5,3,2,3,5,3,5,5,3,3,5,5,3,5,5,2,5,5,2,5,3,5,5,5,3,5,3,3,5,3,5,5,3,3,5,3,3,5,3,5,5,5,3,2,3,
3,3,3,3,3,2,5,3,3,5,5,5,3,3,3,2,5,3,2,3,3,3,2,3,3,5,5,3,5,3,5,3,2,5,2,5,5,2,3,5,2,3,5,2,5,5,3,3,3,5,
3,3,3,5,5,3,3,2,3,3,3,5,5,3,2,3,5,5,3,1,1,1,1,2,5,5,3,1,1,1,1,1,3,3,3,5,5,3,3,5,3,3,3,3,1,1,1,1,3,2,
3,2,3,3,5,3,5,3,2,2,5,3,3,5,3,3,5,3,3,1,1,1,1,2,3,5,2,2,5,3,3,5,5,3,5,5,3,2,5,5,5,3,3,5,3,3,3,5,5,5,
3,3,3,3,5,3,5,3,5,3,1,1,1,1,6,3,5,5,5,3,5,5,5,2,3,3,5,2,5,5,5,3,5,2,3,2,3,3,5,5,3,3,5,5,3,2,5,3,2,3,
3,2,5,3,5,5,5,3,3,3,2,3,5,5,3,3,3,3,5,5,3,5,5,2,2,5,3,5,5,3,5,3,3,3,5,3,5,5,5,3,3,2,2,3,5,3,2,5,3,5,
2,5,5,3,3,5,3,3,5,3,2,5,3,3,2,5,5,2,3,3,2,3,5,3,3,3,3,5,5,5,3,3,5,2,5,5,3,2,3,5,3,5,3,3,2,5,3,5,3,3,
5,5,3,3,3,5,3,3,5,3,3,3,3,3,3,5,2,3,5,5,2,5,3,3,3,2,5,3,3,5,3,3,2,3,3,5,3,2,3,3,5,5,2,3,3,3,2,5,3,3,
3,3,5,3,5,5,3,5,2,3,2,5,5,3,3,3,5,2,3,5,5,3,5,3,5,3,5,3,3,5,5,3,3,3,3,5,3,2,3,2,5,3,5,5,2,5,3,2,2,3,
5,3,3,5,5,3,3,3,5,5,3,5,3,2,1,1,1,1,1,2,3,3,3,3,3,3,5,3,3,3,3,3,2,3,5,3,3,5,3,5,3,3,3,2,5,3,3,2,3,5,
5,5,3,3,3,5,2,3,5,5,3,3,5,3,5,5,5,2,3,5,3,5,3,3,5,2,5,3,3,5,3,3,2,3,3,5,3,5,5,3,2,3,3,5,5,3,3,3,2,2,
5,3,5,3,5,3,5,3,1,1,1,1,3,5,2,2,5,3,5,3,3,2,5,3,3,3,3,3,3,2,2,2,2,5,5,3,3,3,3,2,3,1,1,1,1,2,5,5,3,5,
5,2,3,3,5,3,5,3,3,5,3,5,3,2,3,5,5,5,2,3,3,4,4,4,3,1,1,1,1,3,4,1,1,1,2,5,3,5,3,5,5,5,2,5,2,3,3,5,5,3,
5,3,5,5,2,5,3,3,4,4,4,3,2,2,3,2,3,3,3,5,5,3,5,3,3,5,5,3,3,5,2,3,5,2,1,1,1,1,3,3,3,5,5,2,5,3,5,3,3,3,
5,5,5,3,5,5,3,3,3,3,5,3,5,5,3,5,5,3,3,5,5,3,5,5,2,3,1,1,1,1,3,5,3,5,2,3,2,5,2,2,3,3,6,1,6,1,4,3,3,3,
5,3,5,3,5,3,2,3,5,3,3,5,3,5,3,3,5,5,3,3,3,3,5,3,5,3,3,3,3,3,5,3,5,3,5,3,4,4,4,3,3,3,3,5,3,3,3,3,3,3,
3,2,5,3,5,5,2,3,5,5,3,3,5,5,3,3,5,3,3,5,3,3,3,3,3,2,3,5,3,3,2,5,5,3,5,2,3,5,5,5,3,5,3,3,5,5,2,3,5,5,
2,1,1,1,1,1,1,3,3,5,5,3,3,4,4,4,3,5,3,5,5,2,5,3,3,2,5,3,2,3,5,3,3,2,3,2,3,3,3,2,2,5,3,3,5,5,5,3,5,5,
5,2,5,2,5,2,5,2,3,5,2,5,5,2,3,2,2,5,3,1,1,1,1,3,3,5,3,5,5,3,5,3,3,3,3,3,3,3,3,3,5,3,3,3,5,5,3,3,3,5,
5,2,3,3,3,2,3,3,5,5,3,2,3,1,4,1,4,3,5,5,3,5,5,5,3,5,5,2,2,2,3,3,3,5,2,5,3,2,3,5,5,3,3,5,3,3,3,3,5,5,
3,3,2,5,3,5,2,2,3,5,3,5,3,5,3,3,3,5,3,3,3,3,3,3,2,3,5,3,3,5,2,5,3,3,3,2,3,1,1,1,1,3,2,2,3,3,3,2,5,2,
5,3,3,5,3,1,1,1,1,3,5,3,3,2,2,5,3,5,3,3,3,3,3,5,5,3,3,5,3,3,3,3,2,5,3,5,2,2,2,2,3,3,5,2,5,3,5,3,3,5,
3,3,2,5,2,3,3,5,3,5,5,2,3,3,5,3,5,3,3,3,3,3,3,5,5,3,2,2,2,2,2,1,4,1,1,1,1,1,2,3,5,3,3,5,3,3,3,3,3,5,
5,5,3,5,3,3,3,5,5,3,3,2,5,5,3,5,5,3,3,3,3,5,3,5,3,5,3,5,3,5,5,3,2,5,2,5,5,3,5,5,2,3,2,5,5,3,3,2,2,5,
3,5,3,3,5,3,5,3,2,3,3,3,3,1,1,1,4,3,5,5,3,5,5,2,2,3,3,5,5,2,2,3,5,5,3,3,5,2,5,5,3,3,3,5,3,3,3,3,3,3,
3,5,3,3,3,3,3,5,3,3,5,2,3,5,2,3,5,5,5,3,5,5,5,3,5,3,3,5,5,5,2,3,3,2,5,3,5,3,3,5,5,3,3,3,3,3,3,2,3,5,
3,5,2,5,5,3,5,3,5,3,3,5,2,5,5,3,3,3,3,5,3,3,3,3,3,2,2,5,5,3,3,5,5,3,3,1,1,1,1,2,3,5,3,3,5,3,5,5,2,3,
5,5,3,3,5,3,3,3,3,3,3,3,5,3,3,3,3,5,5,5,3,3,3,5,3,3,5,3,5,2,3,3,5,5,3,3,5,2,3,3,2,3,3,3,3,5,5,3,2,3,
3,3,3,5,3,3,2,5,5,3,5,3,5,3,1,1,1,1,2,3,3,2,3,2,5,2,5,2,5,2,5,2,3,2,3,2,3,2,3,3,5,3,5,2,3,3,3,3,5,3,
5,3,5,3,3,3,5,5,3,2,5,3,5,3,3,3,5,2,5,3,3,5,3,2,4,1,1,1,1,1,1,1,1,1,4,3,5,5,3,5,5,5,2,2,2,5,5,3,3,3,
3,3,3,3,3,5,5,3,3,2,3,5,2,5,5,3,2,3,5,5,5,3,1,1,1,1,1,1,1,2,5,5,5,3,5,3,1,4,1,1,1,1,2,3,3,5,5,3,3,2,
5,5,3,3,3,2,3,2,3,5,3,3,2,3,2,3,3,5,5,5,3,5,5,5,3,2,5,5,5,2,3,3,3,3,3,1,1,1,4,2,3,5,3,2,5,3,3,3,2,3,
3,3,5,3,3,3,3,5,5,3,3,3,3,3,2,2,5,3,3,3,5,2,3,3,2,3,5,5,2,5,5,3,3,5,3,3,5,5,3,3,3,3,2,5,3,5,5,2,5,3,
3,3,5,5,2,2,2,3,3,3,5,3,3,3,2,5,2,5,5,3,5,5,3,2,5,3,5,3,3,2,3,3,5,3,5,3,2,3,3,3,3,3,
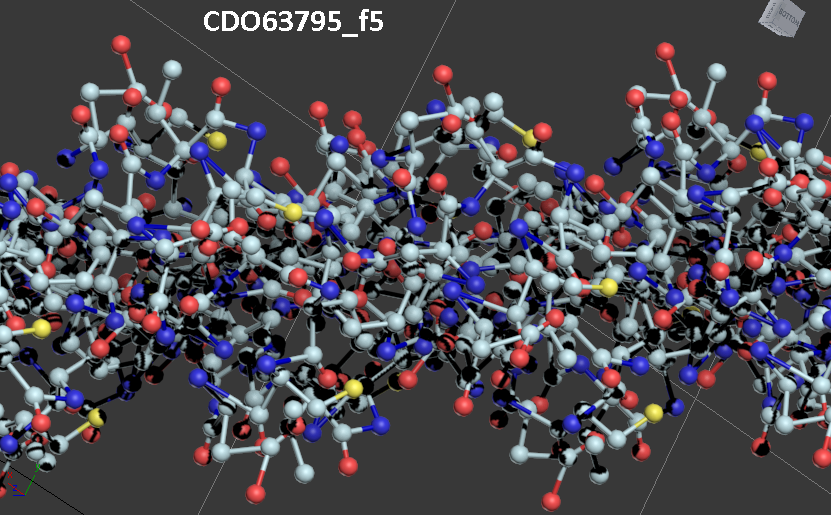
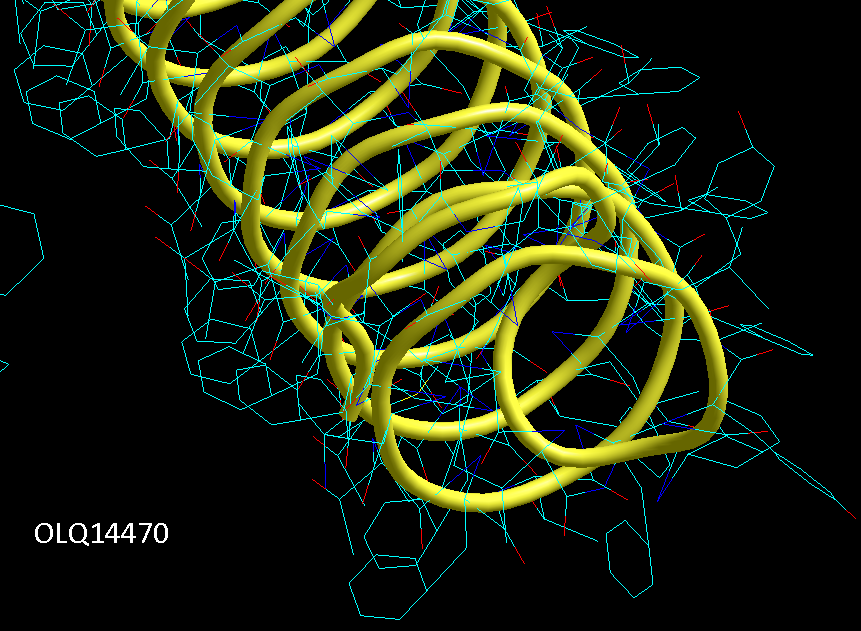
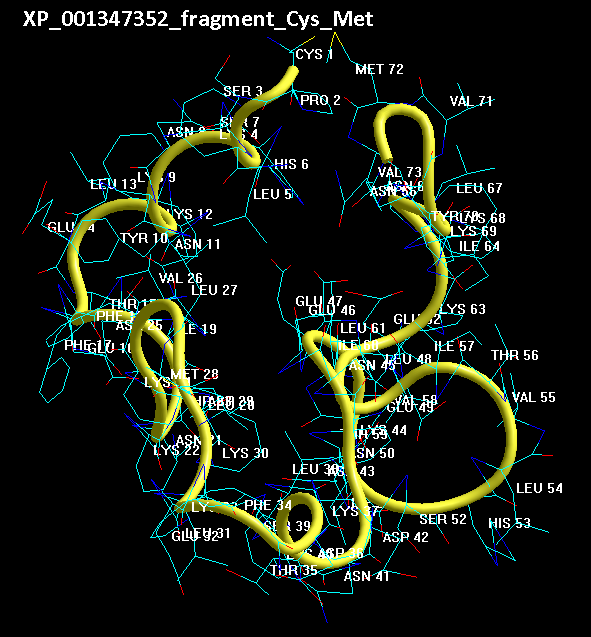
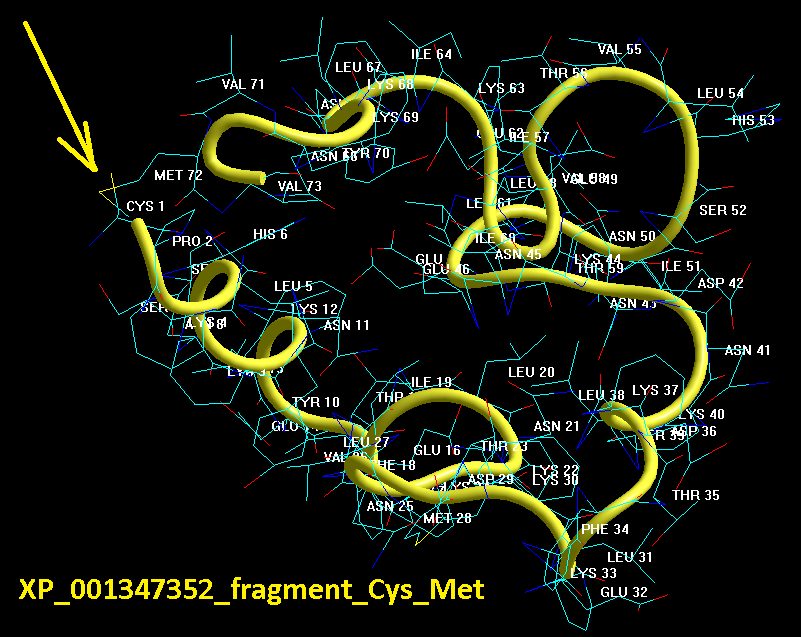
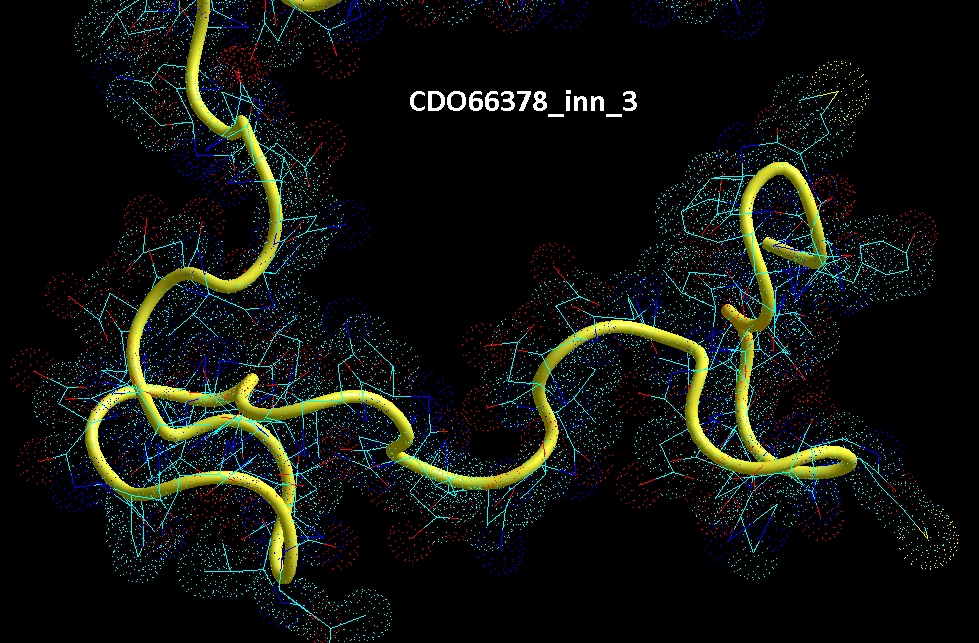
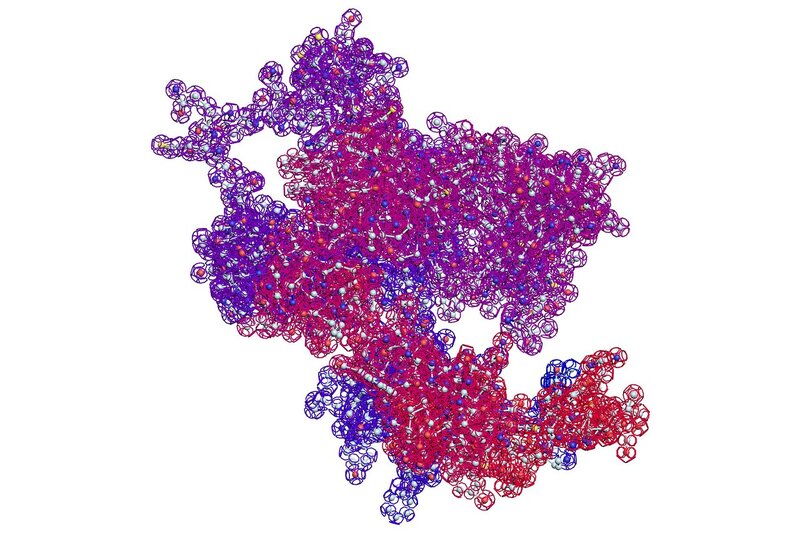
Вторичная структура (Пикотех-2017) весит около 6 мегабайт в стандарте PNG. На яндекс-фотки не грузится. Но самый интересный фрагмент удалось вырезать в программе Paint:

Сиреневым цветом отмечен виток необычной метиониновой спирали. В отличие от альф-спирали у неё более длинный шаг. Метиониновая спираль отличается от обычной альфа-спирали примерно как резьба с более крупным шагом от резьбы с менее крупным шагом:

Шаг метиониновой спирали примерно на 25% крупнее шага обычной альфа-спирали. При этом на один виток приходится тоже 3.6...3.7 аминокислотных остатка Met.

Ещё один интересный участок этого белка 3916...4164. Он содержит все типы композиционных кодов: 1,2,3,4, но ... не содержит ни одного витка спирали! Интересно оценить вероятность случайной комбинации композиционных кодов такого типа. Напомню, что спираль возникает при трёхкратном (или более) повторении кодов 1, 4 или их комбинации. При этом третий код должен быть "4", либо спиральных кодов (1,4), идущих подряд, должно быть больше 3.
ОСНОВА ПИОТЕХНОЛОГИИ - КОМПОЗИЦИОННЫЙ ГЕНЕТИЧЕСКИЙ КОД