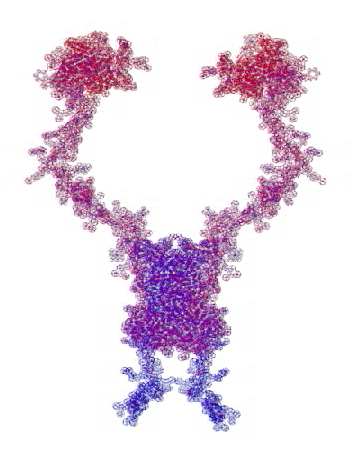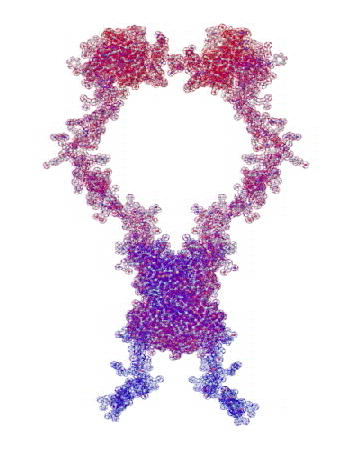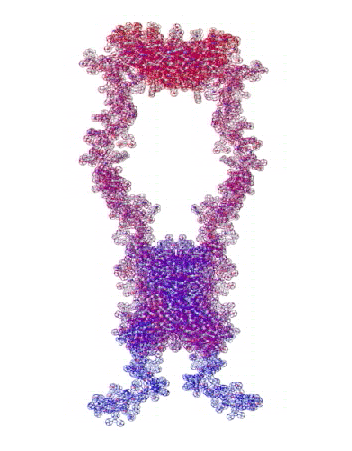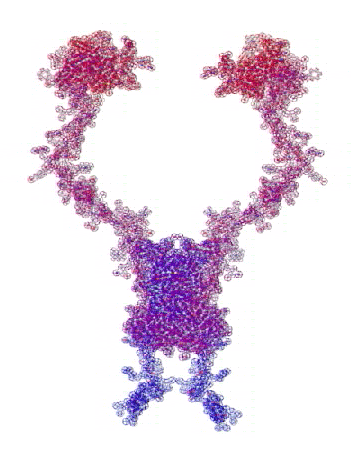Бумажный вариант книги "ПИКОТЕХНОЛОГИЯ БЕЛКОВ" стоит в 74,90 ЕВРО.
Купить книгу "ПИКОТЕХНОЛОГИЯ БЕЛКОВ" у издателя

ПИКОТЕХНОЛОГИЯ БЕЛКОВ создана на базе открытия А.Ю.Кушелевым в 1992 году композиционного генеического кода
https://nano-world-articles.nethouse.ru/articles/314915
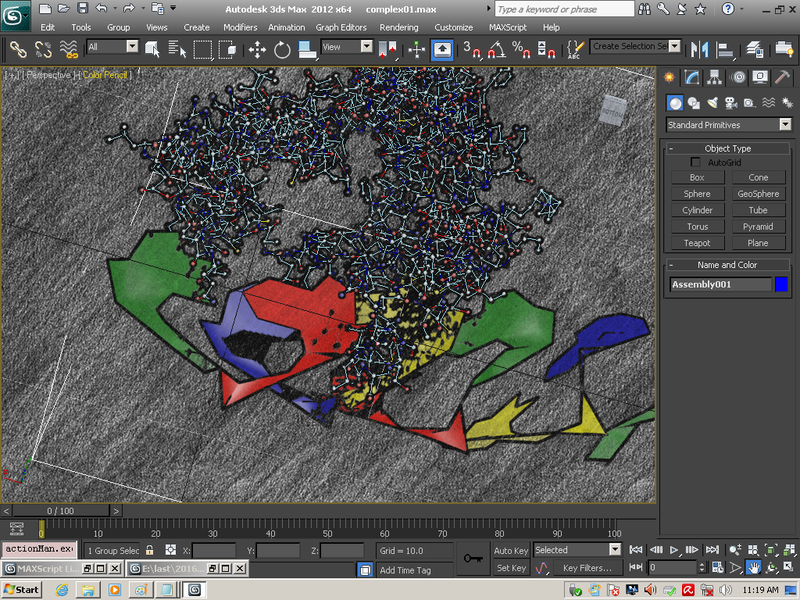
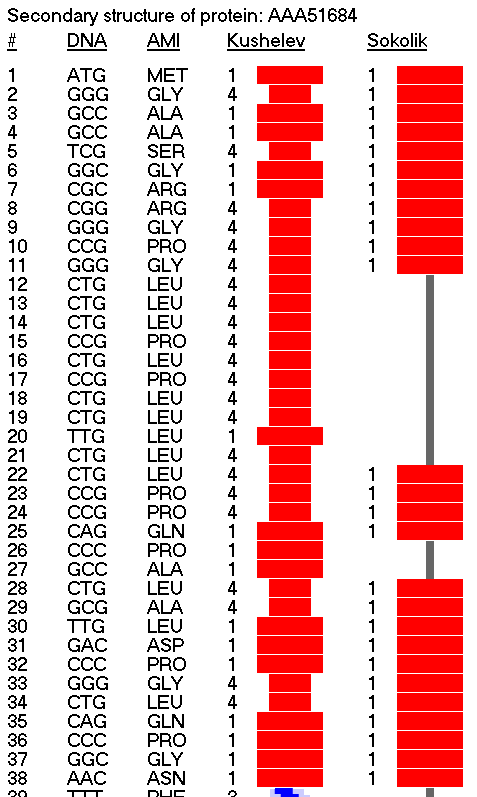
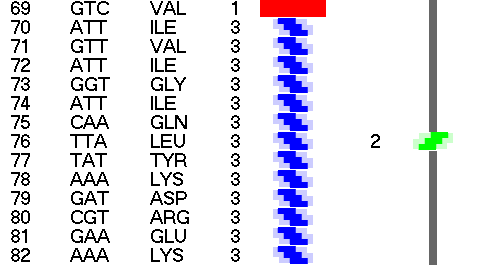
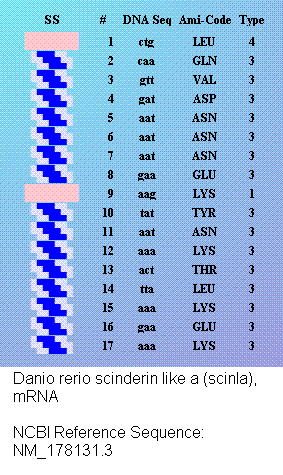
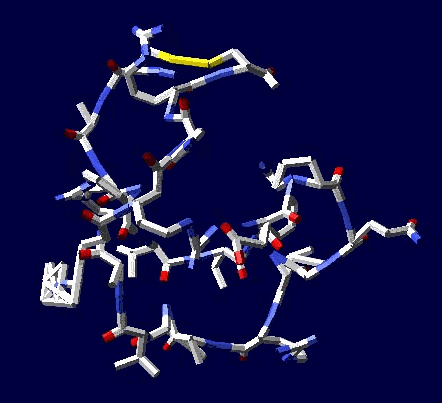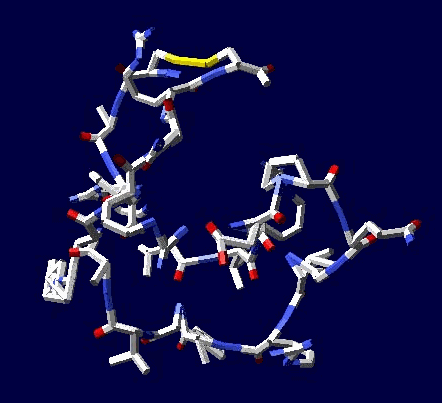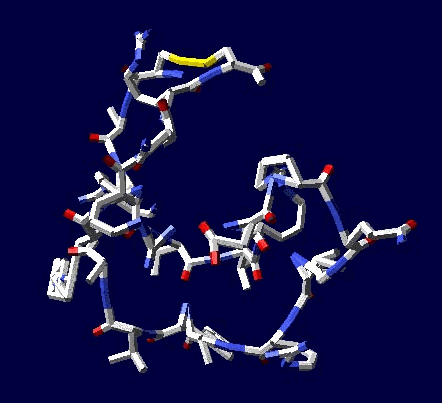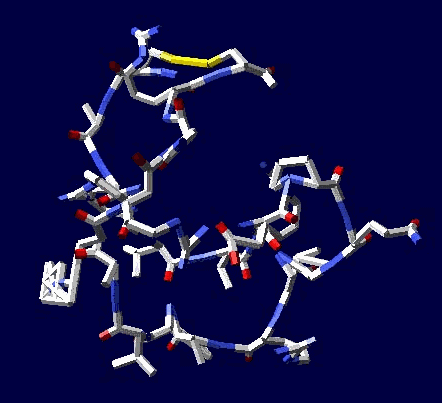
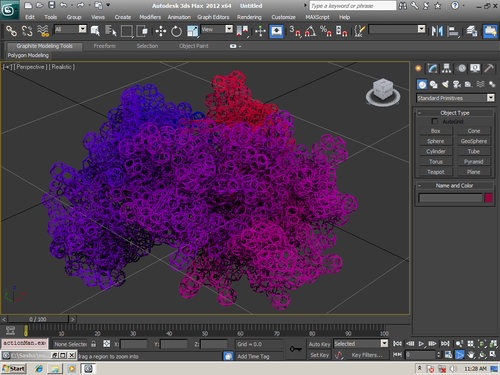
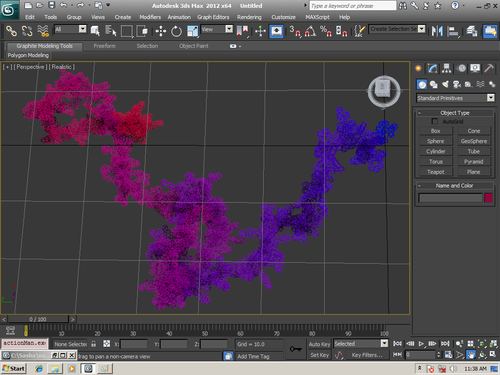
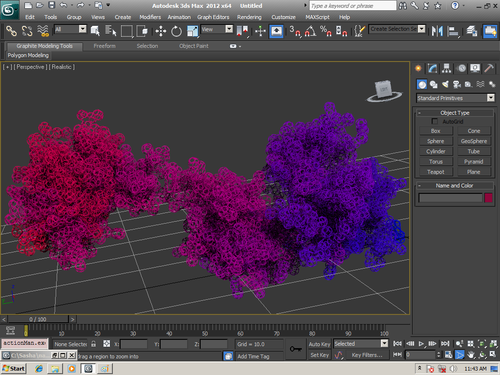
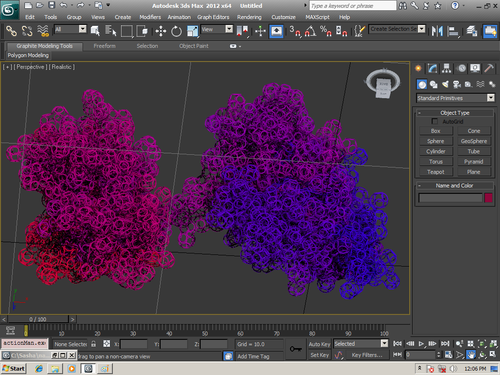
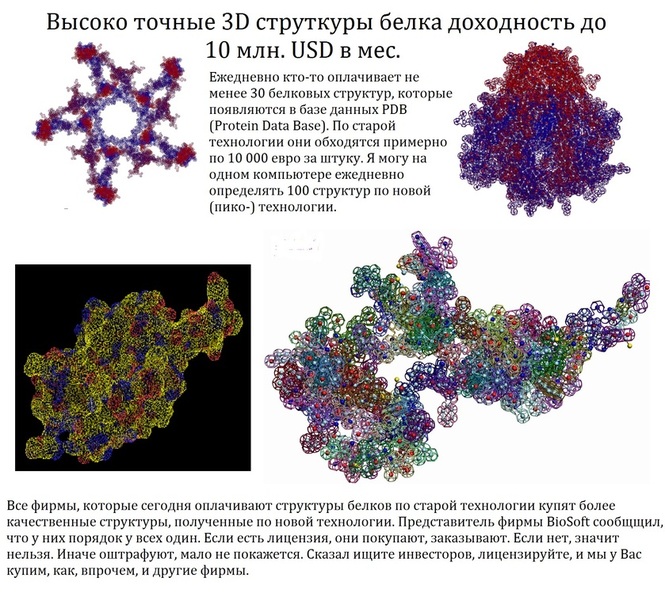
В приложении-8 показаны примеры 100 структур белков, которые я определил за 1 рабочий день на одном персональном компьютере. Точность новой технологии (пикотехнологии) в 1000 раз выше точности нанотехнологии, скорость примерно в миллион раз быстрее. Кроме того, Рентгеном можно определять структуры только закристаллизованных белков, а это всего 3%. Остальные не присталлизуются. Для новой технологии такого ограничения нет. По старой технологии за каждую структуру белка в среднем кто-то платит 10 000 евро.
Потенциальные заказчики сообщили, что они смогут заказывать структуры в лаборатории Наномир, если будет официальное разрешение от NCBI. Такой комплект документов может стоить от 100 000 USD. Зато имея это разрешение можно зарабатывать миллион евро в день, имея один персональный компьютер. Приложение-8 с примерами белковых структур можно скачать бесплатно здесь: http://nanoworld.org.ru/topic/1150/
К 2023 году производительность компьютеров выросла, в день можно стало определять 10 000 структур, интернет пережил революционные изменения.
В 2023 года для внедрения Пикотехнологии актуальны публикации в западных рецензируемых журналах с указанием ссылок на программы моделирования структур белков, а не специальное лицензирование .
Прогноз доходности на 2015 год
2016.10.14 21:46:18
В книге, которую рецензенты советуют использовать как первый учебник по пикотехнологии, в т.ч. белков, показана работоспособность классической физики в масштабах микромира. Нанотехнология против пикотехнологии - что сабля против пулемёта:http://subscribe.ru/archive/science.new … 50955.html
Экономический эффект от внедрения пикотехнологии составит 10^17 евро за десятилетие.
В расширенном электронном варианте можно заменить статические иллюстрации на динамические 3D-модели:
2016.09.22 14:04:19
Вкусная реклама книги "Пикотехнология белков"
Приложение 8.
100 структур в формате PDB, PNG ... https://yadi.sk/d/nWEkBJRdhpP4D
На 2016-09-22 скачано 63 раза.
За каждую структуру по старой технологии заказчики платят в среднем по 10 000 евро.
100 структур, которые я определил за один рабочий день специально для публикации в книге "Пикотехнология белков", обошлись бы заказчикам в миллион евро
А список этих 100 белков был составлен Викторией Соколик:

Вы уже заказали себе бумажный вариант книги "Пикотехнология белков"?
Или Вас устроит электронный вариант, который в 10 раз дешевле?
Вкусная реклама книги "Пикотехнология белков"
Рецензенты:
д.ф.-м.н. ШУЛЬГА Сергей Михайлович;
д.б.н. БОЖОК Галина Анатольевна
Соколик В.В., Кушелев А.Ю.
С 54 Геометрия живого наномира. Пикотехнология белков / В.В. Соколик, А.Ю. Кушелев. – Харьков: Издательство, 2015. – 255 с.
ISBN 978-3-659-92862-8
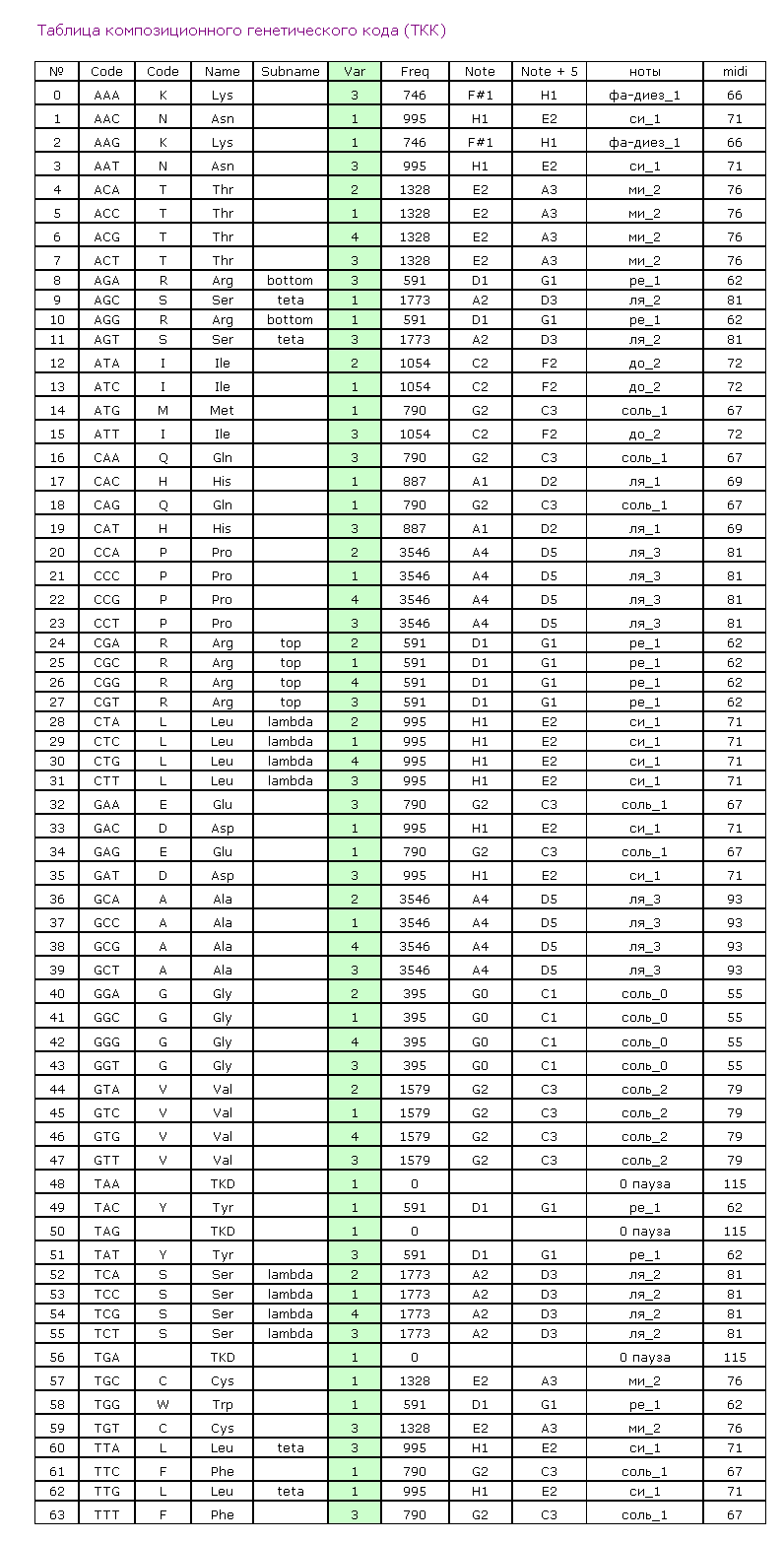
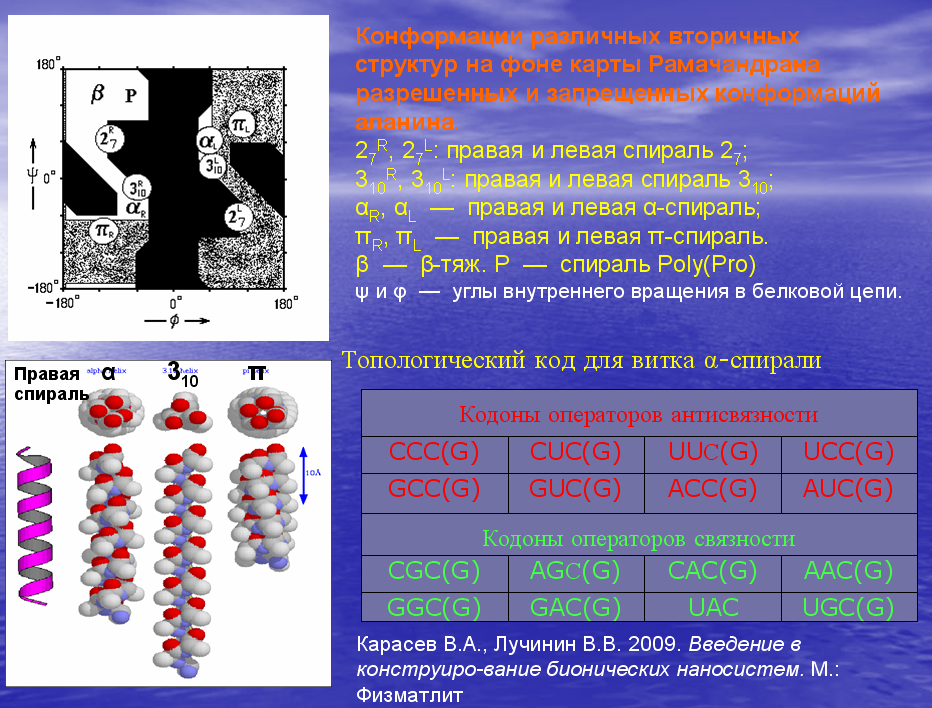
Монография посвящена 3D-структуре молекул и полимеров живых систем. Дан анализ современного понимания таких фундаментальных понятий, как физический объём атома, химическая связь, генетический код. На основе статистического анализа экспериментальных данных о структуре белка обосновано кодирование его вторичной структуры и структурного полипептидного шаблона в геноме. Предложена дополненная таблица генетического кода белков и пептидов, которая легла в основу геометрического алгоритма программ декодирования структурного шаблона белка Molecular Constructor и Picotech. Сформулирована гипотеза о перекодировании информации третьего нуклеотида кодона в соответствующий ротамер пептидной связи непосредственно 3D-структурой изоакцепторной тРНК. Математический анализ сопряженности значений углов φ и ψ (карта Рамачандрана) выявил периодичность их изменения, что позволило обосновать механизм посттрансляционного фолдинга белка.
Книга рассчитана на специалистов, занимающихся исследованиями в области молекулярной биологии, биоинформатики, биохимии, биофизики.
Табл.: 36 . Ил.:117 . Библиогр.: 227 назв.
Sokolik VV, Kushelev AY
Geometry live nanoworld. Pikotechnology proteins / VV Sokolik, AY Kushelev. - Kharkov: Publishing, 2015. – 255 p.
ISBN 978-3-659-92862-8
The monograph is devoted to the 3D-structure of molecules and polymers in living systems. The analysis of the modern understanding of the fundamental concepts of the physical volume of the atom, chemical bonding, and the genetic code is presented. Based on statistical analysis of the experimental data on the protein structure is justified coding secondary structure and structural polypeptide template of protein in the genome. Propose additions table of the genetic code of proteins and peptides, which formed the basis of the geometric algorithm software decoding structural template protein, are Molecular Constructor and Picotex. The hypothesis of recoding information to third nucleotide codon in the corresponding peptide bond rotamer directly 3D-structure isoacceptors tRNA is formulated. Mathematical analysis of contingency angles φ and ψ (Ramachandran map) revealed their frequency changes as possible to substantiate the mechanism of post-translational protein folding.
The book is intended for professionals involved in research in the field of molecular biology, bioinformatics, biochemistry and biophysics.
Table: 36. Ill: 117. Refs: 227 titles.
ПРЕДИСЛОВИЕ РЕЦЕНЗЕНТОВ
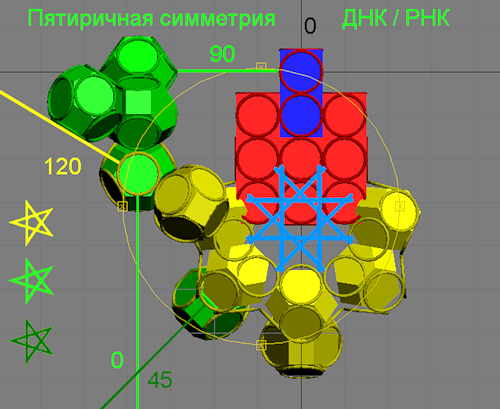
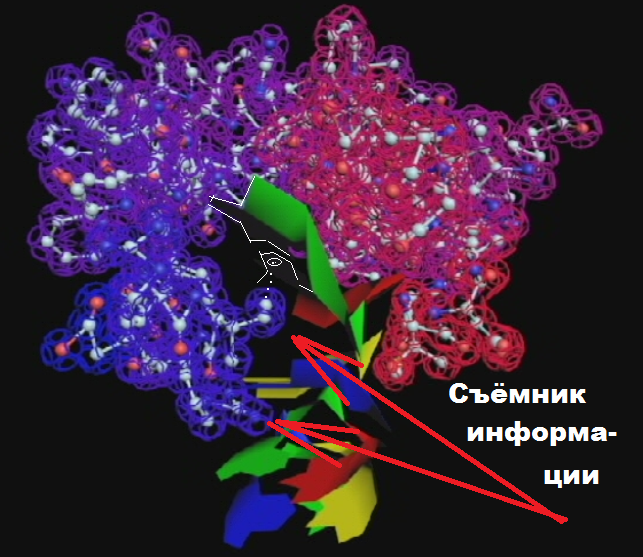
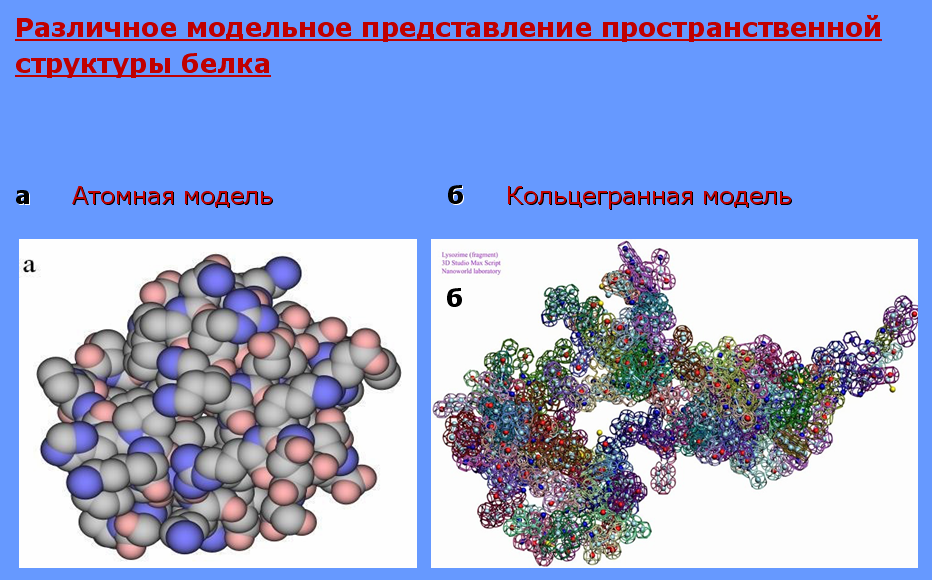
В 21-м столетии в задаче моделирования нанообъектов таких как атом углерода, органические молекулы, пары комплементарных нуклеотидов и многих других используется кольцегранная модель строения атома. В монографии В.В. Соколик и А.Ю. Кушелева «Геометрия живого наномира. Пикотехнология белков» при помощи кольцегранного подхода к строению атома наглядно (образ – модель) реализуются различные виды, свойства и особенности межатомных взаимодействий, валентные углы в молекулах, разновидности химических связей и их соотношение. Кольцегранная модель реалистично объясняет гибридизацию электронов при формировании химических связей в молекулах, что является краеугольным камнем всей биохимии белкового мира, поскольку атомы углерода в состоянии sp3-гибридизации своих электронов входят в состав скелета аминокислот и таким способом детерминируют углы между химическими связями в их молекулах, а в последующем и в структуре белка в целом.
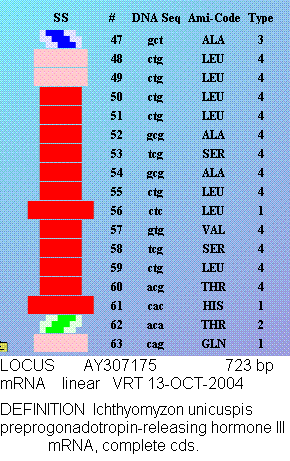
В монографии В.В. Соколик и А.Ю. Кушелева «Геометрия живого наномира. Пикотехнология белков» сформулирована идея композиционного генетического кода, кодирования ротамерии пептидной связи и структурного шаблона белка. Показано, что в геноме третьим нуклеотидом кодона детерминирован один из трёх ротамерных вариантов пептидной связи, которым аминокислотный остаток (закодированный дуплетом первых двух нуклеотидом кодона) присоединяется к растущей полипептидной цепи. Ротамерный вариант пептидной связи реализуется в процессе синтеза белка в рибосоме и поэтому с неё сходит индивидуальный структурный шаблон белка в соответствие с информацией, содержащейся в его гене. Данный механизм трансляции генетической информации является эволюционно новым. Его формирование у эукариот было обусловлено необходимостью синтеза больших и сложных белков в виде структурного шаблона, максимально приближенного к функциональной конформации этих белков, чтобы их фолдинг имел наибольшие эффективность и однозначность. У прокариот и органелл эукариот третий нуклеотид кодонов в генах небольших полипептидов ещё не является информационным, поэтому на нём и наблюдается воблирование.
Выше изложенные положения легли в основу алгоритма авторских компьютерных программ, которые по нуклеотидной последовательности мРНК позволяют смоделировать схему вторичной структуры и 3D-структуру индивидуального структурного шаблон любого белка. Эту первичную информацию о белке можно использовать в дальнейшем моделировании фолдинга функциональной конформации белка с учетом физ-химии его микроокружения, посттрансляционных модификаций, взаимодействия с лигандами методами молекулярной динамики и доккинга наравне с информацией о наиболее стабильном конформере, которую извлекают из рентгенограмм кристаллов белков. Преимущество данного подхода состоит в возможности быстрого моделирования индивидуальной пространственной структуры отдельной молекулы любого белка с точностью до электрона (пикотехнология), опираясь лишь на информацию о нём в геноме. То есть in silico воспроизводится трансляция генетической информации в индивидуальный структурный шаблон белка. Не исключено, что большинство белков именно из конформации своего структурного шаблона максимально быстро, а главное однозначно, фолдируют в нативную конформацию с минимумом свободной энергии, формиру таким образом «устойчивое большинство» конформационно лабильного белкового пула.
Авторами монографии предложен современный, точный и удобный методологический подход в арсенале молекулярной биологии для моделирования пространственной структуры белков, исходя из той информации генома о них, которой располагает сама клетка.
Итак, перед читателем книга, вводящая в мир идей и результатов, ориентированных на применение в протеомике кольцегранной модели и молекулярный полиморфизм, основанный на структурном разнообразии биомакромолекул. В этой области причудливым и невероятным образом пересекаются достижения многих областей современной науки: физики и химии, биологии и медицины, математики и информатики.
Доктор физико-математических наук С.М. Шульга
В настоящее время моделирование пептидов и белков относится к современным и востребованным методическим подходам, позволяющим не только дополнять, но и порой с успехом заменять условно гуманные эксперименты на лабораторных животных, касающиеся взаимодействия различных биогенных соединений с клеточными структурами. К сожалению, данными подходами владеют немногие естествоиспытатели, успешно работающие в своих областях наук. И в этом случае монография В.В. Соколик и А.Ю. Кушелева «Геометрия живого наномира. Пикотехнология белков» позволяет если не овладеть, то, по крайней мере, понять суть применяемых авторами методов построения моделей белков и их кодирования в геноме. Тем более, что книга очень увлекательно и доступно написана. Достигается эта легкость понимания материала тем, что в монографии четко прослеживается научная логика рассуждений и методических подходов авторов. Читателя знакомят с развитием теории строения атома, как с традиционными уже исторически устоявшимися сведениями, так и с новыми интерпретациями кольцегранной структуры атома и аппроксимации геометрии кольцегранной электронной оболочки архимедовыми телами. Авторы пользуются новой «системой координат», их пикотехнология – это технология электронного уровня, разрешение и точность которой сопоставимы с толщиной закольцованного луча-электрона (пикометр – 10-12 м) в атомах белка. Именно этот подход лежит в основе разработанного авторами геометрического алгоритма определения атомного радиуса.
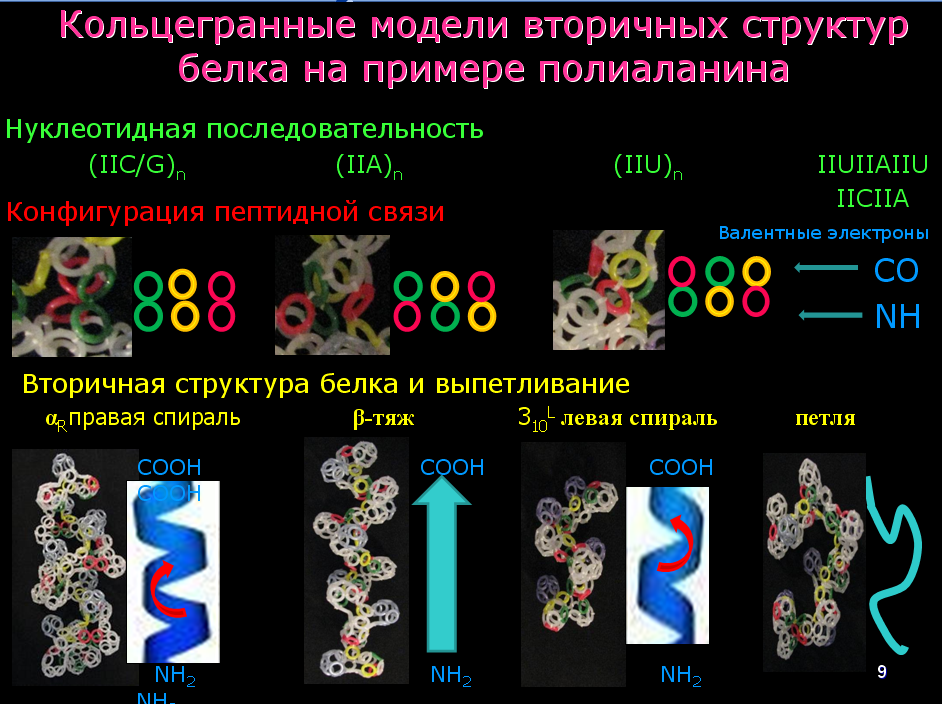
Одним из важнейших итогов материала монографии В.В. Соколик и А.Ю. Кушелева является реальная возможность с помощью законов геометрии макромира рассчитывать положения атомов в молекуле, не прибегая к квантово-механическим функциям. Такие перспективы обеспечиваются ещё и закономерностями формообразования молекул, которые определены структурой внешних кольцегранных оболочек их атомов, которая, как показано авторами, типична для элементов каждой группы таблицы Менделеева. Сопоставление в монографии экспериментальных данных с представленными моделями протеиногенных аминокислот, рассчитанными геометрическим способом для шаблона многогранных моделей аминокислот, аргументирует убедительность и логичность методологии авторов. Кроме моделей самих аминокислот авторы уточняют способ объединения их в полипептидах, возможность формирования трех видов ротамеров пептидной связи (R, 0 или L-ротамеров), которые интерпретируются авторами в качестве прототипов соответствующих конформеров вторичной структуры в белках.
Через призму теории формирования внешней электронной кольцегранной оболочки атома авторы рассматривают последовательно весь геометрический процесс построения пептидов – от пространственной структуры аминокислот до вторичной конформации и этапов фолдинга белковой молекулы. Логичным фрагментом исследований, изложенных в монографии, является объяснение способа и механизма кодирования и декодирования информации о структурном шаблоне белка в геноме.
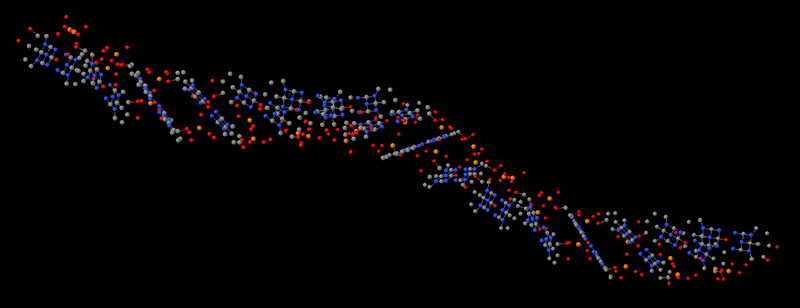
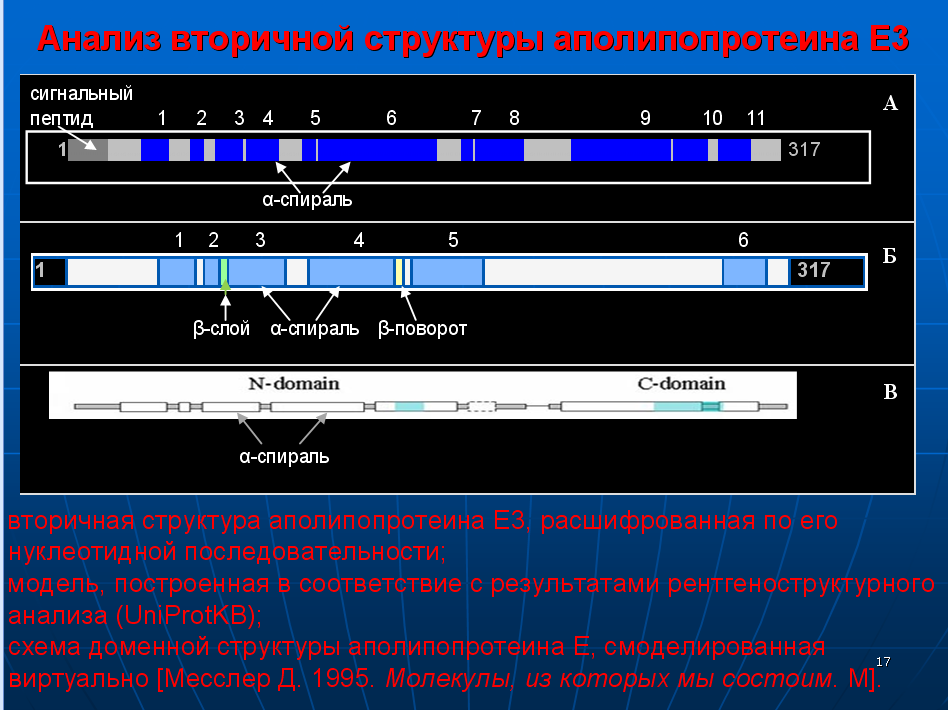
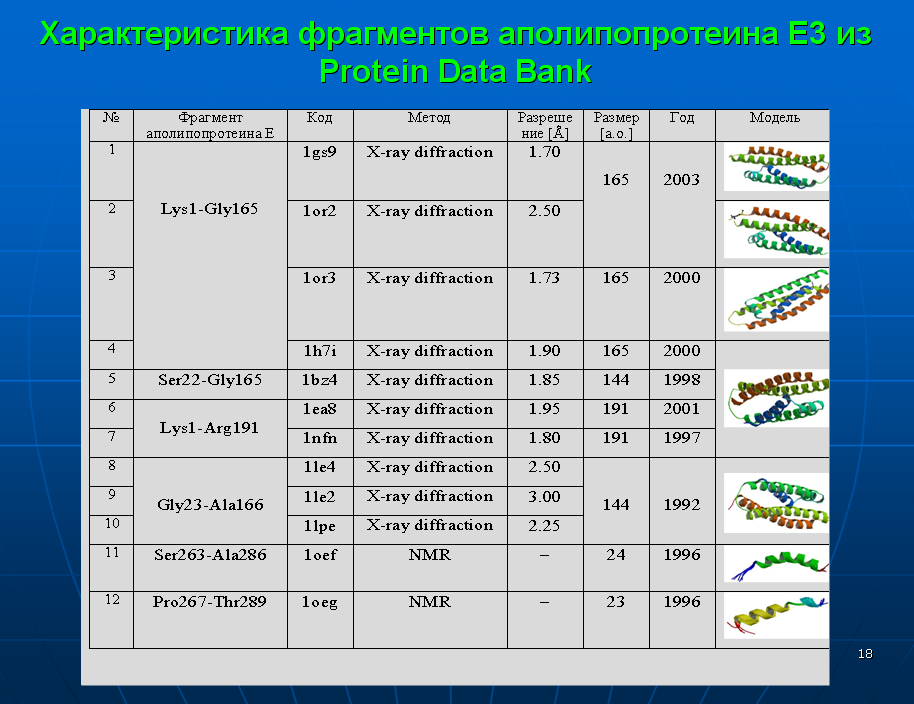
Детально охарактеризованы декодированные в программах Molecular Constructor и Picotech В.В. Соколик и А.Ю. Кушелева структурные шаблоны белков в качестве субъектов последующего фолдинга. Представлен количественный сравнительный анализ декодированных структур с соответствующими экспериментальными моделями из базы данных PDB. Особое внимание уделено способам описания пространственной структуры белка и характеристике элементов вторичной структуры, а также современным методам моделирования in silico.
Следует отметить, что данная монография актуальна не только как научный труд. Собственный интерес авторов к своей работе заражает, более чем полный массивный объем информации, увлекательная и яркая форма ее изложения и оформления делает монографию применимой также и в качестве учебника для студентов естественнонаучных специальностей, особенно таких, как биохимия, биофизика и биотехнология.
Доктор биологических наук Г.А.Божок
Вкусная реклама книги "Пикотехнология белков"
Здравствуйте.
Меня интересует вопрос, зачем при репликации удаляются праймеры? Я понимаю что это кусок рнк, был создан для начала репликации, но ведь он полностью совпадает с родительской цепочкой ДНК, зачем удалять эти нуклеотиды и вставлять такие же?
Это связано с механизмом репликации. В процессе эволюции возник механизм, который работает через удаление с последующим восстановлением.
Аналогичный механизм используется для реализации композиционного генетического кода. Это механизм трансляции: http://nanoworld88.narod.ru/data/227.htm
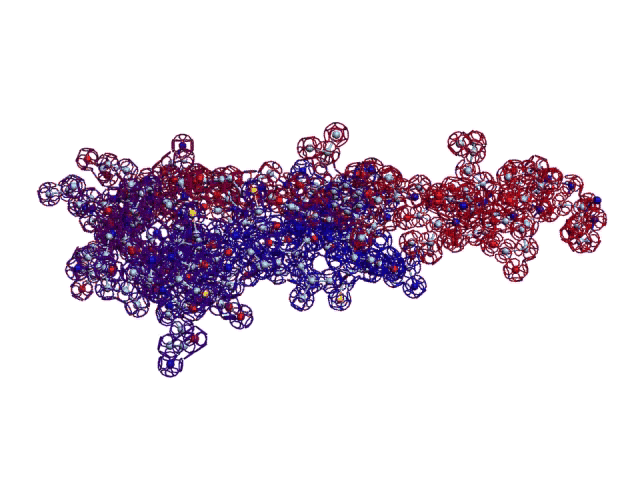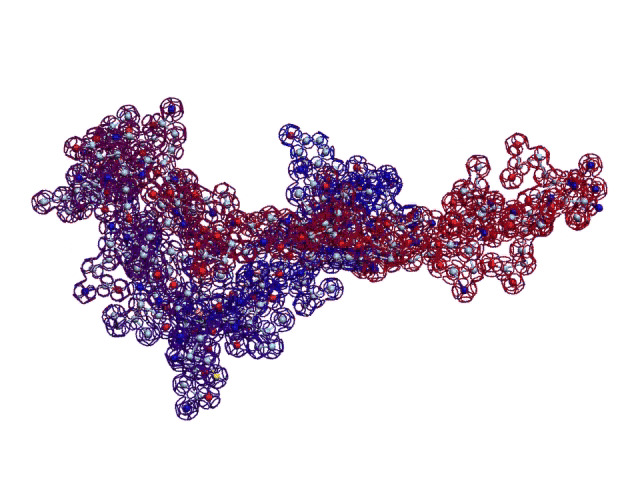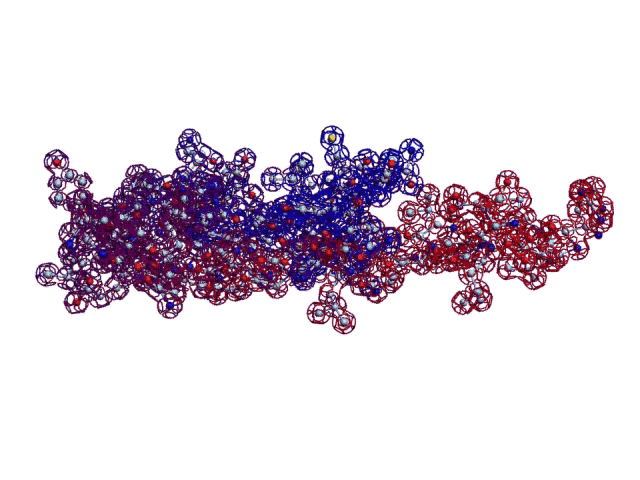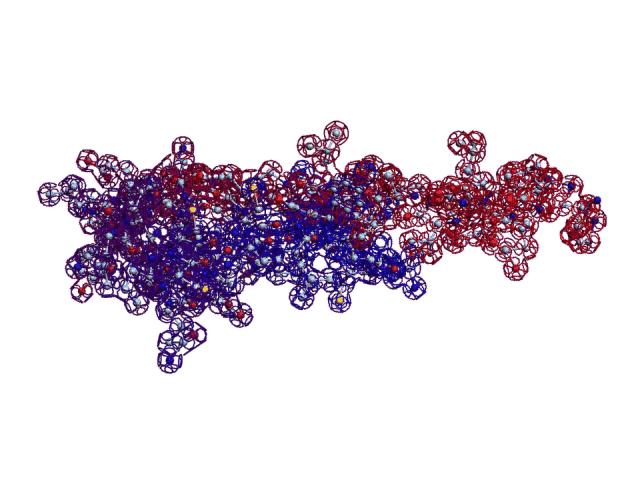
тРНК подплывает к рибосоме антикодоновым концом и ... срезает триплет иРНК, который находится на сайте трансляции. При этом тРНК продолжает вращаться:

По инерции тРНК может повернуться больше, чем на 360 градусов, но третье азотистое основание срезанного триплета тРНК останавливается раньше, встретив комплементарное основание композиционного сайта рибосомальной РНК. Так кодируется угол поворота аминокислоты, которая вращается вместе с тРНК на ССА-конце:

После установки аминокислоты под закодированным углом в растущую структуру белка триплет иРНК возвращается на место, и восстановленная структура иРНК продвигается на один триплет к выходу из рибосомы.
При репликации работает аналогичный механизм, который удаляет праймер. Этот праймер в отрезанном виде выполняет некую функцию, после чего надо вернуть его или его копию на место.
В 2015-ом году опубликована статья, в которой показан результат сканирования поверхности атомов с помощью АСМ (атомного силового микроскопа). Этот эксперимент подтвердил модельные эксперименты, которые привели к созданию пикотехнологии, в т.ч. белков:http://subscribe.ru/archive/science.new … 14532.html

Понятно, что это научное направление будет развиваться и поможет проследить работу не только механизма трансляции, но и механизма репликации во всех подробностях.
Вам может очень пригодиться книга "Пикотехнология белков":
Купить книгу "ПИКОТЕХНОЛОГИЯ БЕЛКОВ" у издателя

2016.12.05 16:50:57
ПЕРЕПИСКА С МЕНЕДЖЕРОМ
[21:01:05] Andrei: кстати ответ про отличие точности с нано не дан до конца
[21:01:21] Andrei: непонятно что измеряется в нано
[21:01:30] Andrei: молекулы?
[21:03:32] Andrei: есть ли конкретно две картинки одного и того же в нано и пико?
[21:03:40] Andrei: но чтобы рядом было
[21:03:42] Кушелев Александр Юрьевич: В нанометрах можно измерять и размеры атомов, молекул, и длины волн. А пикометр 10^-12 отличается от нанометра 10^-9 в 1000 раз
[21:04:44] Кушелев Александр Юрьевич: Здесь есть сравнение нанотехнологических моделей с пикотехнологическими: http://nanoworld88.narod.ru/data/212.htm
[21:05:21] Andrei: а есть две картинки рядом чтобы было понятно людям?
[21:09:22] Кушелев Александр Юрьевич:
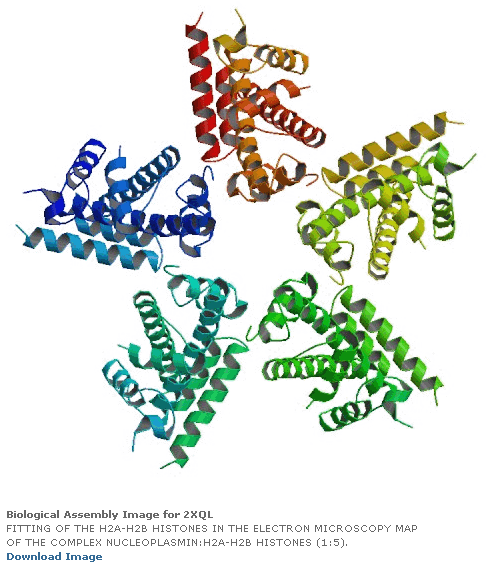

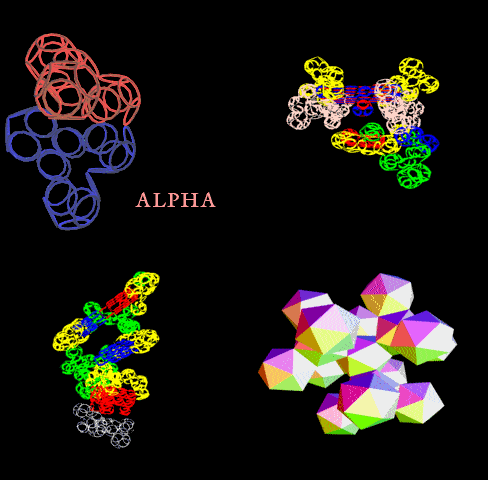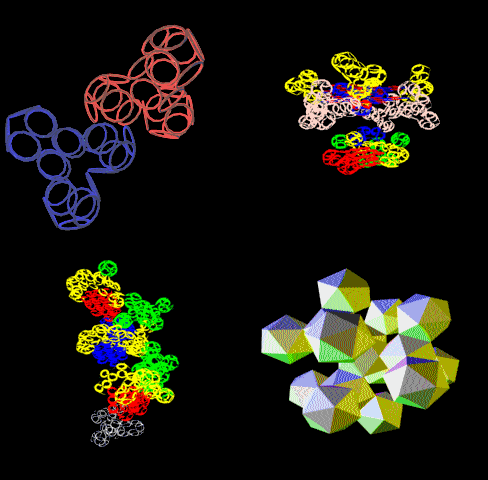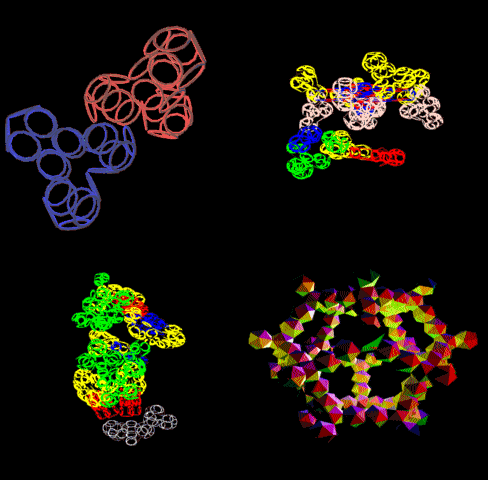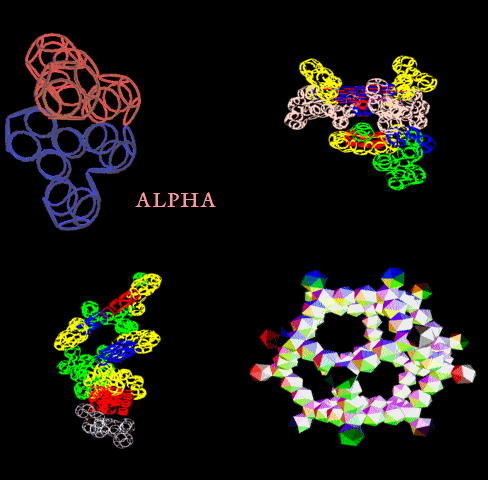
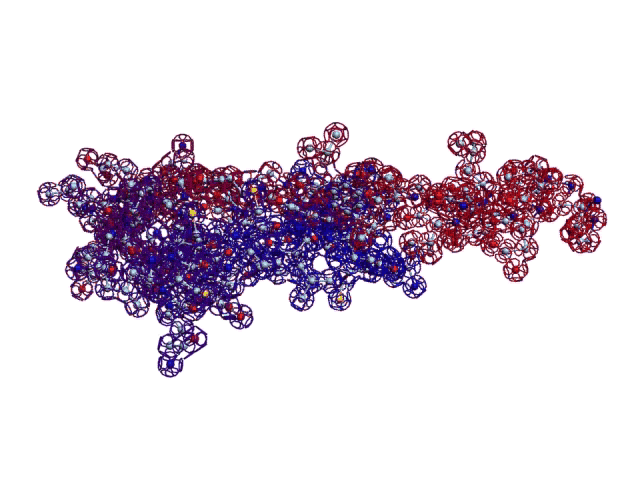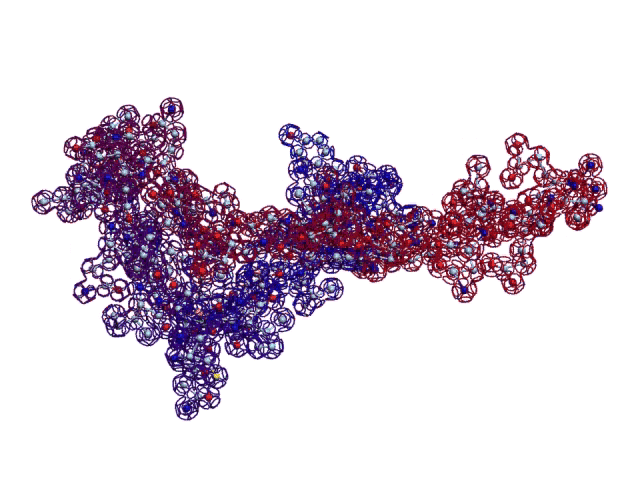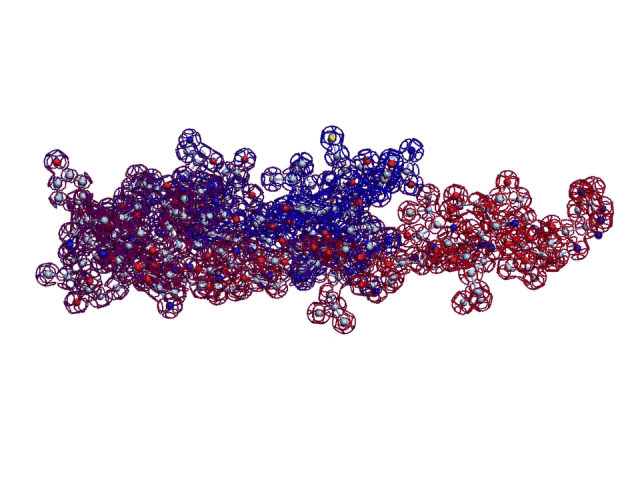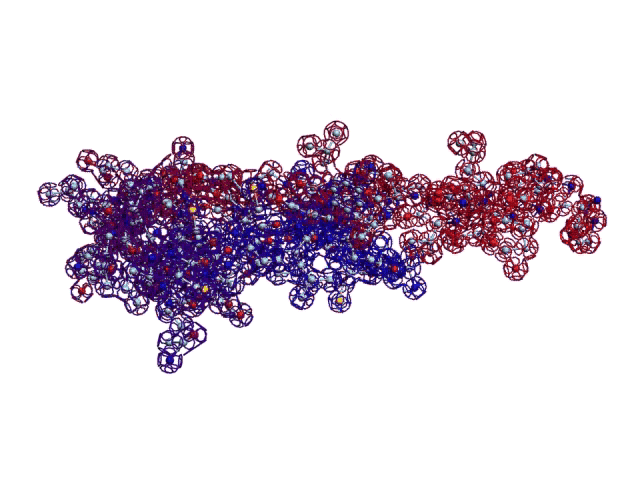
[21:09:54] Кушелев Александр Юрьевич: Верхняя картинка - нано, нижняя - пико
[21:10:44] Andrei: то есть нижняя картинка должны быть точкой на верхней, так?
[21:10:52] Andrei: и где эта точка?
[21:11:10] Andrei: почему нужно гадать?
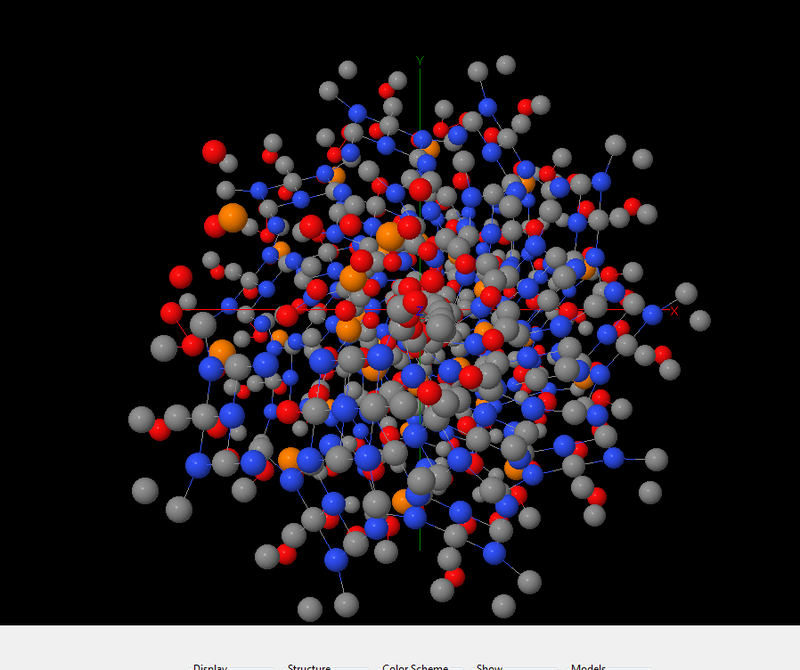
[21:13:40] Кушелев Александр Юрьевич: На нижней картинке пико-точность, там видно в 1000 раз больше деталей.
[21:14:05] Кушелев Александр Юрьевич: Каждое колечко на нижней картинке - электрон. На верхней Вы электронов вообще не видите
[21:14:18] Кушелев Александр Юрьевич: Как, впрочем и целых атомов.
[21:14:32] Andrei: а что видно на верхней? как это называется?
[21:14:32] Кушелев Александр Юрьевич: На верхней картинке Вы видите только символическое изображение альфа-спиралей
[21:14:46] Andrei: хорошо, сейчас понятно
[21:18:23] Andrei: кстат и там 2 нано картинки в чем отличие?
[21:19:34] Кушелев Александр Юрьевич: Вы отличие видите?
[21:20:07] Andrei: да, спирали отличаются
[21:20:55] Кушелев Александр Юрьевич: ОК
[21:20:56] Andrei: вверху цветные и более растянутые
[21:21:21] Andrei: какая картинка правильней?
[21:22:53] Andrei: эти картинки сделаны в разных программах или что?
[21:23:47] Andrei: надпись на английском относится к верхней картинке?
[21:23:56] Andrei: или к нижней?
[21:25:11] Andrei: просто не хочется путать людей если спросят
[21:26:40] Andrei: по логике могу лишь сделать гадание что верхняя картинка это программа сделала а внизу видимо снимок
[21:26:50] Andrei: так?
[21:28:19] Andrei: кстати про разницу между терминами нано и пико объяснено за путанно, можно проще объяснить
[21:29:00] Andrei: это и так ясно что разница в 3 порядка и без объяснений
[21:30:12] Кушелев Александр Юрьевич: Все картинки сделаны в программах.
[21:30:53] Andrei: было бы неплохо это подписать для достоверности
[21:31:41] Andrei: там написано снимок с микроскопа, это тоже программа?
[21:32:30] Кушелев Александр Юрьевич: Изображения из под микроскопов - результат компьютерной обработки неких данных
[21:33:00] Andrei: хорошо, внизу данные из микроскопа а вверху откуда?
[21:33:47] Andrei: или это те же данные но в разных программах?
[21:35:51] Кушелев Александр Юрьевич: Мои данные из программы Пикотех.
[21:36:11] Кушелев Александр Юрьевич: Нанотехнологические данные - результат компьютерной обработки данных РСА
[21:37:18] Andrei: то есть Пикотех и нанокартинки строит?
[21:38:15] Andrei: кстати на пикокартинке не видны спирали отчетливо
[21:38:34] Andrei: так, как на нанокартинке
[21:39:00] Andrei: как так?
[21:41:00] Кушелев Александр Юрьевич: Программа Пикотех выдаёт стандартный файл с координатами атомов, по которому стандартные браузеры построят всё по нанотехнологическому стандарту. Но дополнительно выдаёт и более детальную информацию, т.е. может показать все электроны. Нанотех электроны показать не может. Да и атомы показывает не там, где они есть...
[04.12.2016 21:41:38] Andrei: так, хорошо, это я запишу себе для людей
[04.12.2016 21:43:14] Andrei: уже проясняется
[04.12.2016 21:46:25] Andrei: а такой вопрос - если два белка столкнуть вместе рядом - в этом есть какой-то смысл?
[04.12.2016 21:46:43] Andrei: бывает ли такое в реальности?
[04.12.2016 21:47:15] Andrei: как белки взаимодействуют между собой и окружающим веществом?
[04.12.2016 21:48:41] Andrei: там спирали сталкиваются ли на уровне атомов происходит взаимодействие?
[04.12.2016 22:44:02] Кушелев Александр Юрьевич: На этот вопрос даже Гугл знает ответ
[8:15:39] Andrei: http://chem21.info/info/32732/
[8:15:46] Andrei: вот что нашел
[8:17:19] Andrei: кстати а может программа расчитать четвертичную структуру?
[8:17:28] Andrei: http://www.agromage.com/stat_id.php?id=720
[11:17:21] Кушелев Александр Юрьевич: Существующая версия программы Пикотех определяет только вторичную и третичную структуры белка, причём такими, какими их делает рибосома.
[11:19:10] Andrei: А на картинке написана четверичная структура
[11:20:37] Andrei: гистон 5
[11:20:48] Andrei: там где спирали
[11:20:51] Andrei: как так?
[11:21:02] Andrei: или это опечатка?
[11:22:14] Кушелев Александр Юрьевич: Четвертичную структуру я могу определять лишь вручную
[11:22:35] Andrei: а они как сделали?
[11:23:43] Andrei: в этом их программа сильней?
[11:27:02] Кушелев Александр Юрьевич: Четвертичную структуру хорошо показывает РСА. Более того, её можно иногда увидеть даже в микроскоп ![]()
[11:28:52] Кушелев Александр Юрьевич: Программа Пикотех моделирует работу рибосомы, а рибосома делает только третичную структуру. Как потом соберутся субъединицы белков четвертичную структуру, определяет физико-химическое взаимодействие. И это не очень сложная задача, т.е. как собрать домик из блоков известной формы.
[11:29:57] Кушелев Александр Юрьевич: Наиболее востребована именно третичная структура, которая собирается программой Пикотех автоматически. Именно эти результаты Пикотех я и предлагаю делать на заказ. Всё остальное не так эффективно.
Инвестирование, лицензии, менеджемент, продвижение, представительство
Работа с менеджером по нескольким научным направлениям 1 2 3 4 5 6 7