2016.12.12 17:59:23
Работа с менеджером по нескольким научным направлениям...
Andrei01 пишет:
Вот запрос от специалиста. Вы понимаете вопрос?
the requested protein code (it is publicly available data base) which is in our use. Please provide 2D & 3D predictions results files and also the Simulation time for calculating it.
The query PDB code is 1VOL.
The protein sequence can be retrieved from the PDB.
I would like to get as result from the new prediction method a pdb file based on the protein sequence.
Kushelev: We need RNA-sequence for this protein.
2016.12.12 18:04:16
Kushelev: We need RNA-sequence for this protein.
А из публичной базы данный как они говорят вы не можете это извлечь по коду?
Виктория может. Так что если им самим лень, пусть ждут Викторию ![]()
2016.12.12 18:10:30
Этот белок?
https://www.ncbi.nlm.nih.gov/gquery/?term=1VOL
Какая последовательность из 4?
https://www.ncbi.nlm.nih.gov/nuccore/?term=1VOL
2016.12.12 18:12:44
Попросите, чтобы они дали ссылку на конкретную последовательность типа такой: https://www.ncbi.nlm.nih.gov/nuccore/19 … rt=genbank
https://www.ncbi.nlm.nih.gov/nuccore/1VOL_C
2016.12.12 18:14:49
Andrei01 пишет:
А разве у одного белка есть разные последовательности? Вам виднее должно быть, вы же эксперт...
Как видите, белков с общим названием 1VOL не один. Так что просите ссылку на конкретную нуклеотидную последовательность.
2016.12.12 18:16:50
Andrei01 пишет:
Это разные белки или один с разынми модификациями? Сначала подумайте и посоветуйтесь со специалистами, не хочу им глупые вопросы задавать
Кушелев: Если нужно, я могу и обе последовательности отработать.
ggctataaaa gggctg
cagccctttt atagcc
Уточните, это то, что им нужно или это что-то ненужное?
Объясните, что нам в качестве входных данных нужен такой код. Если они не могут его предоставить, то мы не сможем определить структуру.
2016.12.12 18:20:04
Andrei01 пишет:
Я конечно могу их спросить, но если окажется что вопрос глупый и это спрашивает явно дилетант, то все впечатление будет испорчено и они поймут с кем имеют дело. Оно вам нужно?
Вы им объясните, что нам в качестве входных данных нужна конкретная нуклеотидная последовательность (RNA-Sequence). Если они не могут её предоставить, то мы не сможем определить структуру интересующего их белка.
2016.12.12 18:25:11
Andrei01 пишет:
Ладно уточню, если окажется что вопрос глупый ну пусть так ![]()
Вы объясните, что мы не научились ещё читать их мысли. Если они утверждают конкретную нуклеотидную последовательность, мы её отрабатываем. Если это не то, что им нужно, тогда зачем зря напрягаться?
2016.12.12 18:46:11
Работа с менеджером по нескольким научным направлениям...
Andrei01 пишет:
Вот тут только один такой белок
https://www.ncbi.nlm.nih.gov/Structure/pdb/1VOL
Кушелев
Мне нужно понять, это те последовательности, которые кодируют интересующую их структуру белка или это к делу не относится?
ggctataaaa gggctg
cagccctttt atagcc
Во-первых, настораживает число букв = 16. Оно не кратно 3, т.е. кодинует нецелое число аминокислотных остатков. Вполне может быть, что их интересует совсем другая структура с другим генетическим кодом.
2016.12.12 18:49:52
Andrei01
Считаете в базе данных этого белка ошибка?
Кушелев: Я не знаю. Но каждая аминокислота, точнее аминокислотный остаток кодируется триплетом, т.е. тремя буквами генетического кода. В нашем случае я вижу 16 символов
2016.12.12 19:01:59
Andrei01
А может вы что-то не знаете про такие белки? Может это специально такой белок выбрали на засыпку?
Кушелев
Зачем нам засыпки? Пусть присылают нуклеотидную последовательность интересующего их белка, а мы пришлём PDB-файл и 3D-модель (пикотехнологический паспорт).
2016.12.12 19:08:51
Andrei01 пишет:
Так там ведь число 16 вам не нравится, которого быть не может в природе... программа это проглотит?

Кушелев: Судя по 3D модели этого белка, он состоит из десятков аминокислотных остатков. А это значит, что нуклеотидная последовательность. Которая кодирует этот белок (возможно по частям) состоит из сотни букв. Если нам пришлют последовательность этих букв, то мы сможем определить структуру. Если не пришлют, то не сможем.
2016.12.12 19:17:07
Andrei01 пишет:
Так значит 2 последовательности, которые написаны в базе данных их недостаточно?
Кушелев: Я так понял, что перед нами комплекс, состоящий из двух одноцепочечных молекул РНК и двух субъединиц белка. Одноцепочечные РНК имеют длину по 16 нуклеотидов, а субъединицы белка состоят из 204 и 200 аминокислотных остатков соответственно. Мы можем определить структуры субъединиц белка в автоматическом режиме, если нам пришлют их нуклеотидные последовательности длиной 3*204 и 3*200 букв соответственно. Что касается структур РНК и комплекса, состоящего из двух РНК и двух субъединиц бекла, то это уже ручная работа, которая может дать положительный результат, может не дать. Как повезёт ![]()
2016.12.12 19:37:26
Работа с менеджером по нескольким научным направлениям...
Андрей, объясните заказчикам, что мы готовы определять вторичную и третичную структуру белков. Четвертичную и структуру комплексов мы можем пробовать определять в ручном режиме, но в качестве научных исследований, т.е. если нам это будет интересно.

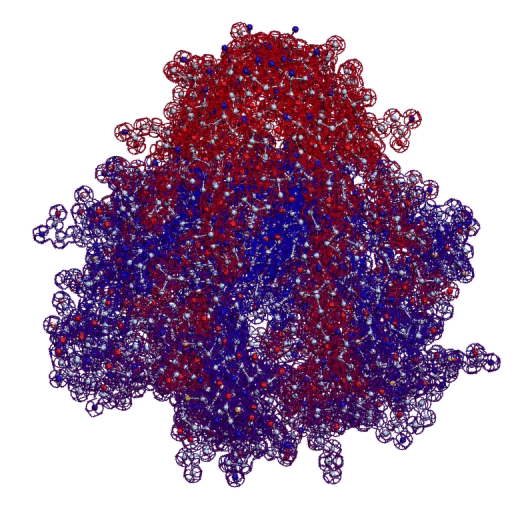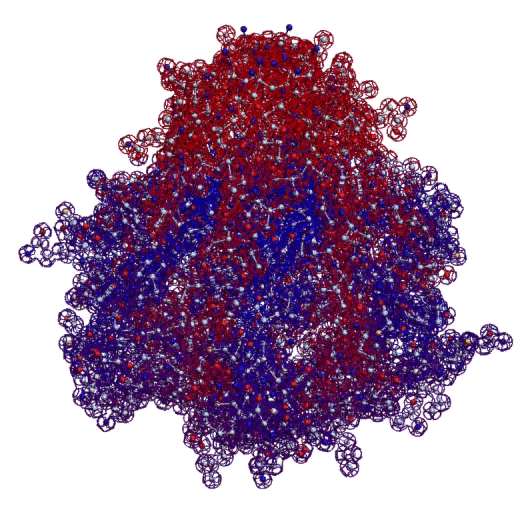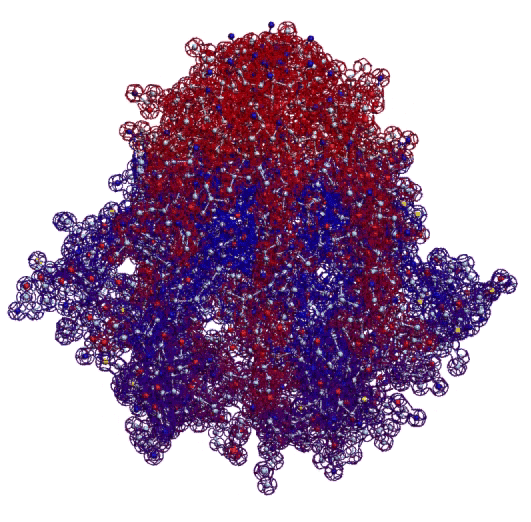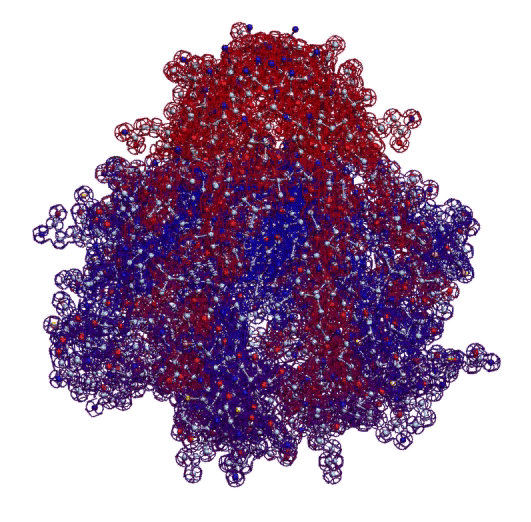
Четвертичная структура (димер) интерлейкина-34 в динамике (с конкурса CASP) и четвертичная структура инсулина (гексамер активной формы).
2016.12.12 19:48:45
Andrei01 пишет:
Так сначала 3D структуру определите... никто еще не просил 4D
Кушелев: Не проблема. Пусть шлют последовательности 3*204 букв и 3*200 букв. Определю структуру двух субъединиц комплекса 1VOL.
Andrei01 пишет:
в задании требуется использовать protein sequence
Кушелев: В последовательности аминокислот (она есть на сайте) не содержится данных о структуре белка.
В качестве входных данных нам нужна только нуклеотидная последовательность. Если она для этого белка ещё не определена, то мы не сможем определить его структуру.
2016.12.12 19:52:31
Andrei01 пишет:Kushelev пишет:
Мы можем определить структуры субъединиц белка в автоматическом режиме, если нам пришлют их нуклеотидные последовательности длиной 3*204 и 3*200 букв соответственно.
Вот ведь все послдовательности
https://files.rcsb.org/download/1VOL.pdb
Кушелев: В файле PDB есть аминокислотная последовательность и координаты атомов. А наши входные данные - нуклеотидная последовательность белковой молекулы (генетический код, RNA-sequence).
2016.12.12 20:00:52
Работа с менеджером по нескольким научным направлениям...
Andrei01 пишет:
Так их что нет в базе или как?
КУшелев: Я сам искать коды особенно не умею. Это умеет Виктория. Если найдёт, значит найдёт, если нет, значит нет. Но у нашего заказчика вполне может быть генетический код интересующего их белка. Вы открытым текстом напишите им, что входными данными для нас является RNA-sequence. Если её нет, то мы не сможем определить структуру белка.
2016.12.13 00:58:19
Работа с менеджером по нескольким научным направлениям...
Andrei01 пишет:
А если вдруг нет этой RNA-sequence, то как ее находят на практике? Секвенированием?
Кушелев: Если учесть, что весь геном человека уже прочтён, то последовательность в геноме можно найти через аминокислотную последовательность. Она даёт первые два символа из трёх для каждого аминокислотного остатка белка.
2016.12.13 01:01:12
Andrei01 пишет:
"Всё, что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB)."
Andrei01 пишет:
Вот дали код белка и сразу приехали....
Кушелев: Чтобы извлечь код из генома, нужно иметь весь геном ![]() Но Вы не переживайте. Заказчик обычно имеет кодирующую последовательность интересующего его белка. Так что спрашивайте заказчика, не стесняйтесь.
Но Вы не переживайте. Заказчик обычно имеет кодирующую последовательность интересующего его белка. Так что спрашивайте заказчика, не стесняйтесь.
2016.12.13 01:03:42
Victoriy
Покопавшись в базах мне удалось выяснить следующее:
В комплексе 1VOL
для транскрипционного фактора TFIIB -- UniProtKB - Q00403 (TF2B_HUMAN) (319 а.о.)
MASTSRLDALPRVTCPNHPDAILVEDYRAGDMICPECGLVVGDRVIDVGSEWRTFSNDKATKDPSRVGDSQNPLLSDGDLSTMIGKGTGAASFDEFGNSKYQNRRTMSSSDRAMMNAFKEITTMADRINLPRNIVDRTNNLFKQVYEQKSLKGRANDAIASACLYIACRQEGVPRTFKEICAVSRISKKEIGRCFKLILKALETSVDLITTGDFMSRFCSNLCLPKQVQMAATHIARKAVELDLVPGRSPISVAAAAIYMASQASAEKRTQKEIGDIAGVADVTIRQSYRLIYPRAPDLF
PTDFKFDTPVDKLPQL
>ENA|AAA61149|AAA61149.1 Homo sapiens (human) transcription factor
ATGGCGTCTACCAGCCGTTTGGATGCTCTTCCAAGAGTCACATGTCCAAACCATCCAGAT
GCGATTTTAGTGGAGGACTACAGAGCCGGTGATATGATCTGTCCTGAATGTGGCTTGGTT
GTAGGTGACCGGGTTATTGATGTGGGATCTGAATGGCGAACTTTCAGCAATGACAAAGCA
ACAAAAGATCCATCTCGAGTTGGAGATTCTCAGAATCCTCTTCTGAGTGATGGAGATTTG
TCTACCATGATTGGCAAGGGCACAGGAGCTGCAAGTTTTGACGAATTTGGCAATTCTAAG
TACCAGAATCGGAGAACAATGAGCAGTTCTGATCGGGCAATGATGAATGCATTCAAAGAA
ATCACTACCATGGCAGACAGAATCAATCTACCTCGAAATATAGTTGATCGAACAAATAAT
TTATTCAAGCAAGTATATGAACAGAAGAGCCTGAAGGGAAGAGCTAATGATGCTATAGCT
TCTGCTTGTCTCTATATTGCCTGTAGACAAGAAGGGGTTCCTAGGACATTTAAAGAAATA
TGTGCCGTATCACGAATTTCTAAGAAAGAAATTGGTCGGTGTTTTAAACTTATTTTGAAA
GCGCTAGAAACCAGTGTGGATTTGATTACAACTGGGGACTTCATGTCCAGGTTCTGTTCC
AACCTTTGTCTTCCTAAACAAGTACAGATGGCAGCTACACATATAGCCCGTAAAGCTGTG
GAATTGGACTTGGTTCCTGGGAGGAGCCCCATCTCTGTGGCAGCGGCAGCTATTTACATG
GCCTCACAGGCATCAGCTGAAAAGAGGACCCAAAAAGAAATTGGAGATATTGCTGGTGTT
GCTGATGTTACAATCAGACAGTCCTATAGACTGATCTATCCTCGAGCCCCAGATCTGTTT
CCTACAGACTTCAAATTTGACACCCCAGTGGACAAACTACCACAGCTA
было выбрано только последние 204 а.о.:
AMMNAFKEITTMADRINLPRNIVDRTNNLFKQVYEQKSLKGRANDAIASACLYIACRQEGVPRTFKEICAVSRISKKEIGRCFKLILKALETSVDLITTGDFMSRFCSNLCLPKQVQMAATHIARKAVELDLVPGRSPISVAAAAIYMASQASAEKRTQKEIGDIAGVADVTIRQSYRLIYPRAPDLFPTDFKFDTPVDKLPQL
нуклеотидная последовательность этого куска такая:
GCAATGATGAATGCATTCAAAGAA
ATCACTACCATGGCAGACAGAATCAATCTACCTCGAAATATAGTTGATCGAACAAATAAT
TTATTCAAGCAAGTATATGAACAGAAGAGCCTGAAGGGAAGAGCTAATGATGCTATAGCT
TCTGCTTGTCTCTATATTGCCTGTAGACAAGAAGGGGTTCCTAGGACATTTAAAGAAATA
TGTGCCGTATCACGAATTTCTAAGAAAGAAATTGGTCGGTGTTTTAAACTTATTTTGAAA
GCGCTAGAAACCAGTGTGGATTTGATTACAACTGGGGACTTCATGTCCAGGTTCTGTTCC
AACCTTTGTCTTCCTAAACAAGTACAGATGGCAGCTACACATATAGCCCGTAAAGCTGTG
GAATTGGACTTGGTTCCTGGGAGGAGCCCCATCTCTGTGGCAGCGGCAGCTATTTACATG
GCCTCACAGGCATCAGCTGAAAAGAGGACCCAAAAAGAAATTGGAGATATTGCTGGTGTT
GCTGATGTTACAATCAGACAGTCCTATAGACTGATCTATCCTCGAGCCCCAGATCTGTTT
CCTACAGACTTCAAATTTGACACCCCAGTGGACAAACTACCACAGCTA
Для TBP (TATA BINDING PROTEIN) – UniProtKB - P28147 (TBP1_ARATH) (200 а.о.) в комплексе 1VOL представлено 187 а.о. без 11 первых и 2-х последних. Кроме этого взяли последовательность TBP1, которая отличается от TBP2 качественно, но не количественно, ну и структурой конечно.
Для всего белка
>sp|P28147|TBP1_ARATH TATA-box-binding protein 1 OS=Arabidopsis thaliana GN=TBP1 PE=1 SV=1
MTDQGLEGSNPVDLSKHPSGIVPTLQNIVSTVNLDCKLDLKAIALQARNAEYNPKRFAAV
IMRIREPKTTALIFASGKMVCTGAKSEDFSKMAARKYARIVQKLGFPAKFKDFKIQNIVG
SCDVKFPIRLEGLAYSHAAFSSYEPELFPGLIYRMKVPKIVLLIFVSGKIVITGAKMRDE
TYKAFENIYPVLSEFRKIQQ
>ENA|AAR28027|AAR28027.1 Arabidopsis thaliana (thale cress) partial TBP1
ATGACTGATCAAGGATTGGAAGGGAGTAATCCAGTTGATCTTAGCAAGCATCCTTCAGGG
ATTGTTCCTACTCTTCAAAACATTGTCTCCACGGTGAACTTAGACTGCAAGCTAGATCTT
AAAGCCATAGCTTTGCAGGCTCGGAATGCTGAATATAATCCCAAGCGTTTTGCTGCGGTG
ATAATGAGGATCAGAGAACCGAAGACTACAGCATTAATATTCGCCTCAGGGAAAATGGTC
TGTACTGGAGCTAAGAGCGAGGACTTTTCGAAGATGGCTGCTAGAAAGTATGCTAGGATT
GTGCAGAAATTGGGATTCCCTGCAAAATTCAAGGATTTCAAGATTCAGAATATTGTAGGT
TCTTGTGATGTCAAATTCCCTATAAGACTTGAAGGTCTTGCTTACTCTCACGCTGCTTTC
TCAAGTTATGAGCCCGAGCTCTTCCCAGGGCTGATTTATAGGATGAAAGTCCCAAAAATC
GTCCTTCTAATCTTTGTCTCTGGGAAGATCGTAATAACAGGAGCCAAGATGAGAGATGAG
ACCTACAAAGCCTTTGAGAATATATACCCCGTGCTCTCGGAATTCAGAAAGATACAGCAA
Для его части (187 а.о.)
VDLSKHPSGIVPTLQNIVSTVNLDCKLDLKAIALQARNAEYNPKRFAAV
IMRIREPKTTALIFASGKMVCTGAKSEDFSKMAARKYARIVQKLGFPAKFKDFKIQNIVG
SCDVKFPIRLEGLAYSHAAFSSYEPELFPGLIYRMKVPKIVLLIFVSGKIVITGAKMRDE
TYKAFENIYPVLSEFRKI
нуклеотидная последовательность
GTTGATCTTAGCAAGCATCCTTCAGGG
ATTGTTCCTACTCTTCAAAACATTGTCTCCACGGTGAACTTAGACTGCAAGCTAGATCTT
AAAGCCATAGCTTTGCAGGCTCGGAATGCTGAATATAATCCCAAGCGTTTTGCTGCGGTG
ATAATGAGGATCAGAGAACCGAAGACTACAGCATTAATATTCGCCTCAGGGAAAATGGTC
TGTACTGGAGCTAAGAGCGAGGACTTTTCGAAGATGGCTGCTAGAAAGTATGCTAGGATT
GTGCAGAAATTGGGATTCCCTGCAAAATTCAAGGATTTCAAGATTCAGAATATTGTAGGT
TCTTGTGATGTCAAATTCCCTATAAGACTTGAAGGTCTTGCTTACTCTCACGCTGCTTTC
TCAAGTTATGAGCCCGAGCTCTTCCCAGGGCTGATTTATAGGATGAAAGTCCCAAAAATC
GTCCTTCTAATCTTTGTCTCTGGGAAGATCGTAATAACAGGAGCCAAGATGAGAGATGAG
ACCTACAAAGCCTTTGAGAATATATACCCCGTGCTCTCGGAATTCAGAAAGATA
Надо моделировать полные версии обоих белков отдельно и посмотреть на их комплекс. Влепить в комплекс ДНК я затруднюсь. Куски белков TFIIB (204 а.о) и TBP (187 а.о.) необходимы лишь для автоматического режима сравнения структур, но их имеет смысл предоставить заказчику наряду с полноразмерными протеиновыми моделями.
Моделированием займемся завтра.
2016.12.13 02:52:10
Victoriy
Не удержалась и смоделировала оба белка из 1VOL комплекса: [
1VOL.jpg
Андрей, спросите у заказчика, работают ли они с молекулярной динамикой и доккингом больших белков. И ещё предупредите, что в моделях структурных шаблонов есть клэш (перекрывание атомов далекоотстоящих друг от друга) и однотипные значения углов. Если её это не смутит и не напугает с непривычки (я уже наблюдала такую реакцию), то я подскажу как с этим справиться. Даже если модели будут разлетаться при оптимизации методами МД, есть способ пошаговой оптимизации с учетом географии медленных кодонов.
2016.12.13 10:31:58
Andrei01 пишет:Kushelev пишет:
Victoriy пишет:
Надо моделировать полные версии обоих белков отдельно и посмотреть на их комплекс. Влепить в комплекс ДНК я затруднюсь. Куски белков TFIIB (204 а.о) и TBP (187 а.о.) необходимы лишь для автоматического режима сравнения структур, но их имеет смысл предоставить заказчику наряду с полноразмерными протеиновыми моделями.
Кушелев: Благодарю!
Можете ли растолковать на человеческом языке, что значит "Влепить в комплекс ДНК я затруднюсь"?
То есть, даже если имеем все необходимые данные по RNA, то все равно неизвестно как слепить 3D структуру этого белка?
Кушелев: С третичной структурой белка проблем нет. Виктория говорит о ручной работе, связанной с определением структуры РНК/ДНК. Ведь это не белок, и мы не можем определить её трёхмерную структуру автоматически. Да и вручную это не всегда возможно. Естественно, что задача определения четвертичной структуры комплекса, куда входят две субъединицы белка и две РНК или ДНК - это более сложная задача, чем определение вторичной и третичной структуры белка. Мы берёмся только за вторичную и третичную структуру белка. Более сложные задачи мы можем пытаться решать, если это нам интересно в научном плане. Их сложность значительно выше, требует "ручной работы", так что это - отдельный разговор...
2016.12.13 10:38:17
Уважаемая Виктория!
Вы смоделировали структуры белков с помощью алгоритма, который учитывает коды 310-спирали или не учитывает?
2016.12.13 12:16:48
Andrei01 пишет:
Виктория, а время вычисления и файл pdb можно получить, согласно заданию?
Кушелев: Время тратится главным образом на подготовку данных, т.е. в данном случае нужно было найти нуклеотидные последовательности, переформатировать их так, чтобы существующие версии программ их поняли. Дело в том, что форматов данных существует много, стандарты меняются, поэтому приходится согласовывать новое представление данных с нашими программами. Далее нужно несколько минут, чтобы программа сформировала по аглоритму PDB-файл с координатами атомов (современный стандарт). В этот файл нужно вручную занести комментарии. Кроме того мы делаем пикотехнологический паспорт молекулы, т.е. пространственную структуру с точностью до пикометра, где виден каждый электрон. Далее эта пикотехнологическая модель поворачивается и создаётся анимация, чтобы исследователи могли полнее представить себе молекулу в пространстве. Мы можем определить активные центры белковой молекулы по характерным звукам, сопутствующим сборке белковой молекулы. Иногда можно заметить и другие нюансы, исследуя 3D-модель пикотехнологической точности. Обычно достаточно одного рабочего дня, чтобы в первом приближении исследовать структуру белковой молекулы. А получить PDB-файлы 100 белков можно тоже за один рабочий день.
2016.12.13 12:46:50
Работа с менеджером по нескольким научным направлениям...
Кушелев: Вот так выглядит входной файл одного из двух белков комплекса 1VOL для существующих версий программ 2D и 3D Пикотех:
FT CDS 1..957
ATGGCGTCTA CCAGCCGTTT GGATGCTCTT CCAAGAGTCA CATGTCCAAA CCATCCAGAT
GCGATTTTAG TGGAGGACTA CAGAGCCGGT GATATGATCT GTCCTGAATG TGGCTTGGTT
GTAGGTGACC GGGTTATTGA TGTGGGATCT GAATGGCGAA CTTTCAGCAA TGACAAAGCA
ACAAAAGATC CATCTCGAGT TGGAGATTCT CAGAATCCTC TTCTGAGTGA TGGAGATTTG
TCTACCATGA TTGGCAAGGG CACAGGAGCT GCAAGTTTTG ACGAATTTGG CAATTCTAAG
TACCAGAATC GGAGAACAAT GAGCAGTTCT GATCGGGCAA TGATGAATGC ATTCAAAGAA
ATCACTACCA TGGCAGACAG AATCAATCTA CCTCGAAATA TAGTTGATCG AACAAATAAT
TTATTCAAGC AAGTATATGA ACAGAAGAGC CTGAAGGGAA GAGCTAATGA TGCTATAGCT
TCTGCTTGTC TCTATATTGC CTGTAGACAA GAAGGGGTTC CTAGGACATT TAAAGAAATA
TGTGCCGTAT CACGAATTTC TAAGAAAGAA ATTGGTCGGT GTTTTAAACT TATTTTGAAA
GCGCTAGAAA CCAGTGTGGA TTTGATTACA ACTGGGGACT TCATGTCCAG GTTCTGTTCC
AACCTTTGTC TTCCTAAACA AGTACAGATG GCAGCTACAC ATATAGCCCG TAAAGCTGTG
GAATTGGACT TGGTTCCTGG GAGGAGCCCC ATCTCTGTGG CAGCGGCAGC TATTTACATG
GCCTCACAGG CATCAGCTGA AAAGAGGACC CAAAAAGAAA TTGGAGATAT TGCTGGTGTT
GCTGATGTTA CAATCAGACA GTCCTATAGA CTGATCTATC CTCGAGCCCC AGATCTGTTT
CCTACAGACT TCAAATTTGA CACCCCAGTG GACAAACTAC CACAGCTA
//
Так выглядит вторичная структура этого белка по версии Кушелева (левая колонка цветных полос) и Соколик (правая колонка цветных полос):

Три ортогональные проекции 3D-модели белка:

Оригинал (3000*2000): https://img-fotki.yandex.ru/get/42618/1 … d_orig.jpg
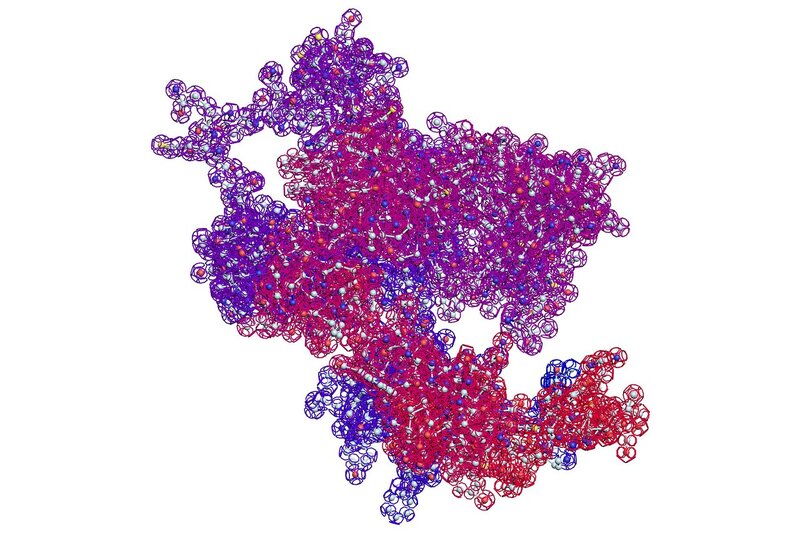
Оригинал (3000*2000): https://img-fotki.yandex.ru/get/170815/ … e_orig.jpg
Оригинал (3000*2000): https://img-fotki.yandex.ru/get/41340/1 … 0_orig.jpg
Анимация: https://cloud.mail.ru/public/KJ8y/1yG19gQTW
PDB-файл с координатами атомов: https://cloud.mail.ru/public/CFfm/qjhzdX63v
2016.12.13 13:29:30
Andrei01: Почему у вас разные спирали? Спираль либо есть либо нет либо фантазия?
2016.12.13 13:41:27
Andrei01 пишет:
Почему у вас разные спирали? Спираль либо есть либо нет либо фантазия?
Кушелев: Вы имеете в виду, почему по моему алгоритму программа определения вторичной структуры показывает, например, 310-спираль (красную узкую) или пи-спираль синего цвета, а по алгоритму Виктории Соколик не показывает?

На это Виктория Соколик сказала: "Мы получим одинаковый результат, но разными способами"
Третичная структура, полученная по алгоритму Виктории Соколик отличается от третичной структуры, полученной по алгоритму Александра Кушелева. Так что заказчик имеет возможность сравнить и решить, воспользоваться ему одним из вариантов, который он посчитает более правильным или использовать оба варианта.
"Наука не стоит на месте", поэтому алгоритмы будут эволюционировать и в будущем. В настоящее время они упрощённые, но Вы видите, что даже разные алгоритмы (Кушелева и Соколик) дают похожие результаты. Какой из них более правильный - покажет время. На международном конкурсе CASP результаты работы алгоритмов Кушелева и Соколик вписались в общую схему, т.е. пока не удалось выяснить, какой из алгоритмов лучше соответствует данным РСА, а данные РСА в свою очередь могут хуже соответствовать реальности, чем данные, скажем, программы Пикотех
2016.12.13 13:44:10
Какой критерий правильности алгоритма?
2016.12.13 13:49:40
Код второго белка (TBP1) комплекса 1VOL:
FT CDS 1..600
ATGACTGATC AAGGATTGGA AGGGAGTAAT CCAGTTGATC TTAGCAAGCA TCCTTCAGGG
ATTGTTCCTA CTCTTCAAAA CATTGTCTCC ACGGTGAACT TAGACTGCAA GCTAGATCTT
AAAGCCATAG CTTTGCAGGC TCGGAATGCT GAATATAATC CCAAGCGTTT TGCTGCGGTG
ATAATGAGGA TCAGAGAACC GAAGACTACA GCATTAATAT TCGCCTCAGG GAAAATGGTC
TGTACTGGAG CTAAGAGCGA GGACTTTTCG AAGATGGCTG CTAGAAAGTA TGCTAGGATT
GTGCAGAAAT TGGGATTCCC TGCAAAATTC AAGGATTTCA AGATTCAGAA TATTGTAGGT
TCTTGTGATG TCAAATTCCC TATAAGACTT GAAGGTCTTG CTTACTCTCA CGCTGCTTTC
TCAAGTTATG AGCCCGAGCT CTTCCCAGGG CTGATTTATA GGATGAAAGT CCCAAAAATC
GTCCTTCTAA TCTTTGTCTC TGGGAAGATC GTAATAACAG GAGCCAAGAT GAGAGATGAG
ACCTACAAAG CCTTTGAGAA TATATACCCC GTGCTCTCGG AATTCAGAAA GATACAGCAA
//
Вторичная структура белка (TBP1):

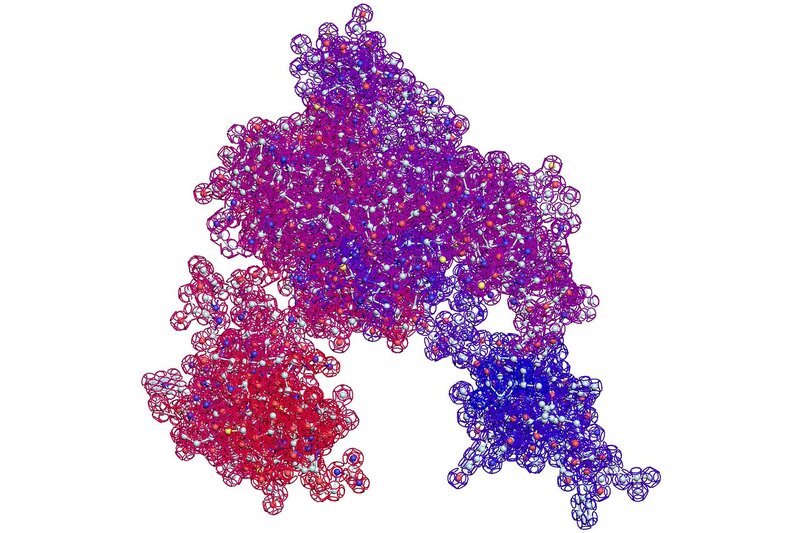
Три ортогональные проекции третичной структуры белка TBP1:

Оригинал (3000*2000): https://img-fotki.yandex.ru/get/42385/1 … 9_orig.jpg

Оригинал (3000*2000): https://img-fotki.yandex.ru/get/57422/1 … 9_orig.jpg

Оригинал (3000*2000): https://img-fotki.yandex.ru/get/56621/1 … 5_orig.jpg
Анимация третичной структуры белка TBP1: https://cloud.mail.ru/public/24yd/Z4zL3JV7c
PDB-файл с координатами атомов: https://cloud.mail.ru/public/G8ec/vgprssf2g
2016.12.13 13:52:19
Andrei01 пишет:
Какой критерий правильности алгоритма?
Кушелев: К сожалению, метод рентгеноструктурного анализа (РСА) слишком грубый, чтобы отличить результаты работы алгоритма Виктории Соколик от результата работы алгоритма Александра Кушелева.
РСА фактически может ответить на вопрос: Есть альфа-спираль или её нет. Алгоритм Виктории Соколик на этом уровне не отличается от алгоритма Александра Кушелева.
Более тонкие критерии нужно ещё найти.
2016.12.13 13:55:19
Andrei01 Так РСА может ответить на вопрос про спираль или нет? Как может быть что спирали нет? Все в воздухе висит там?
2016.12.13 14:03:54
Andrei01 пишет:
Так РСА может ответить на вопрос про спираль или нет? Как может быть что спирали нет? Все в воздухе висит там?
Кушелев: Виктория говорит, что в её алгоритме третичная структура получается в результате дополнительной обработки, фолдинга. Типа того, что вторичная структура формируется не по таблице композиционного генетического кода, а в результате взаимодействия радикалов аминокислот. Эту гипотезу и предстоит ещё проверить.
В моём алгоритме вторичная и третичная структуры белковой молекулы формируются сразу, т.е. их можно определить по таблице композиционного генетического кода.
А у Виктории другая таблица, где не все коды соответствуют структуре. А дальше нужно анализировать её фразу: "Мы получим одинаковые результаты, но разными способами"
Что касается РСА, то он видит только спирали. Более тонкую структуру белка он не видит, но она безусловно есть, и программа Пикотех показывает одинаково хорошо как спиральные участки, так и все остальные.
2016.12.13 16:49:48
Вот что ответила эксперт на вопрос какой белок считать.
The 1VOL is a structure which contains 4 chains(parts) as can be seen in the attached figure.
Each chain in different color.
I'm intrested in chains A and B (the green and light blue in the figure) which are both protein chains and not nucleotiteds.
The 2 other chains (yellow and magneta)
ggctataaaa gggctg
cagccctttt atagcc
are nocleotides and DNA or RNA but not proteins.
2016.12.13 16:55:49

Кушелев: Обратите внимание на то, что программа Пикотех в автоматическом режиме(!) построила внешне похожие по форме структуру белковых молекул из комплекса 1VOL.
На картинке из PDB мы видим альфа-спирали.
По данным программы Пикотех их нет. Это говорит о том, что данные из PDB имеют систематические ошибки, т.е. авторы, анализирующие данные РСА пытаются изображать структуру белка из альфа-спиралей, а в реальности их там нет
2016.12.13 17:07:01
Кстати, пикотехнология белков - это модельно-экспериментальный метод. И как Вы можете убедиться, он в автоматическом режиме показывает правильные контуры белковых молекул. В таком случае он не может показывать неправильно тонкую структуру белка, т.к. она является основой, на которой получены контуры молекулы.
2016.12.13 17:15:01
Skype, 2016-12-13:
[14:14:38] Andrei: Так обе версии Виктория даст?
[14:17:01] Кушелев Александр Юрьевич: Я свою версию уже выложил на форум
[14:17:33] Кушелев Александр Юрьевич: А Виктория может переслать Вам свой вариант PDB-файлов и т.д.
[15:51:04] Andrei: А где pdb файл ваш там?
[15:57:17] Andrei: Также нужно точное время вычислений компьютером
[16:38:00] Кушелев Александр Юрьевич:
https://cloud.mail.ru/public/CFfm/qjhzdX63v
https://cloud.mail.ru/public/G8ec/vgprssf2g
[16:38:26] Кушелев Александр Юрьевич: Это два PDB-файла двух белковых молекул, входящих в комплекс 1VOL
[16:38:43] Кушелев Александр Юрьевич: Точное время вычисления я сказать не могу, но не больше 1 секунды
[16:39:02] Кушелев Александр Юрьевич: Речь идёт о вычислении координат атомов и записи PDB-файла
[16:40:56] Кушелев Александр Юрьевич: Что касается построения пикотехнологической модели, которая выглядит так:
https://cloud.mail.ru/public/KJ8y/1yG19gQTW
https://cloud.mail.ru/public/24yd/Z4zL3JV7c
то на это может уйти несколько минут. Чем больше аминокислот, тем дольше 3DS Max будет строить модель.
2016.12.13 18:07:49
Kushelev пишет:
Андрей, спросите у заказчика, работают ли они с молекулярной динамикой и доккингом больших белков. И ещё предупредите, что в моделях структурных шаблонов есть клэш (перекрывание атомов далекоотстоящих друг от друга) и однотипные значения углов. Если её это не смутит и не напугает с непривычки (я уже наблюдала такую реакцию), то я подскажу как с этим справиться. Даже если модели будут разлетаться при оптимизации методами МД, есть способ пошаговой оптимизации с учетом географии медленных кодонов.
Уважаемая Виктория!
Вы смоделировали структуры белков с помощью алгоритма, который учитывает коды 310-спирали или не учитывает?
А.Ю., Вы знаете ответ на свой вопрос. Напомню, что третий нуклеотид кодонов детерминирует лишь один из трех разновидностей вторичных структур в белке: спираль, бета-тяж или поворот. За разновидности спиралей отвечает специфика хвостов аминокислотных остатков в них входящих. А это уже не к коду, а к фолдингу.
2016.12.16 02:57:51
Татьяна Рясина пишет:
Про потенциальную заказчицу
На основании её постановки задания вы можете догадаться, что именно она исследует и какой результат хочет получить или проверить? И какие свойства ей нужно предсказать?
Кушелев: Она исследует комплекс 1VOL, состоящий из двух субъединиц белка и двух одноцепочечных РНК или ДНК. Цель таких исследований обычно заключается в понимании механизма взаимодействия белков с ДНК/РНК. Правильные модели безусловно могут дать больше информации об этом, чем неправильные. Поэтому я и планирую в ближайшее время создать PDB-файлы ДНК/РНК и всего комплекса 1VOL. По существу такая работа может привести к важным научным открытиям. Сейчас нобелевские премии присуждаются за менее важные и менее достоверные открытия
2016.12.20 10:47:12
[9:37:56] Andrei: так что там в PDB про электронный слой есть?
[10:16:25] Кушелев Александр Юрьевич: В PDB-стандарте задаются только координаты атомных ядер. Чтобы задать координаты электронов, нужно менять стандарт.
[10:16:51] Andrei: а расширить его можно?
[10:17:33] Кушелев Александр Юрьевич: Да. В рассылке я опубликовал проект стандарта PPDB
[10:17:37] Andrei: что дадут электроны как добавка?
[10:17:56] Кушелев Александр Юрьевич: http://nanoworld88.narod.ru/data/297.htm
[10:18:11] Кушелев Александр Юрьевич: Новый стандарт - новый уровень точности
[10:19:35] Andrei: ок
[10:19:50] Andrei: послал им, подождем что скажут
[10:20:42] Кушелев Александр Юрьевич: ОК! Самое интересное, если сработает автоматический докинг, и пикотехнологические модели соберутся в "матрёшку" 1VOL
[10:21:41] Andrei: а вручную почему будет неправильно?
[10:23:25] Кушелев Александр Юрьевич: Вручную правильно, но трудоёмко. А если заказчик запустит автоматический докинг, и "матрёшка" соберётся сама, то как говорится, "комментарии излишни" ![]()
[10:23:45] Кушелев Александр Юрьевич: Таких случайностей не бывает.
[10:24:00] Andrei: я написал им что можно и автоматически собрать
[10:24:10] Кушелев Александр Юрьевич: Отлично!
Подолжение
Совмещение пикомодели комплекса 1VOL с ДНК https://nano-world-articles.nethouse.ru/articles/326684