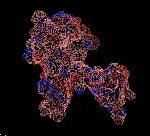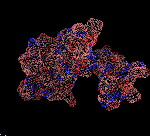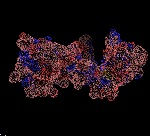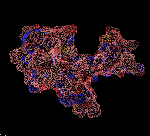
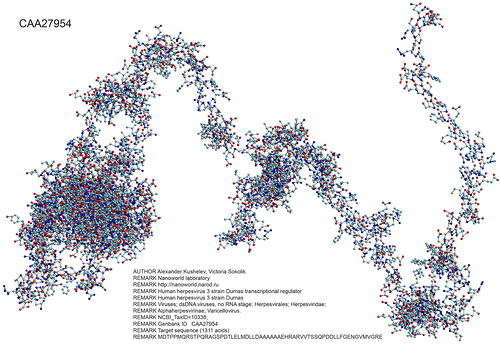
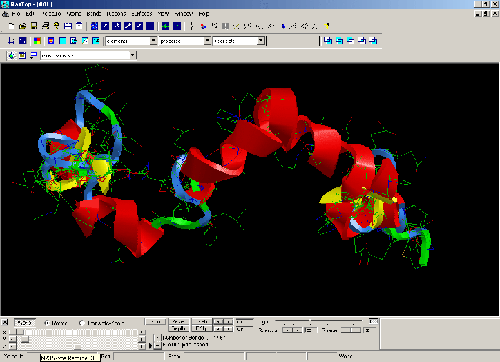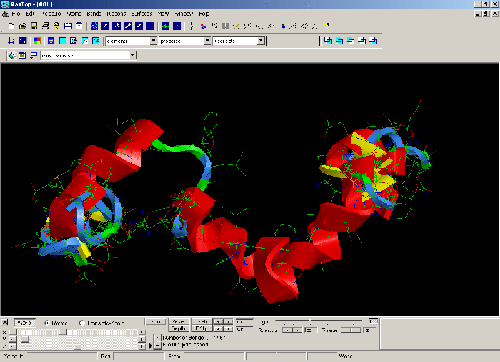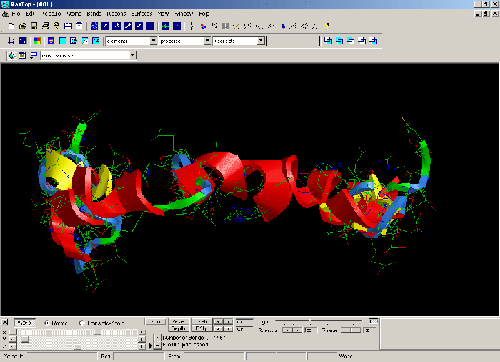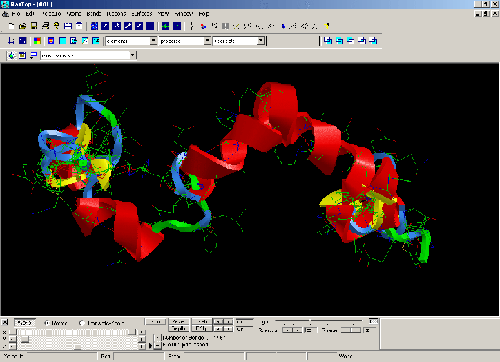
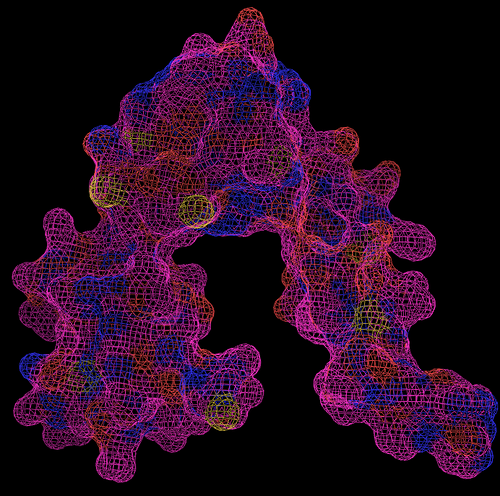
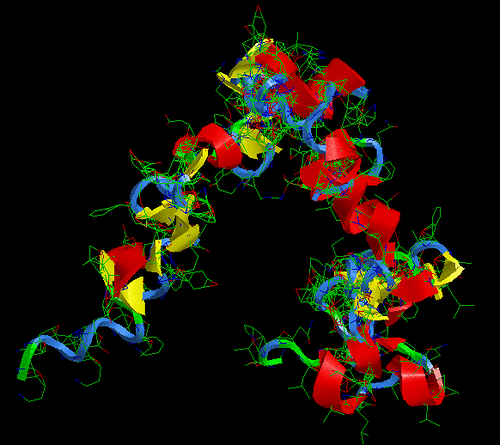
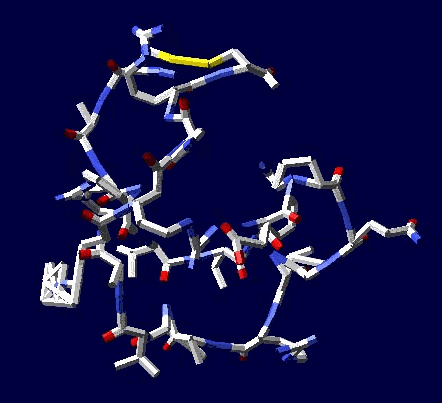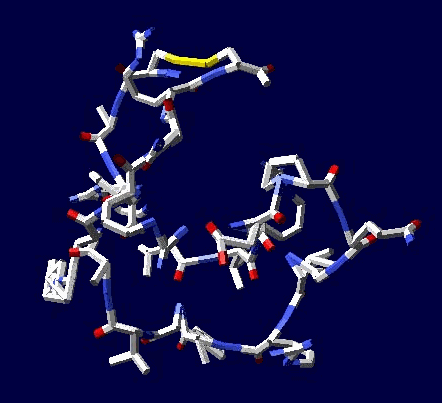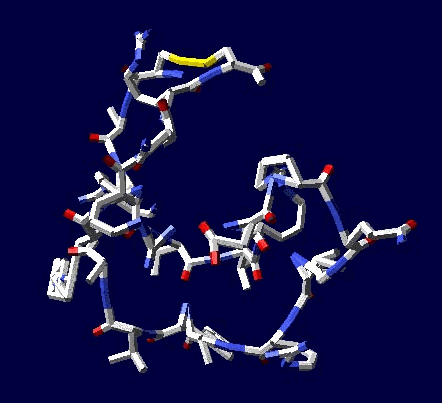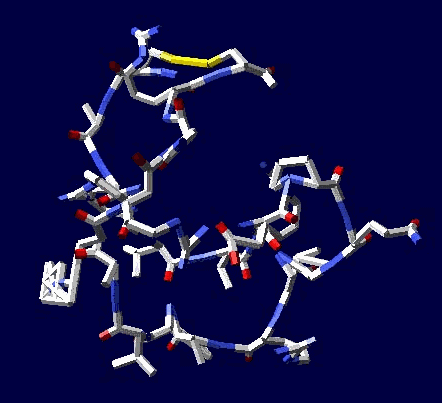
ПРОДУКТ
Kushelev пишет:

Оригинал: http://img-fotki.yandex.ru/get/5907/126 … 2_orig.png

Оригинал: http://img-fotki.yandex.ru/get/5908/126 … 2_orig.png

Оригинал: http://img-fotki.yandex.ru/get/6205/126 … a_orig.png

Оригинал: http://img-fotki.yandex.ru/get/6104/126 … d_orig.png
Оригинал: http://img-fotki.yandex.ru/get/6204/126 … 3_orig.png

Оригинал: http://img-fotki.yandex.ru/get/6104/126 … 4_orig.png

Оригинал: http://img-fotki.yandex.ru/get/6205/126 … 9_orig.png
Кушелев: Концовка хорошая: ... пикотехнология – это современный, точный и удобный методологический подход в арсенале молекулярной биологии для моделирования пространственной структуры белков, исходя из той информации генома о них, которой располагает сама
ПРОДВИЖЕНИЕ
2012.04.03 23:41:25
Как стать лидером РСА, ЯМР, нейтронной дифракции ...
[03.04.2012 10:57:55] Кушелев Александр Юрьевич: Я могу ежедневно делать по 10 PDB-файлов с координатами атомов белка. Такие файлы кто-то покупает более 1000 штук в год по средней цене 10 000 евро. Но выйти на этот рынок пока не удаётся...
[03.04.2012 10:58:34] Кушелев Александр Юрьевич: Советуют выходит через лаборатории РСА, ЯМР и нейтронной дифракции. "Лидером становится тот, кто предлагает максимально широкий спектр услуг"
[03.04.2012 10:58:57] Кушелев Александр Юрьевич: Если удастся заинтересовать лаборатории, то через них можно попасть на этот рынок
[03.04.2012 10:59:08] Кушелев Александр Юрьевич: Тоже своеобразная интернет-реклама нужна...
[03.04.2012 11:16:57] Кушелев Александр Юрьевич: Интересно, может ли сработать интернет-реклама в случае специфического товара (pdb-файлов) ?
[03.04.2012 11:18:13] Кушелев Александр Юрьевич: Мне советовали сделать прямые рассылки по лабораториями, которые продают свои pdb-файлы и предложить им бесплатно (на первых порах) добавлять к своим ещё и мои pdb-файлы, которые являются дополнительной услугой (бесплатной!)
[03.04.2012 11:18:41] Кушелев Александр Юрьевич: Клиенты через некторое время могут оценить мои pdb-файлы, которые по многим показателям превосходят стандартные
[03.04.2012 11:18:55] Кушелев Александр Юрьевич: После этого может появиться спрос
[03.04.2012 11:20:40] Кушелев Александр Юрьевич: Как реагирует первый клиент, можно посмотреть на форуме лаборатории Наномир
[03.04.2012 11:20:48] Кушелев Александр Юрьевич: Irinaval2012
[03.04.2012 11:28:38] T: pdb - это трехмерные?
[03.04.2012 11:30:01] Кушелев Александр Юрьевич: Это координаты атомов, по которым стандартные браузеры показывают трёхмерные модели молекул
[03.04.2012 11:32:01] T: так, а Вы мне скажите вот что, если я обычный пользователь интернета, но мне нужен белок, как я его буду искать в интернете. Т.е. какие примерные поисковые запросы, чтобы я мог оценить эффективность и востребовыемость
[03.04.2012 11:32:08] T: ?
[03.04.2012 11:46:26] T: просто примерно. А там я уже ма подберу. У меня есть статистика запросов. оценим.
[03.04.2012 11:48:57] Кушелев Александр Юрьевич: Обычному пользователю вряд ли нужны структуры белков. Это нужно фармацевтическим компаниям, биотехнологическим фирмам, генным инженерам, нанотехнологам...
[03.04.2012 11:49:17] Кушелев Александр Юрьевич: Обычные люди не заморачиваются строением белков в частности и структурой материи вообще
[03.04.2012 11:49:35] Кушелев Александр Юрьевич: А для любопытных я сделал это
[03.04.2012 11:50:08] Кушелев Александр Юрьевич: Подробнее:http://nanoworld88.narod.ru/data/212.htm
[03.04.2012 11:51:21] T: Ок, компания , которая ищет лабораторию , которая в свлою очередь делает то, что делаете Вы. Как это компания будет искать информацию?
[03.04.2012 11:52:07] T: Для примера:
Мне нужен фильм?
"скачать фильм"
Нужны окна:
"Пластиковые окна в Москве"
[03.04.2012 11:52:53] T: Сколько наименований у различных белков?
[03.04.2012 11:54:18] Кушелев Александр Юрьевич: Лаборатория РСА (по-английски X-ray)
[03.04.2012 11:54:33] Кушелев Александр Юрьевич: Лаборатория ЯМР (ядерного магнитного резонанса)
[03.04.2012 11:54:44] Кушелев Александр Юрьевич: Лаборатория нейтронной дифракции
[03.04.2012 11:54:59] Кушелев Александр Юрьевич: Вот по этим словам выходят на лаборатории, которые продают файлы PDB
[03.04.2012 11:55:10] Кушелев Александр Юрьевич: Хотя может и не через интернет...
[03.04.2012 11:55:22] T: А эти лаборатории только PDB занимаются?
[03.04.2012 11:56:03] T: А формат PDB используется только для построений атомов белков или еще для чего?
[03.04.2012 11:56:12] Кушелев Александр Юрьевич: Они берут у заказчика кристаллы белка (нужно месяца 2-3 выращивать кристалл из исследуемого белка), делают рентгенограмму и анализируют
[03.04.2012 11:56:47] Кушелев Александр Юрьевич: По результатам анализа создают pdb-файл, который имеет реальную точность от 5 до 500 ангстрем
[03.04.2012 11:57:15] Кушелев Александр Юрьевич: Мой файл имеет точность в ~1000 раз выше, но не для всех белков
[03.04.2012 11:57:39] Кушелев Александр Юрьевич: Форма PDB для белков используется точно, а для чего ещё, я не в курсе
[03.04.2012 11:58:50] T: Просто , если для только для белков, то употребление "PDB" в любом поисковом запросе будет означать, что это потенциальный клиент
[03.04.2012 12:00:12] T: Ну не однозначно, конечно. Может человек ищет компанию с таким названием
[03.04.2012 12:00:13] Кушелев Александр Юрьевич: Понятно
[03.04.2012 12:01:47] T: А что за программа генерирует этот PDB файл?
[03.04.2012 12:02:04] Кушелев Александр Юрьевич: Я делаю с помощью программы pdb-файлы, которые точнее стандартных, но стандартные всё же дополняют мои, т.к. я моделирую только рибосому, а рентген просвечивает белки, которые могут модифицироваться после сборки рибосомой. Поэтому хотя у них и хуже качество, зато есть дополнительная информация о постобработке.
[03.04.2012 12:02:15 | Изменены 12:02:21] Кушелев Александр Юрьевич: Моя программа "Пикотехнология"
[03.04.2012 12:02:49] Кушелев Александр Юрьевич: Сделано несколько версий, и будет сделано ещё, т.к. "нет предела совершенству"
[03.04.2012 12:03:46] T: ну как я и предполагал - не однозначно.
"pdb *.PalmDOC (он же — PDB и AportisDoc). Еще один популярный Palm-формат. Его нормально воспринимает большинство программ для чтения электронных книг. Но при этом степень сжатия гораздо ниже, чем у iSilo.PDB, а графика не поддерживается вовсе. "
[03.04.2012 12:04:48] Кушелев Александр Юрьевич: Это другой формат
[03.04.2012 12:04:54] Кушелев Александр Юрьевич: Название совпало
[03.04.2012 12:05:03] Кушелев Александр Юрьевич: Вот биологический:
[03.04.2012 12:05:08] T: Назовите любой белок, хотя я не понимаю о чем говорю. Белок - это часть элемента. Т.е. есть например белок сульфадонатрихлора.
[03.04.2012 12:06:12] Кушелев Александр Юрьевич: Его можно смотреть в текстовом формате
[03.04.2012 12:06:18] Кушелев Александр Юрьевич: редакторе
[03.04.2012 12:06:55] Кушелев Александр Юрьевич: А этот файл сделан моей программой
[03.04.2012 12:07:45] T: так, значит вот название "Bacteroides thetaiotaomicron"?
[03.04.2012 12:08:29] Кушелев Александр Юрьевич: Это название белка, а расширение файла pdb
[03.04.2012 12:09:12] Кушелев Александр Юрьевич: В банке белковых структур, Protein Data Base (PDB) находятся ~80 000 файлов PDB с координатами атомов белковых молекул
[03.04.2012 12:09:26] Кушелев Александр Юрьевич: Но это уже "б/у" файлы
[03.04.2012 12:09:35] Кушелев Александр Юрьевич: За них уже уплачено в среднем по 10 000 евро
[03.04.2012 12:09:53] Кушелев Александр Юрьевич: А каждый год заказчики платят за несколько тысяч новых таких файлов
[03.04.2012 12:10:03] T: да, я понял. А сколько существует известных названий белков, в мире
[03.04.2012 12:10:15] T: ой
[03.04.2012 12:10:21] Кушелев Александр Юрьевич: В человеке по современным оценкам около 5 000 000 разных белков
[03.04.2012 12:10:22] T: вы же написали уже
[03.04.2012 12:10:36] Кушелев Александр Юрьевич: И моя программа практически все их может показать за неделю
[03.04.2012 12:10:51] T: Хм, это очень интересно
[03.04.2012 12:10:55] Кушелев Александр Юрьевич: А рентген может показать только 3%, т.к. остальные 97 не кристаллизуются
[03.04.2012 12:11:16] Кушелев Александр Юрьевич: Поэтому моя программа может как бы в 30 раз больше рентгеновской лаборатории
[03.04.2012 12:11:21] Кушелев Александр Юрьевич: по количеству белков
[03.04.2012 12:11:31] Кушелев Александр Юрьевич: И в ~1000 раз лучше по точности
[03.04.2012 12:11:42] Кушелев Александр Юрьевич: Но только те белки, которые не дорабатываются после сборки
[03.04.2012 12:11:50] Кушелев Александр Юрьевич: Доработку я пока смоделировать не могу
[03.04.2012 12:12:05] Кушелев Александр Юрьевич: Но вручную некоторые доработки можно смоделировать
[03.04.2012 12:12:09] T: ок, а будут вопросы по качеству, т.е. они наверно не поймут почему вы сможете делать с помощью программы то, что делаю на данный момент рентгеном?
[03.04.2012 12:12:22] Кушелев Александр Юрьевич: Это я сейчас и предлагаю первому заказчику Irinaval2012
[03.04.2012 12:13:02] Кушелев Александр Юрьевич: Я несколько лет назад встречался с руководителями трёх лабораторий РСА (рентгено-структурного анализа)
[03.04.2012 12:13:13] Кушелев Александр Юрьевич: Сначала на меня смотрели дикими глазами и говорили:
[03.04.2012 12:13:52] Кушелев Александр Юрьевич: Как же так? У Вас программа показывает спиральные участки с ошибкой до 5 аминокислотных остатков! Рентген не может ошибиться даже на 1
[03.04.2012 12:14:19] Кушелев Александр Юрьевич: Я им объясняю, что ошибка на 1 атом (типовая для рентгена) может означать, что мы перескочили на соседний атом
[03.04.2012 12:14:22] Кушелев Александр Юрьевич: -Ну да...
[03.04.2012 12:14:35] Кушелев Александр Юрьевич: А соседний атом может принадлежать соседнему аминокислотному остатку
[03.04.2012 12:14:36] Кушелев Александр Юрьевич: -Да
[03.04.2012 12:14:48] Кушелев Александр Юрьевич: А соседний остаток может принадлежать соседнему витку альфа-спирали
[03.04.2012 12:14:50] Кушелев Александр Юрьевич: -Верно
[03.04.2012 12:15:08] Кушелев Александр Юрьевич: Вот Вам и ошибка на 5 остатков (если спираль развернуть в цепочку)
[03.04.2012 12:15:11] Кушелев Александр Юрьевич: -Ну надо же...
[03.04.2012 12:15:19] Кушелев Александр Юрьевич: И так реагировали во всех трёх лабораториях
[03.04.2012 12:15:31] T: Тогда все понятно
[03.04.2012 12:15:34] Кушелев Александр Юрьевич: Тогда у меня ещё не было настроенной программы
[03.04.2012 12:15:41] Кушелев Александр Юрьевич: А сейчас ситуация изменилась
[03.04.2012 12:15:47] Кушелев Александр Юрьевич: Я могу делать pdb-файлы
[03.04.2012 12:16:11] Кушелев Александр Юрьевич: Вот и надо бы их предложить лабораториям, чтобы они могли своим клиентам дать более широкий спектр услуг
[03.04.2012 12:16:17] Кушелев Александр Юрьевич: Наши файлы дополняют друг друга
[03.04.2012 12:16:45] Кушелев Александр Юрьевич: Пусть мои файлы "весят" 90%, а их 10%, но на первых порах я готов их делать бесплатно
[03.04.2012 12:16:55] T: т.е. потенциальные коиенты - лабратории, а не прямые заказчики.
[03.04.2012 12:17:09] Кушелев Александр Юрьевич: А когда клиенты разберутся, "кто есть Who", тогда можно и рыночные цены установить
[03.04.2012 12:17:27] Кушелев Александр Юрьевич: Клиенты - заказчики, но на них быстрее выйти через лаборатории
[03.04.2012 12:17:38] Кушелев Александр Юрьевич: Ведь связи с лабораториями у них уже установлены
[03.04.2012 12:17:45] Кушелев Александр Юрьевич: Лабораторий гораздо меньше, чем клиентов
[03.04.2012 12:17:55] Кушелев Александр Юрьевич: Их легче "обойти и убедить"
[03.04.2012 12:18:24] Кушелев Александр Юрьевич: Пусть лаборатории выступят "менеджерами" со своими клиентскими базами
[03.04.2012 12:18:44] Кушелев Александр Юрьевич: Клиентские базы собираются десятилетиями
[03.04.2012 12:19:48] T: ну не скажите. Лабратории это Ваши прямые конкуренты. Они же понимают по сути, что Вы их вытесните
[03.04.2012 12:20:00] T: А это их "бизнес".
[03.04.2012 12:21:14] Кушелев Александр Юрьевич: Вряд ли они так решат. Дело в том, что мы друг друга дополняем, поэтому с одной стороны я их не вытесню до конца, а главное, что та лаборатория, которая будет предлагать расширенный спектр услуг, обойдёт своих конкурентов и сможет делить гораздо бОльшую прибыль со мной
[03.04.2012 12:21:22] Кушелев Александр Юрьевич: Я же не жадный
[03.04.2012 12:21:48] Кушелев Александр Юрьевич: Поэтому те, кто согласятся со мной сотрудничать вырвутся в лидеры и умножат свои доходы однозначно
[03.04.2012 12:21:58] Кушелев Александр Юрьевич: А недоверчивые могут закрепить свои права нотариально
[03.04.2012 12:22:44] T: Это все так, но Вы не учитываете, что "лень" о которой Мы ранее говорили и здесь присутствует - никто ничего не хочет менять, только, если на этом можно заработать
[03.04.2012 12:23:22] Кушелев Александр Юрьевич: Так объяснить, что они смогут работать меньше (лень же!), а получать в десятки раз больше
[03.04.2012 12:23:37] T: Конечно, можно предложить им сотрудничество, но Вы это уже делали же
[03.04.2012 12:23:52] Кушелев Александр Юрьевич: Конечно, откликнутся из 100 лабораторий 1...5, но этого будет вполне достаточно для выхода на рынок
[03.04.2012 12:24:14] Кушелев Александр Юрьевич: Я этого ещё не делал, т.к. не было программы, которая делает pdb-файлы
[03.04.2012 12:24:47] Кушелев Александр Юрьевич: Они тогда (несколько лет назад) поняли, но продавать было нечего. А теперь тех людей уже нет. Кто на пенсию ушёл, кто уволился, нужно с нуля начинать
[03.04.2012 12:27:42] T: Вот всегда настаиваю на самостоятельном видЕнии бизнеса. Т.е. я за открыть лабораторию и искать прямых клиентов. И пусть уже "перекупщики", а именно так их и следует называть - будут прикреплять свои 10% к Вашим 90% и это будет справедливо.
[03.04.2012 12:27:49] Кушелев Александр Юрьевич: И может быть интернет поможет это сделать быстрее и лучше
[03.04.2012 12:27:57] T: Конечно же деньги...
[03.04.2012 12:28:39] Кушелев Александр Юрьевич: Делать свою лабораторию - можно время упустить
[03.04.2012 12:28:53] Кушелев Александр Юрьевич: Пусть они начнут, а как деньги пойдут, так и сделать лабораторию и т.д.
[03.04.2012 12:29:16] T: А могут идею украсть?
[03.04.2012 12:29:19] Кушелев Александр Юрьевич: Они-то сегодня продают. Я сегодня могу 10 файлов по 10 000 евро делать
[03.04.2012 12:29:31] Кушелев Александр Юрьевич: Эту идею украсть очнь сложно
[03.04.2012 12:29:41] T: А патентовать?
[03.04.2012 12:29:44] Кушелев Александр Юрьевич: Дело в том, что я каждый месяц могу модифицировать программу
[03.04.2012 12:29:57] Кушелев Александр Юрьевич: Она будет выдавать всё более качественные файлы
[03.04.2012 12:30:33] Кушелев Александр Юрьевич: Может получиться как с аудио-форматами: Вы купили CD? Теперь купите то же самое на DVD ![]()
[03.04.2012 12:30:47] Кушелев Александр Юрьевич: Одни и те же белки можно будет каждый месяц делать всё точнее
[03.04.2012 12:30:52] Кушелев Александр Юрьевич: А их 5 000 000
[03.04.2012 12:31:02] Кушелев Александр Юрьевич: А сегодня сделано только 80 000
[03.04.2012 12:31:30] Кушелев Александр Юрьевич: Вот покупает клиент стандантный PDB-файл
[03.04.2012 12:31:36] Кушелев Александр Юрьевич: А к нему приложен мой
[03.04.2012 12:31:37] T: Так ок, объясните, кто заказывает в лобраториях эти белки?
[03.04.2012 12:31:55] T: для чего
[03.04.2012 12:32:09] Кушелев Александр Юрьевич: Белки заказываю фармацевтические компании, биотехнологические, нанотехнологические, генно-инженерные
[03.04.2012 12:32:26] Кушелев Александр Юрьевич: Для создания новых лекарств, продуктов питания (в т.ч. ГМО), и т.д.
[03.04.2012 12:32:36] T: понятно
[03.04.2012 12:32:49] Кушелев Александр Юрьевич: Клиент видит, что мой PDB-файл - это 90%
[03.04.2012 12:32:54] Кушелев Александр Юрьевич: Это уже хорошо
[03.04.2012 12:33:13] T: ну при условии, что там будут ваши контактные данные
[03.04.2012 12:33:29] Кушелев Александр Юрьевич: Потом он читает, что его белки, которые не может определить лаборатория РСА, могу определить я, т.е. 97% заказов могу выполнить только я
[03.04.2012 12:33:49] Кушелев Александр Юрьевич: Контактные данные можно замаскировать
[03.04.2012 12:33:59] T: каким образом?
[03.04.2012 12:34:18] Кушелев Александр Юрьевич: Достаточно написать такие слова, как пикотехнология, пикотехнологическая модель, и Яндекс сразу выведет на мой сайт
[03.04.2012 12:34:26] Кушелев Александр Юрьевич: Попробуйте набрать "Пикотехнология"
[03.04.2012 12:34:54] Кушелев Александр Юрьевич: А это слово можно использовать в описании файла
[03.04.2012 12:35:02] T: ну да, как бренд
[03.04.2012 12:35:03] Кушелев Александр Юрьевич: Внутри PDB-ФАЙЛА
[03.04.2012 12:35:07] T: угу
[03.04.2012 12:35:24] T: пикотехнологическая модель
[03.04.2012 12:35:48] Кушелев Александр Юрьевич: Заказчики могут и сегодня знать про меня, но не решаться
[03.04.2012 12:36:08] Кушелев Александр Юрьевич: А если им мои файлы предложат в лаборатории РСА, то это будет совсем другой "компот"
[03.04.2012 12:36:43] T: так, ок. Как я понял - задача найти все вышеописанные лаборатории.
[03.04.2012 12:36:56] Кушелев Александр Юрьевич: Кстати, у меня уже несколько недель висит на форуме тема
[03.04.2012 12:37:16] Кушелев Александр Юрьевич: Но форум-то потенциальные заказчики не находят...
[03.04.2012 12:37:17] T: И донести до них посредством интернета о пикотехнологии
[03.04.2012 12:37:28] Кушелев Александр Юрьевич: Вам виднее, как это сделать
[03.04.2012 12:37:32] Кушелев Александр Юрьевич: У меня не получается...
[03.04.2012 12:37:50] T: так, а что Вы уже пробывали?
[03.04.2012 12:38:10] Кушелев Александр Юрьевич: Мне говорили, что если письмо приходит не с университетского (американского!) сервера, то его сразу в мусорку кидают
[03.04.2012 12:38:32] Кушелев Александр Юрьевич: Я делал попытки рассылать письма, но ответов нет вообще
[03.04.2012 12:38:37] Кушелев Александр Юрьевич: Как в мусорку...
[03.04.2012 12:38:48] Кушелев Александр Юрьевич: Не с того сервера...
[03.04.2012 12:39:05] Кушелев Александр Юрьевич: Сказали, что нужно через университетский американский сервер
[03.04.2012 12:39:12] Кушелев Александр Юрьевич: Тогда письма прочтут
[03.04.2012 12:39:23] Кушелев Александр Юрьевич: И может быть несколько процентов лабораторий отреагируют
[03.04.2012 12:39:39] T: А где он такой есть, и кто имеет доступ в него?
[03.04.2012 12:39:49] T: Или просто можно американский сервер
[03.04.2012 12:39:53] Кушелев Александр Юрьевич: Тут, как разжигание костра. Достаточно, чтобы одна веточка из 100 загорелась
[03.04.2012 12:40:07] Кушелев Александр Юрьевич: Не просто американский, а университетский!
[03.04.2012 12:40:15] Кушелев Александр Юрьевич: Надо там знакомых иметь
[03.04.2012 12:40:27] Кушелев Александр Юрьевич: У меня есть знакомый, но у него так руки и не дошли
[03.04.2012 12:40:34] T: так, Университет в Америке - любой?
[03.04.2012 12:40:44] T: или конкретный
[03.04.2012 12:40:56] Кушелев Александр Юрьевич: Где занимаются нанотехнологией и структурами белков
[03.04.2012 12:41:04] Кушелев Александр Юрьевич: Иначе лажа выйдет
[03.04.2012 12:41:30] T: так, если вариант не со знакомыми
[03.04.2012 12:41:42] Кушелев Александр Юрьевич: Я не представляю...
[03.04.2012 12:42:04] T: Нет, ну я представляю, только вот нельзя так
[03.04.2012 12:42:17] Кушелев Александр Юрьевич: Системные программисты могут просто подставить обратный адрес нужного университета
[03.04.2012 12:42:19] Кушелев Александр Юрьевич: Я так не умею
[03.04.2012 12:42:34] T: ну это понятно
[03.04.2012 12:42:38] Кушелев Александр Юрьевич: Почему нельзя?
[03.04.2012 12:42:49] T: мы именами светим
[03.04.2012 12:43:07] T: представляете приходит письмо от того, кто это письмо не отправлял
[03.04.2012 12:43:16] Кушелев Александр Юрьевич: Надо чтобы по обратному адресу письмо к нам пришло, а не куда-то
[03.04.2012 12:43:18] T: и подпись "наномир"
[03.04.2012 12:43:41] Кушелев Александр Юрьевич: Это - не проблема
[03.04.2012 12:44:31] Кушелев Александр Юрьевич: Можно в письме в конце объяснить, что мы послали письмо из университета, будучи там проездом, а обратное письмо отправляйте нам в лабораторию
[03.04.2012 12:44:59] Кушелев Александр Юрьевич: Университетский адрес - формальность, чтобы письмо начали читать
[03.04.2012 12:45:07] T: думаю, что можно так. Зарегестрировать доменное имя в зоне com. Сделать грамотную сайт-визитку на англ языке.
[03.04.2012 12:45:30] Кушелев Александр Юрьевич: А если уже начнут читать, то кто-то поймёт, что ему предлагают дело, и начнётся деловая переписка
[03.04.2012 12:46:19] Кушелев Александр Юрьевич: Мне объяснили, что существуют фильтры, которые пропускают письма только с униветситетских серверов или адресов.
[03.04.2012 12:46:35] Кушелев Александр Юрьевич: Нам не с людьми нужно бороться, а с фильтрами
[03.04.2012 12:46:45] Кушелев Александр Юрьевич: Если люди письмо прочтут, то дело будет сделано
[03.04.2012 12:46:56] T: Ну так фильтры обойти можно
[03.04.2012 12:47:02] Кушелев Александр Юрьевич: Главное, чтобы письма дошли до человеческих глаз
[03.04.2012 12:47:07] Кушелев Александр Юрьевич: Я об этом и говорю
[03.04.2012 12:47:25] Кушелев Александр Юрьевич: Но я не знаю, как эти фильтры работают
[03.04.2012 12:47:29] T: дойдут. А на анг языке нужно письмо?
[03.04.2012 12:47:36] Кушелев Александр Юрьевич: Может быть там IP имеет значение
[03.04.2012 12:47:43] Кушелев Александр Юрьевич: Конечно на английском
[03.04.2012 12:47:50] T: может и так, но на это тоже есть решение
[03.04.2012 12:47:56] Кушелев Александр Юрьевич: Англичане и американцы по русски не читают
[03.04.2012 12:48:19] Кушелев Александр Юрьевич: А русские за pdb-файлы не платят
[03.04.2012 12:48:34] Кушелев Александр Юрьевич: Хотя может и платят...
[03.04.2012 12:48:37] T: вот до меня сейчас только дошло, что лаборатории все заграничные.
[03.04.2012 12:48:56] Кушелев Александр Юрьевич: Там уровень цен другой
[03.04.2012 12:49:08] Кушелев Александр Юрьевич: А в России всё в застое...
[03.04.2012 12:49:18] T: Письмо отправим
[03.04.2012 12:49:25] T: нужен текст, и грамотно перевести
[03.04.2012 12:49:33] Кушелев Александр Юрьевич: Зато российские заказчики могут по-русски писать и создавать рекламу
[03.04.2012 12:49:51] Кушелев Александр Юрьевич: Текст лучше напишет Виктория Соколик
[03.04.2012 12:49:57] Кушелев Александр Юрьевич: У неё больше 100 научных трудов
[03.04.2012 12:50:13] Кушелев Александр Юрьевич: Она уже писала коротенький. Сейчас попробую найти
[03.04.2012 12:51:08] Кушелев Александр Юрьевич: Виктория Соколик: Уважаемые коллеги, Вашему вниманию предоставляется услуга -- моделирование 2D и 3D структуры любого белка без ограничений в его размере и степени изученности с помощью программного обеспечения, базирующемся на принципиально новом подходе декодирования нуклеотидной последовательности, детерминирующей данный белок.
Всё, что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB).
В течение 1-3 суток мы готовы предоставить Вам схему вторичной структуры заказанного белка (2D), модель его пространственной структуры (3D) в виртуальном пространстве, а также файл .pdb с координатами каждого атома белка.
Файл .pdb может быть использован по аналогии с файлами закристаллизованных белков из PDB банка для дальнейшего конформационного анализа белка методами молекулярной динамики с учётом физико-химической специфики микроокружения белка или его взаимодействия с лигандами.
Таким образом, Вы сможете максимально быстро удобным для Вас способом (по электронной почте, на сайте либо на электронном носителе) получить информацию о структурном шаблоне Вашего белка.
Первые 10 заказов -- бесплатно . Цена следующих заказов -- договорная.
[03.04.2012 12:51:22] Кушелев Александр Юрьевич: Подробнее:http://nanoworld88.narod.ru/data/288.htm
[03.04.2012 12:51:24] T: с обоснованием реальности метода
[03.04.2012 12:51:37] Кушелев Александр Юрьевич: В этом выпуске рассылки наша свежая статья с Викторией
[03.04.2012 12:52:17] Кушелев Александр Юрьевич: Обоснование тут непростое.. Заказчики сами должны "пощупать", сравнить и понять
[03.04.2012 12:52:29] Кушелев Александр Юрьевич: Для этого предлагается сделать пробные бесплатные заказы
[03.04.2012 12:52:44] Кушелев Александр Юрьевич: Очень быстро распробуют и начнут заказывать массово
[03.04.2012 12:52:47] Кушелев Александр Юрьевич: Главное - начать
[03.04.2012 12:53:29] T: ок, на какие адреса отправлять надо письмо
[03.04.2012 12:53:43] Кушелев Александр Юрьевич: Если бы я знал...
[03.04.2012 12:54:51] Кушелев Александр Юрьевич: Мне советовали в первую очередь выходить на коммерческие структуры, т.к. университеты заказывают файлы только у госбюджетных лабораторий и не идут на контакты с коммерческими структурами
[03.04.2012 12:55:08] Кушелев Александр Юрьевич: Мы для них - коммерческая структура
[03.04.2012 12:55:49] Кушелев Александр Юрьевич: А войти в государственную программу - уйдёт много лет
[03.04.2012 12:56:56] T: сложно
[03.04.2012 12:57:10] T: надо поразмыслить
[03.04.2012 12:57:40] Кушелев Александр Юрьевич: Да...
http://nanoworld88.narod.ru/data/280.htm
Участвуем в международном конкурсе определения структуры белка CASP10
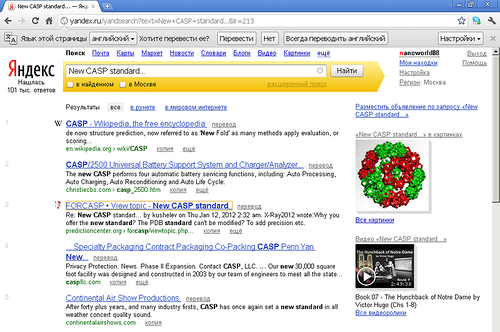
Напомню, что в 1998-ом году я предложил организаторам конкурса CASP новый стандарт представления белковых структур, т.к. старый стандарт позволяет лишь различать "спираль"/"не спираль". Это слишком грубо, не позволяет даже теоретически отличать структуры, которые не являются прямой альфа-спиралью. Организаторы CASP отклонили моё предложение в 1998-ом году. В 2011 году я открыл тему "New CASP standard" на форуме FORCASP
В обсуждении приняло участие 5 специалистов, но администрация CASP через месяц приняла решение уничтожить тему, т.е. сообщения всех участников из разных стран, поэтому нам придется участвовать в конкурсе в рамках устаревшего стандарта, что дает дополнительные возможности для злоупотреблений руководству CASP. Напомню, что в 1998-ом году накануне подведения итогов конкурса 5 лабораторий из 33 неожиданно исчезли из списка участников вместе с их данными, которые накапливались на сервере около полутора месяцев. Вот копия оригинальной таблицы участников 1998 года
Сегодня на сайте CASP можно видеть другую таблицу: http://predictioncenter.org/casp3/stats/statistics.html
В ней Вы не найдёте ни данных, полученных организаторами CASP от лаборатории Наномир, ни даже упоминания об участии лаборатории Наномир в CASP3. Поэтому мы должны быть готовы к "двойным стандартам" и в 2012-ом году. На этот раз на сайте CASP не показан даже список участников конкурса, что открывает полный простор для злоупотреблений.
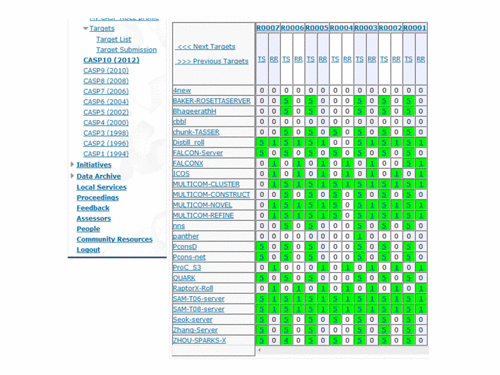
Victoria: Я нашла только список серверов с таблицей результатов:
Каждый участник конкурса может видеть только собственные данные: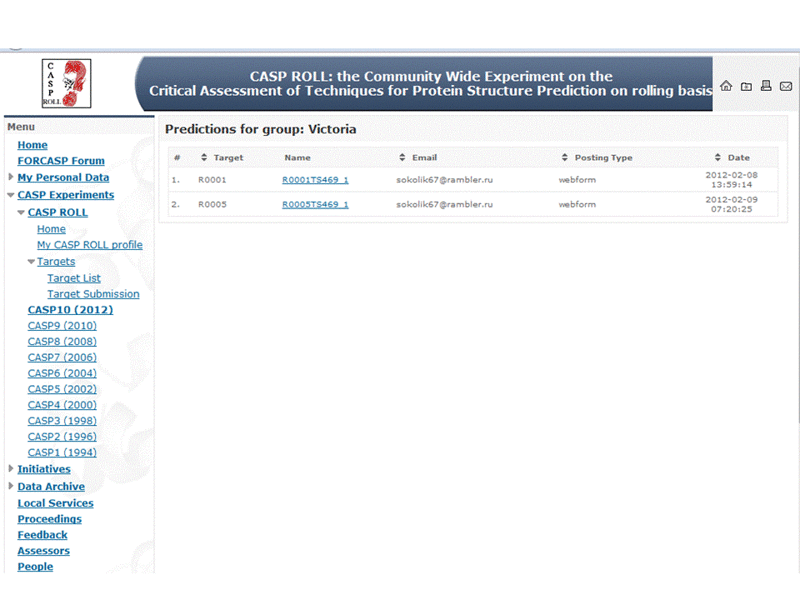
Если Вы участвуете в конкурсе CASP10, то о Вас никто из участников не знает. И Вы знаете только число участников, но не знаете, кто же участвует в конкурсе вместе с Вами? При такой организации можно любому участнику сказать всё, что угодно, например, "Очень жаль, но Вам не повезло... Снимите с него шкуру!" (из мультфильма "Алёша Попович и Тугарин Змей")
Участники могут видеть таблицу входных данных для моделирования. Это - конкурсные аминокислотные последовательности фрагментов белков. Для защиты от произвола организаторов CASP мы решили публиковать наши данные, а именно конкурсные pdb-файлы.
FT CDS 1..549
gggcatgttc ttcaattaga gtccgcatcc gacaaggcgc actatattct atctaaagat ggtaacagga ataactggta cattggtaga gggtcagata acaacaatga ctgtaccttc cactcctatg tacatggtac gaccttaaca ctcaagcagg actatgcagt agttaacaaa cacttccacg taggtcaggc cgttgtggcc actgatggta atattcaagg tactaagtgg ggaggtaaat ggctggatgc ttacctacgt gacagcttcg ttgcgaagtc caaggcgtgg actcaggtgt ggtctggtag tgctggcggt ggggtaagtg tgactgtttc acaggatctc cgcttccgca atatctggat taagtgtgcc aacaactctt ggaacttctt ccgtactggc cccgatggaa tctacttcat agcctctgat ggtggatggt tacgattcca aatacactcc aacggtctcg gattcaagaa tattgcagac agtcgttcag tacctaatgc aatcatggtg gagaacgag
//
R 0001: http://nanoworld88.narod.ru/pdb/R0001.pdb
pdb-файл целого белка
Следующее конкурсное задание, R 0005:
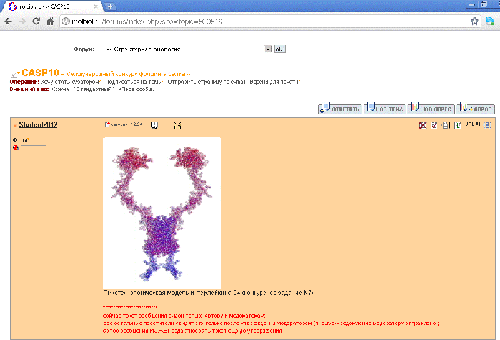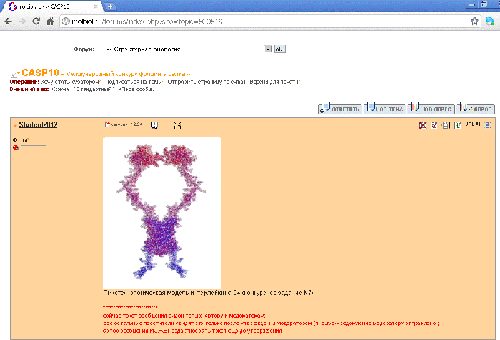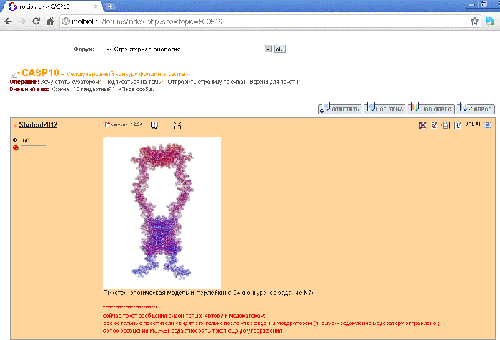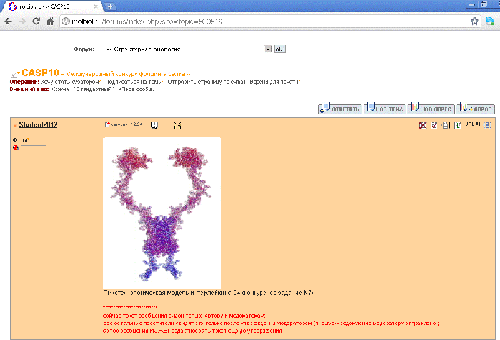
Пикотехнологическая модель
Два кадра анимации высокого качества (4000х3000)
Конкурсный фрагмент белка R 0005 (PyMOL, mesh)
R 0005 (pdb-файл целого белка)
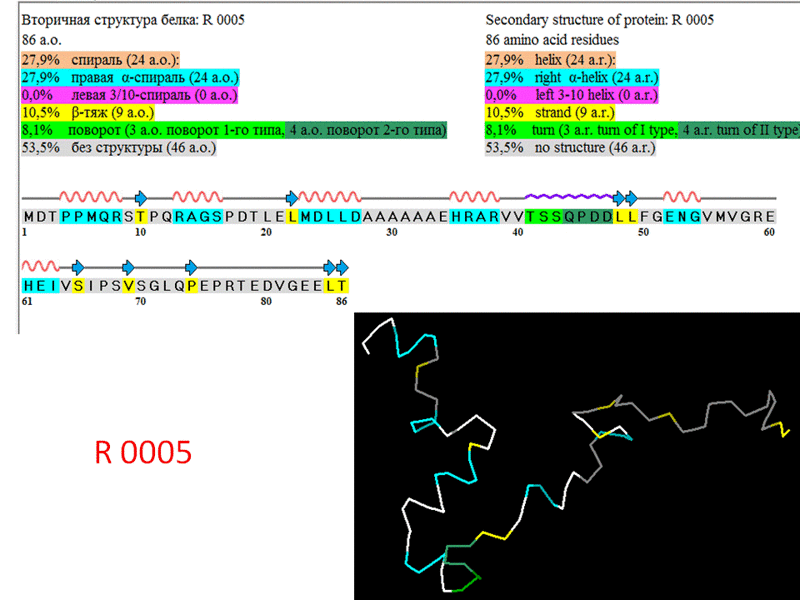
Victoria: Так моя программа TSP показывает R0005
R 0005 в программе RasTop
Подробнее см. на форуме лаборатории Наномир.
Victoria: Мне удалось найти нуклеотидную последовательность для R0006:
FT CDS 1..588
ATGTTAAAAC AACTACTGAC CGTCGTACTG CTGGCAATCT GCCTCATTAA CGTACAGGCG CAGCAACTGA CTCCTCCCGC CGGAACTTTC CGTCTGGGTA TATCGAAAGG AACCGACAGT CATTGGCTGG CTCCTCAGGA AAAAGTGAAA GGAATCGCTT TCCGGTGGAA AGCCCTGCCG GATACCCGCG GCTTTATTCT CGAAGTAGCG GTCACCTCCT TGCAGCAGGC CGATACCTTA TTCTGGAGCT TCGGAAACTG TCAGCCGGAC ATGGATATAA ACGTGTTCAG TGTAGAAGGG CAAGCCTTCA CCTGTTATTA TGGTGAAAGT ATGAAACTCC GCACCTTGCA GGCGGTCACT CCCACTGACG ATATCCGTTT GAGTAACGGC CGTCAGGACA AGACTCCCCT CTTGCTTTAT GAATCGGGGA AACGAACAGA CCGTCCTGTC CTCGCAGGGC GCTGCCCGCT CGCAGCAAAC AGTAAACTTT ATTTCTGTTT CTACGAGCAA AATGCACGAG CAGATTATAA TTATTTTATG TTGCCGGATC TTTTTGCAAA GATTGATGAA TCTAAACATA GTAAAAAA
//
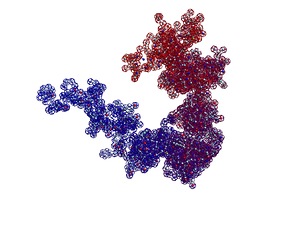
R 0006 (пикотехнологическая модель, 3DS Max)
R0006 (PyMOL, mesh)
Victoria: Программа TSP о белке R0006
R 0006 в программе RasTop
Два кадра анимации высокого качества (4000х3000)
Victoria: R0007 не простой объект ещё и тем, что это изоформа 2 IL-34, а в PDB вся информация только для изоформы 1 этого пептида. Они различаются между собой только единственной аминокислотой (Q), которой нет в 81 позиции в изоформе 1, а значит нет и её кодона в нуклеотидной последовательности. Можно конечно самим вставить любой из двух кодонов глутамина (CAA или CAG), но я переспрошу у организаторов конкурса.
Kushelev: Кушелев: Благодарю! Может быть сразу попросить у них нуклеотидные последовательности всех остальных конкурсных белков? Объяснить, как в прошлый раз, что для нашей программы аминокислотная последовательность - ничто.
Victoria: Вариант1 (САА) нуклеотидной последовательности для R0007:
FT CDS 1..483
AATGAGGAGT GCACTGTCAC GGGTTTTCTG CGGGACAAGC TGCAGTACAG GAGCCGACTT CAGTACATGA AACACTACTT CCCCATCAAC TACAAGATCA GTGTGCCTTA CGAGGGGGTG TTCAGAATCG CCAACGTCAC CAGGCTGCAA AGGGCCCAGG TGAGCGAGCG GGAGCTGCGG TATCTGTGGG TCTTGGTGAG CCTCAGTGCC ACTGAGTCGG TGCAGGACGT GCTGCTCGAG GGCCACCCAT CCTGGAAGTA CCTGCAGGAG GTGCAGACGC TGCTGCTGAA TGTCCAGCAG GGCCTCACGG ATGTGGAGGT CAGCCCCAAG GTGGAATCCG TGTTGTCCCT CTTGAATGCC CCAGGGCCAA ACCTGAAGCT GGTGCGGCCC AAAGCCCTGC TGGACAACTG CTTCCGGGTC ATGGAGCTGC TGTACTGCTC CTGCTGTAAA CAAAGCTCCG TCCTAAACTG GCAGGACTGT GAG
//
Вариант 2 (САG) нуклеотидной последовательности для R0007:
FT CDS 1..483
AATGAGGAGT GCACTGTCAC GGGTTTTCTG CGGGACAAGC TGCAGTACAG GAGCCGACTT CAGTACATGA AACACTACTT CCCCATCAAC TACAAGATCA GTGTGCCTTA CGAGGGGGTG TTCAGAATCG CCAACGTCAC CAGGCTGCAG AGGGCCCAGG TGAGCGAGCG GGAGCTGCGG TATCTGTGGG TCTTGGTGAG CCTCAGTGCC ACTGAGTCGG TGCAGGACGT GCTGCTCGAG GGCCACCCAT CCTGGAAGTA CCTGCAGGAG GTGCAGACGC TGCTGCTGAA TGTCCAGCAG GGCCTCACGG ATGTGGAGGT CAGCCCCAAG GTGGAATCCG TGTTGTCCCT CTTGAATGCC CCAGGGCCAA ACCTGAAGCT GGTGCGGCCC AAAGCCCTGC TGGACAACTG CTTCCGGGTC ATGGAGCTGC TGTACTGCTC CTGCTGTAAA CAAAGCTCCG TCCTAAACTG GCAGGACTGT GAG
//
У нас есть ещё время (почти месяц), чтобы найти в инете или уточнить у организаторов каким из кодонов кодируется этот злополучный глутамин во второй изоформе интерлейкина 34 и выбрать нужный вариант для R0007.
В самом крайнем случае есть возможность представить несколько моделей в виде нескольких вариантов файлов PDB, поэтому отправим оба варианта для R0007.
Kushelev: OK! Кстати, вероятно, можно будет по форме модели догадаться, какой из 2 вариантов правильный ![]()
Victoria: Загрузила на сайте CASP PDB файл для R0006.
Кушелев: Благодарю!
Сохраняйте, пожалуйста, копии странички после каждой загрузки нового файла. Если у нас будут "все ходы записаны", то в случае, если нас выкинут из списка участников, об этом будем знать не только мы с Вами![]()
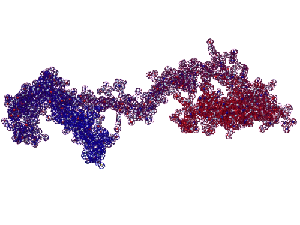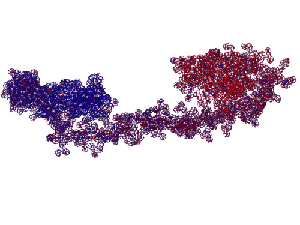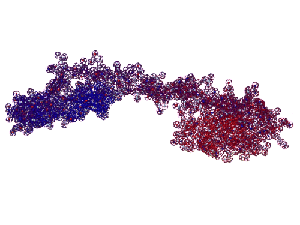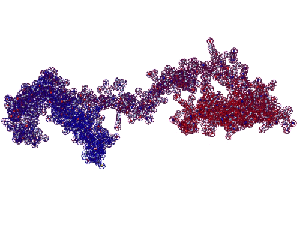
Первый вариант (слева) и второй вариант (справа) R0007.
Kushelev: Очевидно, что правильным является первый вариант.
Два кадра анимации высокого качества (4000х3000)
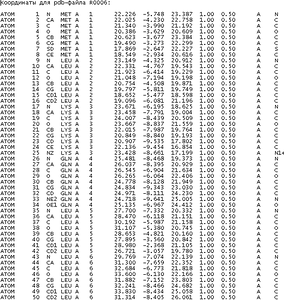
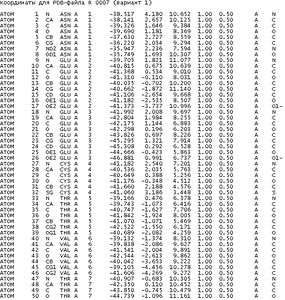
Координаты для pdb-файла R 0007
За развитием событий следите на форуме лаборатории Наномир...
http://nanoworld88.narod.ru/data/282.htm
Продолжаем участвовать в международном конкурсе CASP10
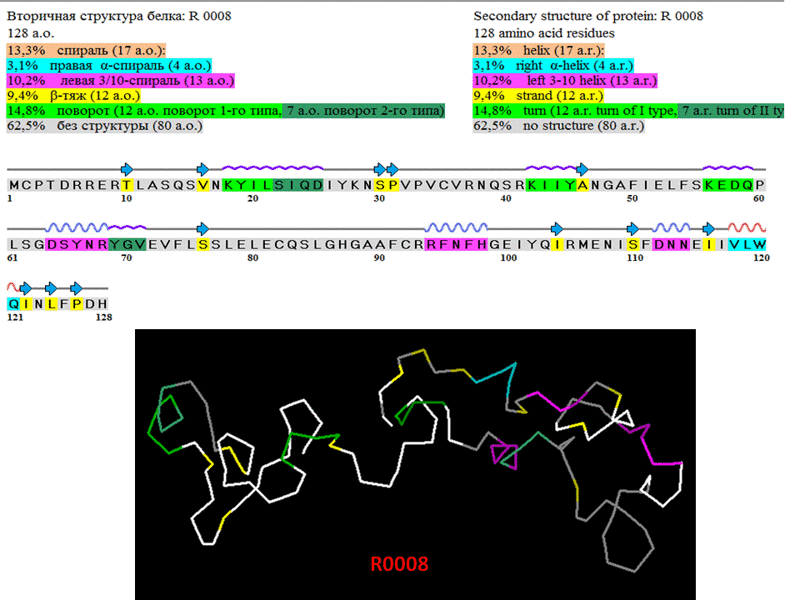
R0008 (PyMOL, mesh)
Victoria: Я ещё раз попросила у CASP нуклеотидные последовательности для их Target:
Ask to send, please, experimenters for me sequences of the nucleotide coding their peptides (R000n).
Our program works by analogy to a ribosome, because it predicts structure only such proteins that are synthesized by a matrix way at determining of its nucleotide sequences. For R0001, R0005, R0006 and R0007 we found this information in GenBank. Nucleotide sequences have not detected at all. Our method it isn’t applicable for the artificial peptides, synthesized by not a matrix way.
To me not to do without nucleotide sequences in yours CASP experiments anyway.
Help, please .
Ждём ответ .
R0008 (384 аминокислотных остатка) (PyMOL, mesh)
Почему нужно ввести новый стандарт представления белковых структур?
Каждые 2 года проводится конкурс определения белковых структур, CASP. В 2012-ом году стартовал
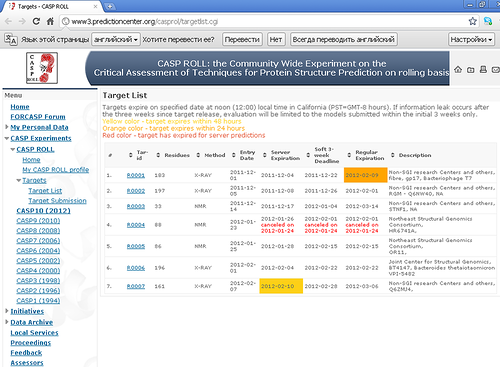
CASP10:http://predictioncenter.org/casp10/index.cgi
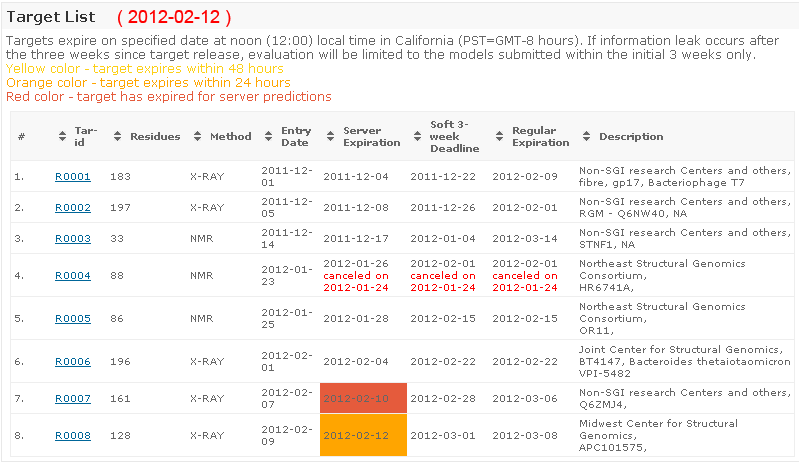 Так выглядит таблица, в которой появляются задания, аминокислотные последовательности белков, структуру которых предстоит определить участникам конкурса. Через некоторое время структура будет определена одним из экспериментальных методов, и можно будет узнать, кто же точнее смог "предсказать" эту структуру?
Так выглядит таблица, в которой появляются задания, аминокислотные последовательности белков, структуру которых предстоит определить участникам конкурса. Через некоторое время структура будет определена одним из экспериментальных методов, и можно будет узнать, кто же точнее смог "предсказать" эту структуру?
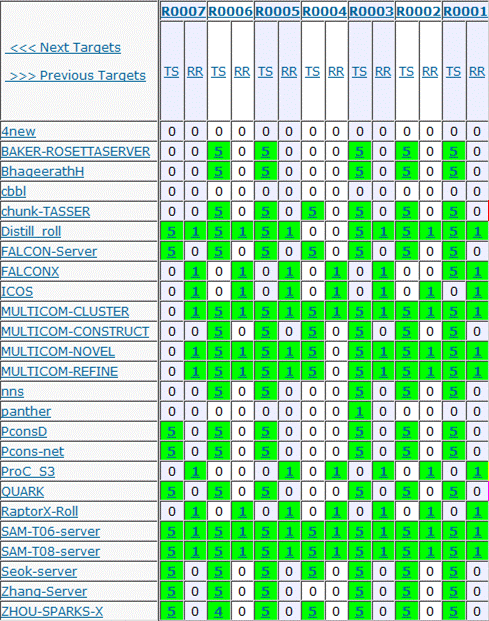
Результаты работы участников конкурса можно видеть в этой сводной таблице. К сожалению, не все участники отображаются здесь. Те, кто присылает результаты по почте или ставит на сервер CASP в режиме online, в настоящее время не отображаются. На форуме FORCASP несколько участников интересуется, можно ли увидеть общий список участников
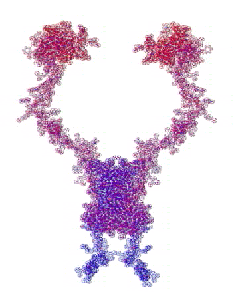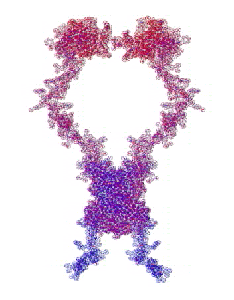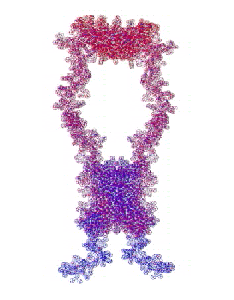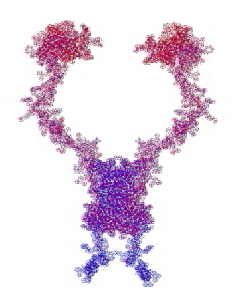
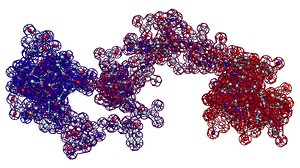
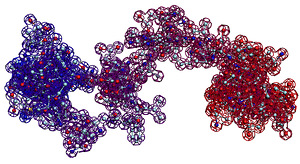
Самое точное "предсказание" - это эксперимент. В данном случае модельный эксперимент, который удалось автоматизировать в виде программы "Пикотех". В данном случае Вы видите результат моделирования динамики димера интерлейкина-34 / interleukin-34. На конкурсе CASP10 требуется определить лишь фрагмент структуры мономера.
Автоматизация модельных пикотехнологических экспериментов открывает золотой век пикотехнологии.

Достаточно открыть в программе файл с генетическим кодом белка, и через несколько минут или даже секунд Вы уже видите его структуру на экране Вашего компьютера.

В программе Пикотех можно регулировать уровень детализации модели белка. Например, эта гиперспираль коллагена построена из 14-гранников, символизирующих аминокислотные остатки.

Та же модель коллагеновой спирали, детализированная до уровня электронов.
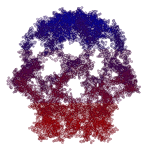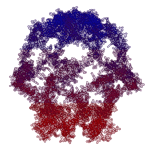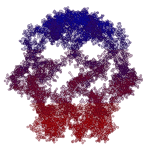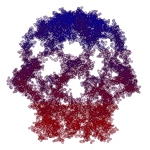

Программа Пикотех с легкостью определяет структуры шаперонов

гистонных комплексов



и начинает определять структуры ДНК и РНК...

При желании можно детализировать структуру до "путешествия к центру атома"

и даже увидеть тонкую структуру нуклонов! Но это уже не пико-, а фемтотехнология, которая биологам пока не нужна.
Лаборатория Наномир уже не первый раз принимает участие в международном конкурсе CASP. В прошлый раз организаторы конкурса поступили некорректно. Участники конкурса полтора месяца определяли и присылали результаты на конкурс. На сервере были накоплены результаты от 33 участников по 43 структуры
Накануне подведения итогов конкурса данные не сервере внезапно изменились. Число участников неожиданно уменьшилось с 33 до 28. Организаторы объяснили это "техническими причинами", но нетрудно догадаться, что они просто избавились от лишних участников, уничтожив их данные, которые накапливались полтора месяца на сервере международного конкурса.
В 2012-ом году организаторы поступили ещё хитрее. Они вообще скрывают список участников конкурса, чтобы можно было незаметно избавиться от любых участников. Но в век Internet сделать это становится практически невозможно, т.к. каждый участник может опубликовать в Internet свои результаты независимо от желания организаторов конкурса...
Но это лишь "надводная часть айсберга" ...
Ещё в 1998-ом году я предложил организаторам CASP ввести новые стандарты для представления вторичной и третичной структур белка. Дело в том, что используемый стандарт позволят отличать лишь спиральные участки белка от всего остального. Это слишком грубо. Ведь уже известно существование не только альфа-спирали, но и 310-спирали, пи-спирали, бета-спирали и множество специфических участков белка, которые не отражены в стандарте представления данных на конкурсе CASP.
В 1998-ом году организаторы конкурса написали, что рассмотрят моё предложение, но прошло 14 лет, "а воз и ныне там"
Тем не менее лаборатория Наномир снова участвует в конкурсе CASP и публикует собственные данные по мере их поступления. Поэтому все желающие могут следить за развитием событий.
Кстати, а Вы не желаете принять участие в международном конкурсе CASP10 ?
Материал с форума лаборатории Наномир:
Victoria: Silvia Crivelli (LBNL, UC Davis, член CASP с 1998) организует совместную группу, чтобы участвовать в CASP10 и возможно в CASP ROLL, если успеет. Этот совместный эксперимент позволит различным группам или индивидуумам работать над различными компонентами предсказания структуры белка (такими как выравнивание, моделирование петли, выигрыш, и т.д.) таким образом позволяя усилить экспертизу в большем масштабе. Начальная цель состоит в том, чтобы сосредоточиться на задачах. Определенные группы могут участвовать в совместном решении всех задач с самого начала, в то время как другие могут сделать только несколько из них.
Если Вы хотели бы узнать больше об этом намерении, пожалуйста, посетите wefold.wordpress.com или свяжитесь с Сильвией непосредственно в SNCrivelli@lbl.gov.
Дама стратегически мыслит, сколачивая большую группу с разделением труда для участия в конкурсе CASP ROLL .
Kushelev: Может быть напишем ей, расскажем о пикотехнологии? Нам любые зацепки могут помочь выйти на рынок белковых структур. Если предложит объединить усилия, то почему бы и нет? Главное условие, чтобы наше участие было задокументировано. Всё остальное - мелочи. ... если у нас есть наиболее точные координаты атомов, а у Сильвии есть другие возможности, которых нет у нас, то объединение усилий - это шанс на победу по многим параметрам. Ведь CASP - это всего лишь тактика, а выход на клиентов - часть стратегии...
http://nanoworld88.narod.ru/data/282.htm
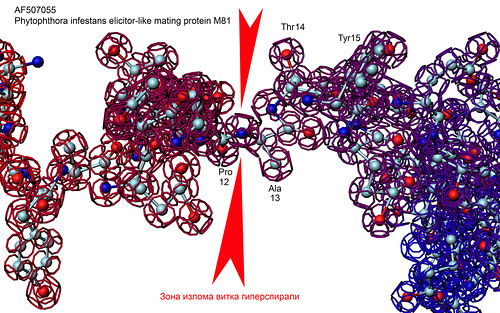
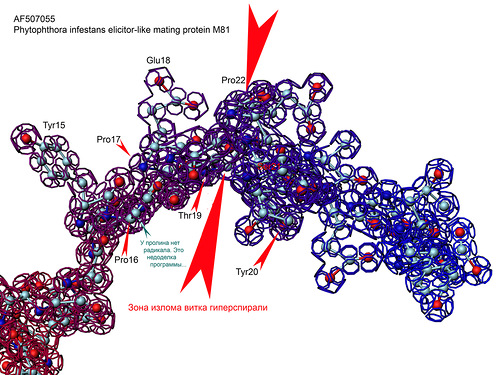
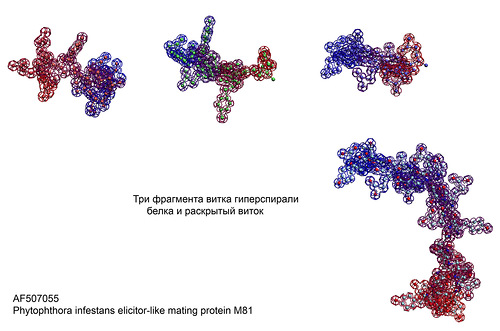
Гиперспираль белка M81
Phytophthora infestans (potato late blight agent)
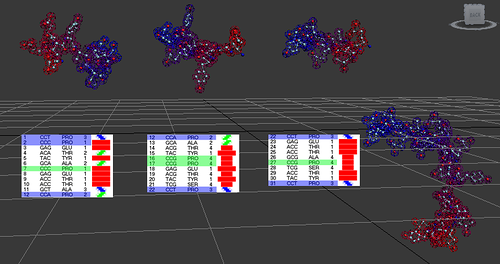
Схема вторичной структуры белка M81 показывает повторяющиеся участки:

"Распущенная" гиперспираль
3,1,1,2,1,2,1,1,1,1,3,2,2,4,1,4,4,1,4,1,4,3,1,1,1,4,4,4,1,1,
Повторяющийся участок композиционного кода.
Разомкнутый виток гиперспирали.
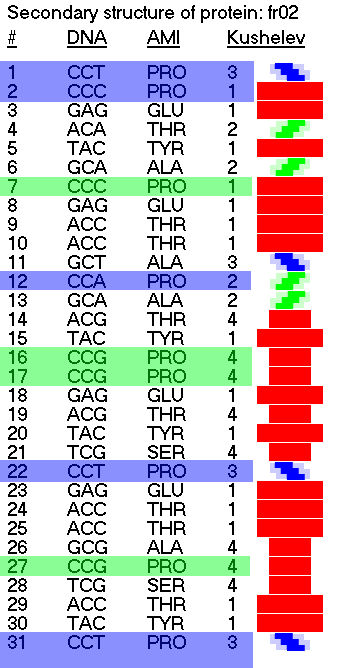
Виток разделён на три гнутых участка альфа-спирали, разделённых гибкими пролинами. В составе альфа- и 310-спиралей пролин менее гибкий, т.к. стабилизирован водородными связями. таким образом мы приходим к выводу, что гиперспираль этого белка близка в сечении к криволинейному треугольнику.
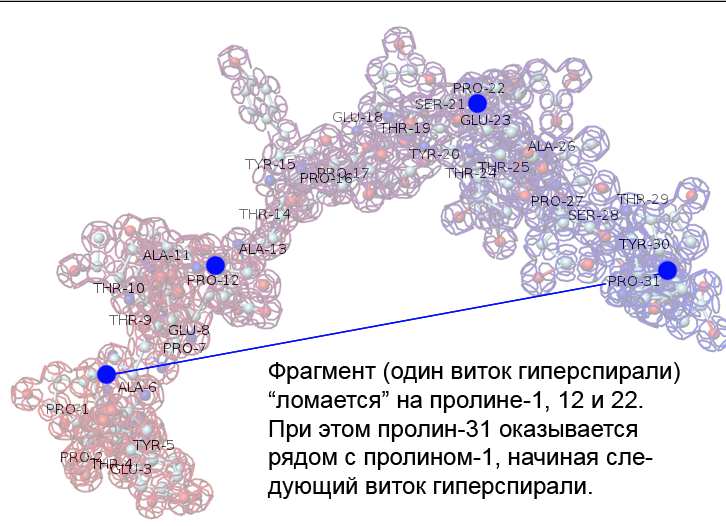
Атом, принадлежащий пролину (Pro22), помечен зелёным цветом. Именно в этом месте нужно согнуть модель белка
Попробуем собрать виток гиперспирали белка в виртуальном пространстве 3DS Max...
Согнём виток гиперспирали белка на пролине 12 и на пролине 22. В результате получается виток вовсе не треугольного, а вполне круглого сечения!
Так выглядит один виток этой необычной гиперспирали белка.
Модель гиперспирали. Конечно, эта модель имеет разную точность в разных участках, но даёт представление о гиперспирали белка. Более точную модель можно будет построить позже, когда появятся технические возможности...
Эндолизин, да не тот...

Кушелев: Нашёл нуклеотидную последовательность эндолизина, но другого:
FT CDS <8..305
ACTTTGAAGGAAAATATATTACGCAGCAAACTCGCTGCTACTATATCGTACATATCTAGA
CCAGCTCCAACGTACCATTGAAGTTCTTGATTCGCTCATCGAGGATAATGGAGCTCAGAA
CCCAATCACCATCCCGAGACAACTCGCCCTCGAGCCTTTGACCTCCATCAACAAGACGCA
CATTTCGCGCCGAGTTTCCGAATCCACCACCACCTTGGCTACCACCGCCAGGGACCTGGA
GGGTCTGTCCAGGGTAAATCAGGTCAGGGTTGCTAATGTTGTTCTGCCGAGCAATCTCTT
CGAATGAAGTGTTGAATCGGGCAGCAATGCTTCGGAGAGTGTCTCCGTGCTGGACTGTGA
CCTGGGAAGAACCACCGTGATGGCCGCCAGCAGACCAACGGAAGCTTCCTCTATCATTCT
CCAGTAGCTTGTTGAGGGAGATGGATGTGTGCTGGTAGTCG
//
Эндолизин (PuMOL, mesh)
Уважаемая Виктория! Давайте пошлём этот pdb-файл на конкурс с оговоркой, что это - структура другого эндолизима, нуклеотидную кодирующую последовательность которого нам удалось найти в генбанке. Если организаторы найдут нуклеотидную последовательность конкурсного эндолизина, то они смогут получить и его структуру. А если не смогут, значит "не судьба"...
Администрация форума Molbiol.ru считает международный конкурс CASP темой, недостойной для обсуждения...