http://nanoworld88.narod.ru/data/227.htm
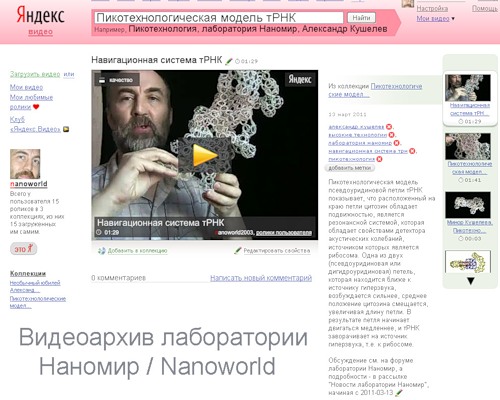
Пикотехнологическая модель псевдоуридиновой петли тРНК показывает, что расположенный на краю петли цитозин обладает подвижностью, является резонансной системой, которая обладает свойствами детектора акустических колебаний, источником которых является рибосома. Одна из двух (псевдоуридиновая или дигидроуридиновая) петель, которая находится ближе к источнику гиперзвука, возбуждается сильнее, среднее положение цитозина смещается, увеличивая длину петли. В результате, петля начинает двигаться медленнее, и тРНК заворачивает на источник гиперзвука, т.е. к рибосоме.
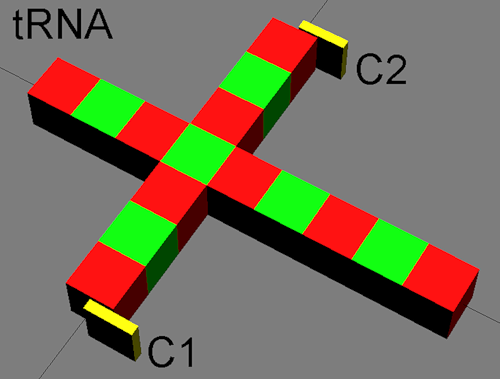
Схематическое изображение тРНК. На краях псевдоурациловой и дигидроурациловой петель изображены азотистые основания (цитозины) в невозбуждённом состоянии. В отсутствии акустического сигнала рибосомы тРНК движется по прямой, вращаясь вокруг большой оси.

Если же появляется акустический сигнал рибосомы, то цитозины начинают колебаться, причём амплитуда колебаний возрастает, если цитозин приближается к рибосоме. При этом ближний к рибосоме цитозин сильнее тормозит, в результате чего тРНК заворачивает к источнику акустического синала, т.е. к рибосоме.

Приблизительно так работает навигационная система тРНК
Упрощённая модель тРНК, визуализирующая механизм навигации тРНК.
Крайние цитозины псевдоурациловой и дигидроурациловой петель тРНК подвижны и являются элементами резонансного рулевого управления тРНК. Амплитуда асимметричных колебаний цитозинов тем выше, чем ближе они к источнику гиперзвука, которым является активный центр рибосомы. Таким образом, тРНК плывут на зов рибосомы.
Скрипт для 3DS Max:
b1 = box length:1 width:1 height:1 pos:[-3,0, -0.5] wirecolor: [255,0,0]
b2 = box length:1 width:1 height:1 pos:[-2,0, -0.5] wirecolor: [0,255,0]
b3 = box length:1 width:1 height:1 pos:[-1,0, -0.5] wirecolor: [255,0,0]
b4 = box length:1 width:1 height:1 pos:[0,0, -0.5] wirecolor: [0,255,0]
b5 = box length:1 width:1 height:1 pos:[1,0, -0.5] wirecolor: [255,0,0]
b6 = box length:1 width:1 height:1 pos:[2,0, -0.5] wirecolor: [0,255,0]
b7 = box length:1 width:1 height:1 pos:[3,0, -0.5] wirecolor: [255,0,0]
b8 = box length:1 width:1 height:1 pos:[0,1, -0.5] wirecolor: [255,0,0]
b9 = box length:1 width:1 height:1 pos:[0,2, -0.5] wirecolor: [0,255,0]
b10 = box length:1 width:1 height:1 pos:[0,3, -0.5] wirecolor: [255,0,0]
b11 = box length:1 width:1 height:1 pos:[0,-1, -0.5] wirecolor: [255,0,0]
b12 = box length:1 width:1 height:1 pos:[0,-2, -0.5] wirecolor: [0,255,0]
b13 = box length:1 width:1 height:1 pos:[0,-3, -0.5] wirecolor: [255,0,0]
b14 = box length:1 width:1 height:1 pos:[0,-4, -0.5] wirecolor: [0,255,0]
b15 = box length:1 width:1 height:1 pos:[0,-5, -0.5] wirecolor: [255,0,0]
g1 = group #(b1,b2,b3,b4,b5,b6,b7,b8,b9,b10,b11,b12,b13,b14,b15)
c1 = box length:1 width:0.2 height:1 pos:[-3.65,-0.55, -0.5] wirecolor: [255,255,0]
c2 = box length:1 width:0.2 height:1 pos:[3.65,-0.55, -0.5] wirecolor: [255,255,0]
c1.pivot = [-3.6,-0,-0,5]
rotate c1 -90 [0,0,1]
c2.pivot = [3.6,0,-0,5]
rotate c1 90 [0,0,1]
На современном компьютере можно будет создать компьютерную графику с менее упрощённой моделью тРНК...
Материал с форума лаборатории Наномир:
Пространственная структура биомолекул
Victoria: Крайние цитозины псевдоуридиловой и дигидроуридиловой петель формируют канонические комплементарные пары с гуанинами даже во вторичной структуре клеверного листа.
Кушелев: Нет, на схеме показано, что они не образуют комплементарных пар:
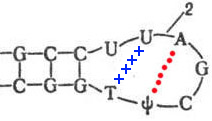
Крайние гуанин и цитозин соединены только через фосфатные группы.
Victoria: Такие пары G-C расположены в начале каждой из трёх петель тРНК (а также входят в состав всех стеблей тРНК). Поэтому "крайние" цитозины быть "ластами" тРНК при её движении на зов рибосомы НЕ МОГУТ.
Кушелев: В начале петель действительно расположены комплементарные пары G-C, но на краю петель расположены одиночные нуклеотиды G и C, которые не образуют между собой водородных связей.
Victoria: Понятно, Вы имели в виду цитозин на другом краю псевдоуридиловой петли.Но в дигидроуридиловой петле цитозина то НЕТ. Что мы будем с этим делать. Кто будет ластами махать вместо цитозина?
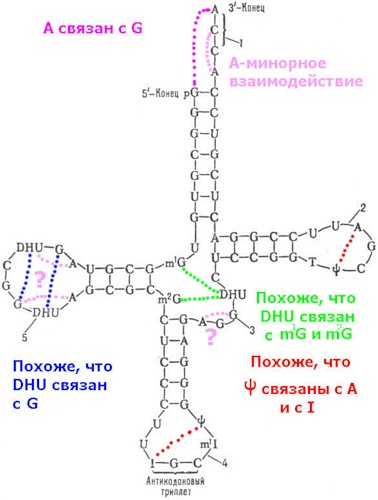
Кушелев: А если внимательнее посмотреть? ![]()
Виктория Соколик: На этой схеме ошибка. Я специально сверялась с данными тРНК-банка: цитозина в дигидроуридиловой петле нет. Обе петли (дигидроуридиловая и псевдоурациловая) состоят из 7 нуклеотидов каждая. Посмотрите сообщение №39 этой темы.
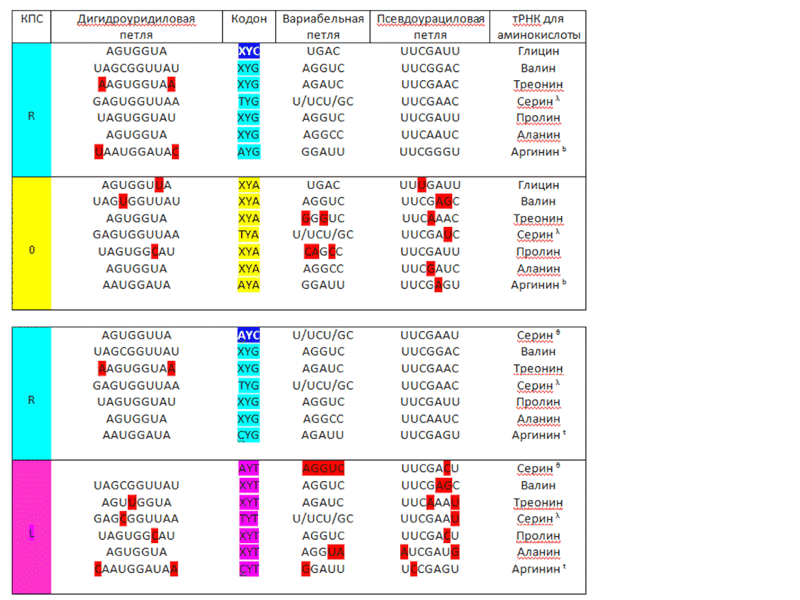
Кушелев: Если не CG, то UG. Резонансные частоты могут совпадать. Кстати, в таблице есть очень полезная информация. Особенно по вариабельным петлям... Кстати, нет ли у Вас таблицы, где вместо кодонов типа XYA находятся 64 кодона в явном виде?
Виктория Соколик: Если Вы спрашиваете о таблице с первичной структурой петель изоакцепторных тРНК, то я её составляла сама по данным из тРНК банка. В последнем приведена информация не для всех разновидностей изоакцепторных тРНК для каждой аминокислоты. В моей таблице представлена информация по тем аминокислотам, для которых информация наиболее полная. Что же до дуплета первых двух нуклеотидов кодона, то что Вам мешает подставить туда соответствующие буквы (G, C, U или А) вместо X и Y.
***
Кушелев: Модельный эксперимент поможет выяснить, какова степень балансировки тРНК. Если разбалансировка на уровне одного нуклеотида, то моя гипотеза подтвердится. Но это не будет означать, что Ваша гипотеза будет опровергнута. Ведь органические молекулы, в частности, тРНК многофункциональны...
***
Кушелев: L-образная форма тРНК - гипотеза, которая фактически уже опровергнута моим модельным экспериментом. Хотя любая тРНК может "склеить ласты", и стать L-образной.
Виктория Соколик: L-образная форма тРНК -- это экспериментальный факт, который Вы "опровергли" модельным экспериментом только для себя.
Кушелев: А с чего Вы взяли, что это вообще факт, а не гипотеза? И даже если в каком-то эксперименте закристаллизовали "стаю" тРНК, и эти тРНК в процессе кристаллизации "склеили ласты", то отсюда не следует, что до кристаллизации тРНК были L-образными. Согласны?
***
Виктория Соколик: ... рассчитайте резонансную частоту для тРНК по формуле Томсона, а потом в эксперименте с флюоресцентно-меченными тРНК продемонстрируйте их "столпотворение" на зов синтезатора. Это будет отличным Вашим собственным неопровержимым результатом к пикотехнологическому конгрессу.
Кушелев: Частоту колебаний цитозинового "руля" можно оценить по формуле Томсона для пружинного и математического маятников. Длина маятника порядка нанометра. Масса маятника на порядок превышает массу атома углерода, т.е. составляет ~120 углеродных единиц. С коэффициентом упругости, конечно, сложнее, но можно схитрить. Можно оценить верхнее значение линейной скорости колебаний азотистого основания. Она должна быть того же порядка, что средняя скорость теплового движения молекул воды, т.е. ~10^3 м/с или меньше. Если скорость 10^3 м/с, то нанометр с этой скоростью можно пройти за 10^-9 / 10^3 = 10^-12 сек, т.е. за пикосекунду. Таким образом, верхняя оценка частоты собственных колебаний цитозинового "руля" составляет 1THz. Таким образом, можно начать с терагерца и постепенно понижать частоту синтезатора. Как только частота совпадёт с собственной частотой цитозиновых "рулей", тРНК поплывут к источнику гиперзвука, и это можно будет увидеть в оптический микроскоп.
Разгадана технология трансляции!
Два изомера метилинозина. Слева изомер Александра Кушелева, справа - Виктории Соколик.
Начинаем сборку антикодоновой петли тРНК
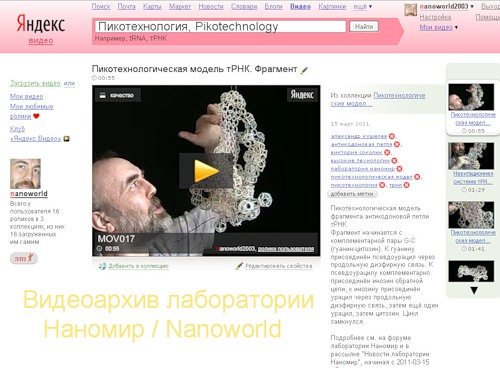
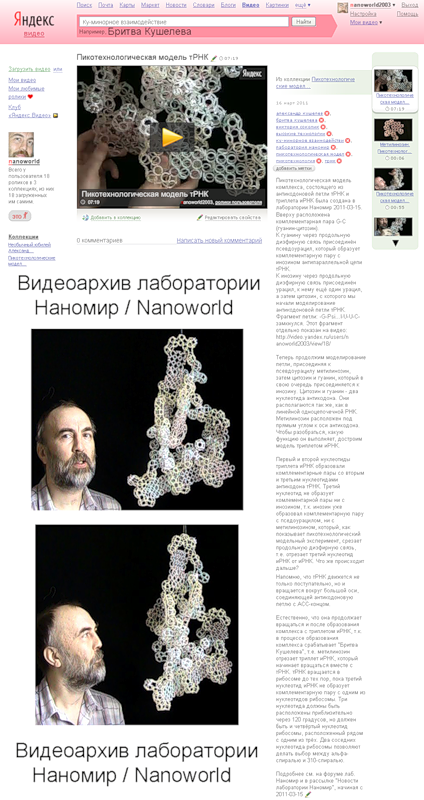
Пикотехнологическая модель комплекса, состоящего из антикодоновой петли тРНК и триплета иРНК была создана в лаборатории Наномир 2011-03-15. Вверху расположена комплементарная пара G-C (гуанин-цитозин).
К гуанину через продольную диэфирную связь присоединён псевдоурацил, который образует комплементарную пару с инозином антипараллельной цепи тРНК. К инозину через продольную диэфирную связь присоединён урацил, к нему ещё один урацил, а затем цитозин, с которого мы начали моделирование антикодоновой петли тРНК. Фрагмент петли: -G-Psi...I-U-U-C- замкнулся. Этот фрагмент отдельно показан на видео: http://video.yandex.ru/users/nanoworld2003/view/18/
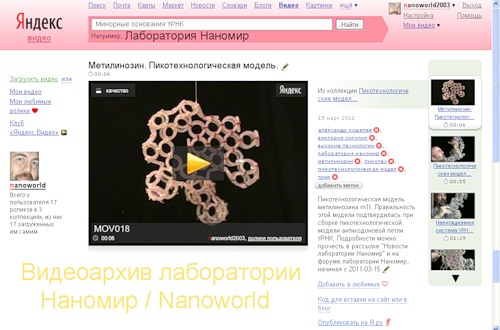
Пикотехнологическая модель метилинозина m1I. Правильность этой модели подтвердилась при сборке пикотехнологической модели антикодоновой петли тРНК.
Виктория Соколик: Вы в моей модели метилинозина выпятили метильную группу и оголили торцы блока. Не красивый у Вас "правильный" метилинозин вышел и скорее всего малоустойчив, поскольку открытая и неравновесная структура. Вы уж простите меня на добром слове, Ваша новая модель метилинозина еще хуже предыдущей.
Теперь продолжим моделирование петли, присоединяя к псевдоурацилу метилинозин, затем цитозин и гуанин, который в свою очередь присоединяется к инозину. Цитозин и гуанин - два нуклеотида антикодона. Они располагаются так же, как в линейной одноцепочечной РНК. Метилинозин расположен под прямым углом к оси антикодона. Чтобы разобраться, какую функцию он выполняет, достроим модель триплетом иРНК.
Первый и второй нуклеотиды триплета иРНК образовали комплементарные пары со вторым и третьим нуклеотидами антикодона тРНК. Третий нуклеотид не образует комлементарной пары ни с инозином, т.к. инозин уже образовал комплементарную пару с пседоурацилом, ни с метилинозином, который, как показывает пикотехнологический модельный эксперимент, срезает продольную диэфирную связь, т.е. отрезает третий нуклеотид иРНК от иРНК. Что же происходит дальше?

Напомню, что тРНК движется не только поступательно, но и вращается вокруг большой оси, соединяющей антикодоновую петлю с АСС-концом.
Естественно, что она продолжает вращаться и после образования комплекса с триплетом иРНК, т.к. в процессе образования комплекса срабатывает "Бритва Кушелева", т.е. метилинозин отрезает триплет иРНК, который начинает вращаться вместе с тРНК. тРНК вращается в рибосоме до тех пор, пока третий нуклеотид иРНК не образует комплементарную пару с одним из нуклеотидов рибосомы. Три нуклеотида должны быть расположены приблизительно через 120 градусов, но должен быть и четвёртый нуклеотид рибосомы, расположенный рядом с одним из трёх. Два соседних нуклеотида рибосомы позволяют делать выбор между альфа-спиралью и 310-спиралью.
Виктория Соколик: Всё это занимательно, но слишком сложно. Природа выбирает изящные и лаконичные решения. Я не разделяю Ваших представлений и не буду даже аргументировано опровергать, потому что ВСЁ не так.